1. Ermitteln Sie die Absorption der RNA-Lösung
Die Absorption bei 280, 320, 230 und 260 nm stellt die Werte der Nukleinsäure, des Hintergrunds (Lösungstrübung), der Salzkonzentration und der organischen Substanz wie Protein dar.Betrachten Sie im Allgemeinen nur OD260/OD280 (Verhältnis, R).Bei einem Wert von 1,8 bis 2,0 gehen wir davon aus, dass die Kontamination von Proteinen oder anderen organischen Stoffen in der RNA toleriert werden kann. Es ist jedoch zu beachten, dass der R-Wert bei Verwendung von Tris als Puffer zum Nachweis der Absorption größer als 2 sein kann (im Allgemeinen sollte er <2,2 sein).Wenn R <1,8 ist, ist die Verschmutzung durch Proteine oder andere organische Stoffe in der Lösung offensichtlicher und das Schicksal der RNA kann je nach Bedarf bestimmt werden.Wenn R > 2,2, bedeutet dies, dass die RNA zu einer einzelnen Nukleinsäure hydrolysiert wurde.
2.Elektrophoretisches Muster der RNA
Im Allgemeinen wird für die RNA-Elektrophorese ein denaturierendes Gel verwendet. Wenn es jedoch nur zum Nachweis der RNA-Qualität dient, ist ein denaturierendes Gel nicht erforderlich und es kann ein gewöhnliches Agarosegel verwendet werden.Der Zweck der Elektrophorese besteht darin, die Integrität der 28S- und 18S-Banden und deren Verhältnis oder die Integrität des mRNA-Abstrichs festzustellen.Wenn die 28S- und 18S-Banden hell, klar und scharf sind (bezogen auf die klaren Kanten der Banden) und die Helligkeit von 28S mehr als doppelt so hoch ist wie die der 18S-Bande, betrachten wir die Qualität der RNA im Allgemeinen als gut.
Die oben genannten Methoden sind die beiden Methoden, die wir üblicherweise verwenden, aber keine dieser beiden Methoden kann uns eindeutig sagen, ob in der RNA-Lösung restliche RNase vorhanden ist.Wenn sich in der Lösung eine sehr geringe Menge an RNase befindet, ist es für uns schwierig, diese mit der oben genannten Methode nachzuweisen, aber die meisten nachfolgenden enzymatischen Reaktionen werden bei über 37 Grad und über einen langen Zeitraum durchgeführt.Wenn also nur eine sehr kleine Menge RNase in der RNA-Lösung vorhanden ist, gibt es eine sehr geeignete Umgebung und Zeit, um ihre Rolle in den nachfolgenden Experimenten zu spielen, und natürlich wird das Experiment zu diesem Zeitpunkt kalt sein.Im Folgenden stellen wir eine Methode vor, mit der bestätigt werden kann, ob in der RNA-Lösung restliche RNase vorhanden ist.
3. Hitzekonservierungstest
Ziehen Sie entsprechend der Probenkonzentration zwei 1000-ng-RNAs aus der RNA-Lösung, geben Sie sie in ein 0,5-ml-Zentrifugenröhrchen, ergänzen Sie sie mit Tris-Puffer mit pH 7,0 auf ein Gesamtvolumen von 10 ul und verschließen Sie dann den Deckel des Röhrchens.Legen Sie eines davon in ein Wasserbad mit konstanter Temperatur von 70 °C und halten Sie es 1 Stunde lang warm.Der andere Teil wurde 1 Stunde lang in einem Kühlschrank bei -20 °C gelagert.Wenn die Zeit abgelaufen ist, entnehmen Sie die beiden Proben zur Elektrophorese.Nachdem die Elektrophorese abgeschlossen ist, vergleichen Sie die elektrophoretischen Banden der beiden.Wenn die Banden der beiden konsistent sind oder keinen signifikanten Unterschied aufweisen (natürlich erfüllen ihre Banden auch die Bedingungen in Methode 2), bedeutet dies, dass in der RNA-Lösung keine restliche RNase-Kontamination vorhanden ist und die Qualität der RNA sehr gut ist.Wenn die bei 70 °C inkubierte Probe hingegen einen offensichtlichen Abbau zeigt, weist dies darauf hin, dass in der RNA-Lösung eine RNase-Kontamination vorliegt.
2 Experimentelle Methoden und Techniken zur RNA-Extraktion
Bei der Extraktion von RNA treten häufig folgende Probleme auf: (1) Die RNA-Ausbeute ist gering;(2) RNA weist eine schwere Salzverschmutzung auf;(3) RNA weist eine schwerwiegende Verschmutzung durch organische Lösungsmittel auf;(4) Probenverschlechterung und andere Probleme
1. Häufig verwendete Gesamt-RNA-Extraktionsreagenzien
Die Guanidinisothiocyanat-Methode und die Trizol-Methode sind die am häufigsten verwendeten Methoden zur Extraktion von Gesamt-RNA aus tierischen Geweben und tierischen Zellen.Es eignet sich besonders für kleine Proben und Gewebe, die besonders schwer zu extrahieren sind, wie z. B. die Extraktion von Gesamt-RNA aus Kaninchenhaut und tierischem Bindegewebe;Darüber hinaus kann Trizol als Allzweck-Lysereagenz auch zur Extraktion von Pflanzengewebe, Bakterien, Pilzen und anderen Geweben verwendet werden.Bei pflanzlichen Geweben, die Polysaccharide und Polyphenole enthalten, wie z. B. Camellia oleifera, Teeblätter, Raps usw., kann die CTAB-Methode auch zur Extraktion von Gesamt-RNA verwendet werden.
Als herkömmliche Methode erfreut sich die Doppelsäulenmethode auch großer Beliebtheit, da sie bei normaler Temperatur betrieben werden kann, keine RNase-Zugabe erforderlich ist und sicher ist – kein Chloroform, Phenole und andere organische Reagenzien für die Extraktion.(Empfohlene Produkte )

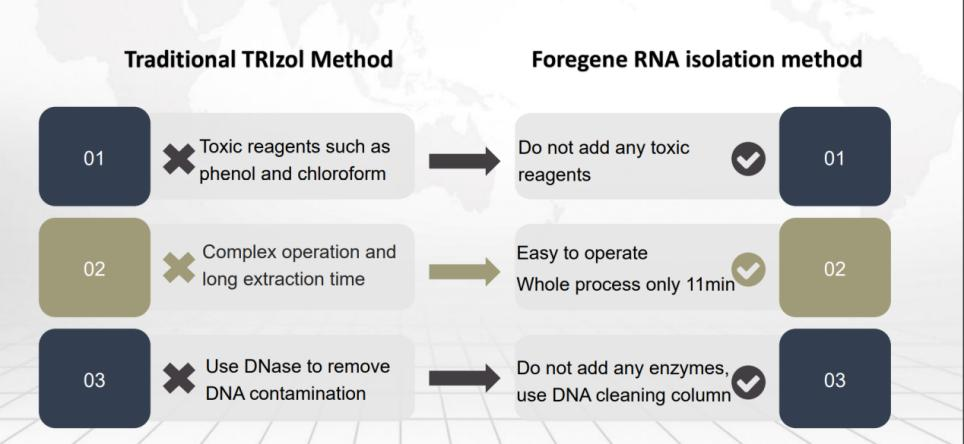
2. Extraktion der Gesamt-RNA aus tierischen Geweben
(1) Versuchen Sie, frisches Gewebe zu wählen, wenn es nicht frisch ist (vorzugsweise innerhalb von drei Monaten – 80 °C im Kühlschrank oder in flüssigem Stickstoff eingefroren). Wenn Sie Gewebe schneiden, schneiden Sie es nicht direkt bei Raumtemperatur, legen Sie es unbedingt auf die Eisbox und vermeiden Sie wiederholtes Einfrieren und Auftauen.
(2) Schneiden Sie mit einer sauberen Schere und einer Pinzette ein kleines Stück Gewebe ab. Versuchen Sie beim Schneiden der Probe, den mittleren Teil des Gewebes zu durchtrennen, oder schneiden Sie zuerst das große Gewebestück in der Mitte ab und schneiden Sie dann die Probe an der frischen Schnittposition.Das entnommene Gewebe sollte vollständig zerkleinert sein, das zerkleinerte Gewebe in ein EP-Röhrchen ohne RNase geben, das Lysat hinzufügen, das zerkleinerte Gewebe sollte vollständig dem Lysat ausgesetzt sein und für die Homogenisierung vorbereitet werden.
(3) Wählen Sie für normales Gewebe mungbohnengroßes Gewebe (30–60 mg) zur Homogenisierung aus.Wenn das Gewebe eine große Menge Protein, Fett oder dichtes Fasergewebe wie Leber enthält, erhöhen oder verringern Sie die Menge des geschnittenen Gewebes entsprechend (optional). Wählen Sie 10–20 mg).
(4) Wenn Fischmuskeln, Garnelenfleisch, Quallen und andere Gewebe mit hohem Wassergehalt extrahiert werden, sollte das Probenvolumen entsprechend erhöht werden (empfohlen 100–200 mg).
(5) Wenn die Bedingungen es zulassen, kann das tierische Gewebe nach der Homogenisierung mit einem Hochdurchlass-Gewebehomogenisator direkt entnommen werden, sofern keine solche Ausrüstung vorhanden ist.
(6) Die nach der letzten Extraktion erhaltene RNA muss sofort auf die Eisbox gelegt werden, um den Abbau der RNA zu reduzieren.
3. RNA-Extraktion aus tierischen Zellen
(1) Suspensionszellen: Direkt zentrifugieren und das Medium verwerfen, 1-2 Mal mit sterilem PBS waschen, dann mit einer geeigneten Menge PBS suspendieren und dann Lysat zur Lyse hinzufügen.Geben Sie das Lysat nicht direkt zu den präzipitierten Zellen, nachdem Sie die Flüssigkeit vollständig verworfen haben.Dies führt dazu, dass das Histonpaket, das nach der Lyse der Zellen auf der Außenschicht freigesetzt wird, an der Außenseite der präzipitierten Zellen haftet, wodurch der Kontakt der Zellen im Pellet mit dem Lysat begrenzt wird., was zu einer unvollständigen Zelllyse und einer verringerten RNA-Ausbeute führt.
(2) Halbadhärente oder nicht fest haftende Zellen: Nachdem Sie das Medium verworfen haben, waschen Sie es ein- bis zweimal mit PBS, nehmen Sie dann direkt eine entsprechende Menge PBS auf und blasen Sie die Kulturschale mit einer Pipette oder Pistole aus, um die Zellen abzublasen, und übertragen Sie sie auf RNA-freie Zellen.Geben Sie das Lysat zur Extraktion in das EP-Röhrchen des Enzyms.
(3) Anhaftende Zellen: müssen zuerst mit Trypsin verdaut, dann in RNase-freien EP-Röhrchen gesammelt, zentrifugiert werden, um den Überstand zu entfernen, ein- bis zweimal mit PBS gewaschen werden, um überschüssiges Trypsin zu entfernen, und mit einer geeigneten Menge PBS resuspendiert werden. Fahren Sie dann mit dem Extraktionsschritt fort.
4. Pflanzen-RNA-Extraktion
Pflanzengewebe sind reich an Phenolverbindungen oder reich an Polysacchariden, enthalten einige nicht identifizierte Sekundärmetaboliten oder weisen eine hohe RNase-Aktivität auf.Diese Substanzen verbinden sich nach der Zelllyse eng mit der RNA und bilden unlösliche Komplexe oder kolloidale Niederschläge, die schwer zu entfernen sind.Wenn wir Pflanzengewebe extrahieren, müssen wir daher ein Set für Pflanzen auswählen.Das im Kit enthaltene Lysat kann die Probleme der leichten Oxidation von Polyphenolen und der Trennung von Polysaccharidverbindungen und Nukleinsäuren wirksam lösen.
(Für die Polysaccharid-Polyphenol-Pflanzen-RNA-Extraktion werden folgende Produkte empfohlen:
(1) Schale, Fruchtfleisch, Samen, Blätter usw. der Pflanze sollten in einem Mörser vollständig zermahlen werden.Während des Mahlvorgangs sollte rechtzeitig flüssiger Stickstoff nachgefüllt werden, um ein Schmelzen der Probe zu vermeiden.Die gemahlene Probe sollte schnell zum Lysat gegeben und geschüttelt werden, um einen RNA-Abbau zu vermeiden.
(2) Bei faserreichen Proben wie Reis- und Weizenblättern sollte die Extraktionsmenge entsprechend reduziert werden, da sonst die Gewebezerkleinerung und -lyse nicht vollständig ist, was zu einer geringen Ausbeute an extrahierter RNA führt.
(3) Bei Pflanzengeweben mit hohem Wassergehalt, wie Granatapfelfrüchten, Wassermelonenfrüchten, Pfirsichfrüchten usw., sollte die Probengröße entsprechend erhöht werden (100–200 mg sind optional).
(4) Bei Pflanzengeweben wie Pflanzenblättern, Rhizomen, harten Früchten und anderen Materialien wird im Allgemeinen empfohlen, die Zutaten mit flüssigem Stickstoff gründlich in einem Mörser zu mörsern und dann mit dem Extraktionsschritt fortzufahren.Herkömmliche Gewebehomogenisatoren sind bei der Homogenisierung von Pflanzengewebe möglicherweise nicht wirksam und werden im Allgemeinen nicht empfohlen.
5. Vorsichtsmaßnahmen für die RNA-Extraktion
(1) Gewebeproben sollten möglichst frisch sein, um wiederholtes Einfrieren und Auftauen zu vermeiden.
(2) Das Gewebe sollte bei der Extraktion vollständig zerkleinert werden und die Gewebemenge sollte nicht zu gering, geschweige denn zu groß sein.
(3) Nach Zugabe des Lysats sollte ausreichend Inkubationszeit eingehalten werden, um die Probe vollständig zu lysieren.
(4) Bei Verwendung der Trizol-Methode zur Extraktion gilt beim Absorbieren des Überstands nach der Schichtung das Prinzip „Lieber weniger inhalieren als mehr inhalieren“ und darf nicht bis zur mittleren Schicht extrahiert werden, da es sonst zu einer schwerwiegenden Kontamination der genomischen DNA kommt.
(5) Beim Waschen sollte die Waschflüssigkeit vollständig in die Rohrwand eindringen, um eine gründliche Reinigung zu gewährleisten.
(6) Bei der Säulenextraktionsmethode sollte die Adsorptionssäule zusätzlich zum Abnehmen der Säule nach dem Waschen auch in eine ultrareine Bank gestellt und 5–10 Minuten lang geblasen werden, um das organische Lösungsmittel vollständig zur Trockne zu verdampfen.
(7) Bei der letzten Elution der Säulenmethode sollte nach Zugabe von DEPC-Wasser 3–5 Minuten lang inkubiert werden, oder das DEPC-Wasser sollte vorher auf 60 °C erhitzt werden, um die Elutionsausbeute zu erhöhen.Bei der traditionellen Trizol-Spaltung und Isopropanol-Fällungsmethode wird die endgültige RNA in DEPC-Wasser gelöst, daher sollte eine angemessene Zeit zum Auflösen gegeben werden und der Boden des Zentrifugenröhrchens kontinuierlich mit einer Pipettenspitze geblasen werden.
3 TDrei Ursachen und Lösungen für niedrige RNA-Konzentration/schlechte Qualität
1. Der Ertrag ist zu gering
Die extrahierte Probe ist zu niedrig, die Gesamtmenge ist unzureichend oder die extrahierte Probe ist zu groß und die Lyse ist nicht vollständig;Für die Extraktion sollten Gewebe oder Zellen geeigneter Qualität verwendet werden, die Vorbehandlung der Probe muss gut durchgeführt werden und die Lyse sollte ausreichend sein.
2. Genomreste
Wenn bei der Extraktion mit der Trizol-Methode der Überstand nach dem Schichten in die mittlere Schicht gesaugt wird, kommt es zu einer ernsthaften Genomkontamination;Beim Schichten ist besondere Vorsicht geboten, um ein Einsaugen in die Mittelschicht zu vermeiden.Wenn die Säulenmethode zur Extraktion verwendet wird, kann ein Kit mit DNase I zur Extraktion ausgewählt werden.Die an der Membran adsorbierte Nukleinsäure wird direkt mit DNase I verdaut, wodurch DNA-Reste stark reduziert werden können.
3. RNA-Abbau
Dabei kann es sich um die Zersetzung der extrahierten Probe selbst oder um die während des Extraktionsprozesses verursachte Zersetzung handeln.Soweit möglich sollten frische Proben für die RNA-Extraktion verwendet werden, und die gesammelten Proben sollten rechtzeitig in flüssigem Stickstoff oder im Kühlschrank bei -80 °C gelagert werden, und wiederholtes Einfrieren und Auftauen sollte vermieden werden.Für den RNA-Extraktionsprozess sollten RNase/DNase-freie Spitzen, Zentrifugenröhrchen und andere Materialien verwendet werden.Der Extraktionsprozess sollte so schnell wie möglich sein.Die extrahierte RNA sollte auf eine Eisbox gelegt und bei -80 °C gelagert werden.Wenn die extrahierte RNA durch Gelelektrophorese nachgewiesen werden muss, sollte die Elektrophorese unmittelbar nach der Extraktion durchgeführt und der Elektrophoresepuffer durch einen neu zubereiteten ersetzt werden.
4. Salz- und organische Lösungsmittelrückstände
Die Extraktionsreagenzien enthalten Phenol- und Guanidinsalze und die Waschlösung enthält Ethanol.Während des Extraktionsprozesses wurde das Lysat nicht vollständig absorbiert und verworfen und die Waschlösung wurde nicht vollständig getrocknet.Restsalze und organische Lösungsmittel sind schädlich für die anschließende Reverse Transkription und PCR.Da es unterschiedliche Hemmgrade gibt, sollte das Gewebelysat während des Extraktionsprozesses vollständig entfernt werden und die Spülung sollte ausreichend sein, damit die umgebenden Wände des Röhrchens gewaschen werden können.Darüber hinaus ist das Entleeren und Ausblasen des Rohrs ein notwendiger Schritt, der die Rückstände organischer Stoffe weiter reduziert.
Weitere Informationen zur RNA-Extraktion finden Sie auf unserer Website:
Weitere Informationen finden Sie unter www.foreivd.com.

Zeitpunkt der Veröffentlichung: 01.12.2022