Ausgangsmaterial: RNA
Die quantitative Reverse-Transkriptions-PCR (RT-qPCR) ist eine experimentelle Methode, die in PCR-Experimenten mit RNA als Ausgangsmaterial verwendet wird.Bei dieser Methode wird zunächst Gesamt-RNA oder Messenger-RNA (mRNA) durch Reverse Transkriptase in komplementäre DNA (cDNA) umgeschrieben.Anschließend wurde eine qPCR-Reaktion unter Verwendung der cDNA als Matrize durchgeführt.RT-qPCR wurde in einer Vielzahl molekularbiologischer Anwendungen eingesetzt, darunter Genexpressionsanalyse, RNA-Interferenzvalidierung, Microarray-Validierung, Pathogenerkennung, Gentests und Krankheitsforschung.
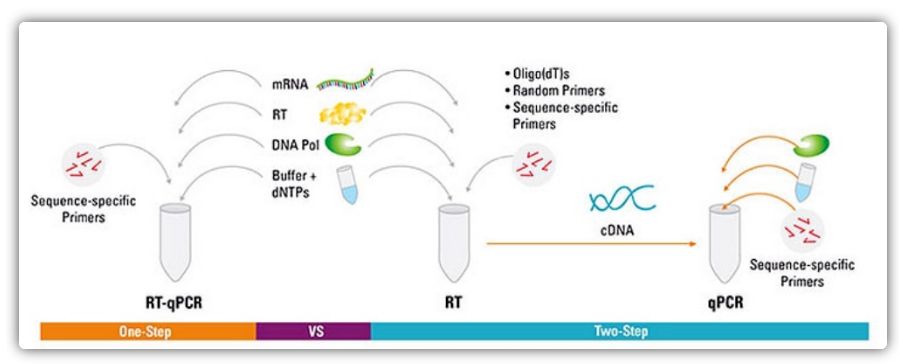
Ein- und zweistufige Methoden für RT-qPCR
RT-qPCR kann mit einer einstufigen oder zweistufigen Methode durchgeführt werden.Die einstufige RT-qPCR kombiniert reverse Transkription und PCR-Amplifikation und ermöglicht es Reverse Transkriptase und DNA-Polymerase, die Reaktion im selben Röhrchen unter denselben Pufferbedingungen abzuschließen.Für die einstufige RT-qPCR ist lediglich die Verwendung sequenzspezifischer Primer erforderlich.Bei der zweistufigen RT-qPCR werden die reverse Transkription und die PCR-Amplifikation in zwei Röhrchen unter Verwendung verschiedener optimierter Puffer, Reaktionsbedingungen und Primer-Designstrategien durchgeführt.
| Vorteil | Nachteil | |
| Ein Schritt | Diese Methode weist weniger experimentelle Fehler auf, da beide Reaktionen in einem Röhrchen durchgeführt werden
Weniger Pipettierschritte verringern das Kontaminationsrisiko
Geeignet für Amplifikation/Screening mit hohem Durchsatz, schnell und reproduzierbar | Zweistufige Reaktionen können nicht separat optimiert werden
Da die Reaktionsbedingungen durch die Kombination der zweistufigen Reaktion beeinträchtigt werden, ist die Empfindlichkeit nicht so gut wie die der zweistufigen Methode
Die Anzahl der von einer einzelnen Probe erfassten Ziele ist gering |
| Zwei schritte | Möglichkeit zur Erstellung stabiler cDNA-Bibliotheken, die über einen längeren Zeitraum gelagert und in mehreren Reaktionen verwendet werden können
Zielgene und Referenzgene können aus derselben cDNA-Bibliothek amplifiziert werden, ohne dass mehrere cDNA-Bibliotheken erforderlich sind
Reaktionspuffer und Reaktionsbedingungen, die die Optimierung einzelner Reaktionsläufe ermöglichen
Flexible Auswahl der Triggerbedingungen | Die Verwendung mehrerer Röhrchen und mehr Pipettierschritte erhöhen das Risiko einer DNA-Kontamination. und zeitaufwändig.
Erfordert mehr Optimierung als die einstufige Methode |
Verwandte Produkte:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
RT-qPCR Easyᵀᴹ (One Step)-Taqman
RT Easyᵀᴹ I Master Premix für die Erststrang-CDNA-Synthese
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Auswahl von Gesamt-RNA und mRNA
Beim Entwurf eines RT-qPCR-Experiments ist es wichtig zu entscheiden, ob Gesamt-RNA oder gereinigte mRNA als Vorlage für die reverse Transkription verwendet werden soll.Obwohl mRNA möglicherweise eine etwas höhere Empfindlichkeit bieten kann, wird Gesamt-RNA immer noch häufig verwendet.Der Grund dafür ist, dass Gesamt-RNA als Ausgangsmaterial einen größeren Vorteil hat als mRNA.Erstens erfordert der Prozess weniger Reinigungsschritte, was eine bessere quantitative Rückgewinnung der Vorlage und eine bessere Normalisierung der Ergebnisse auf die Ausgangszellzahlen gewährleistet.Zweitens wird der mRNA-Anreicherungsschritt vermieden, wodurch die Möglichkeit verzerrter Ergebnisse aufgrund unterschiedlicher Gewinne verschiedener mRNAs vermieden werden kann.Da in den meisten Anwendungen die relative Quantifizierung des Zielgens wichtiger ist als die absolute Empfindlichkeit des Nachweises, ist Gesamt-RNA in den meisten Fällen besser geeignet.
Reverse-Transkriptions-Primer
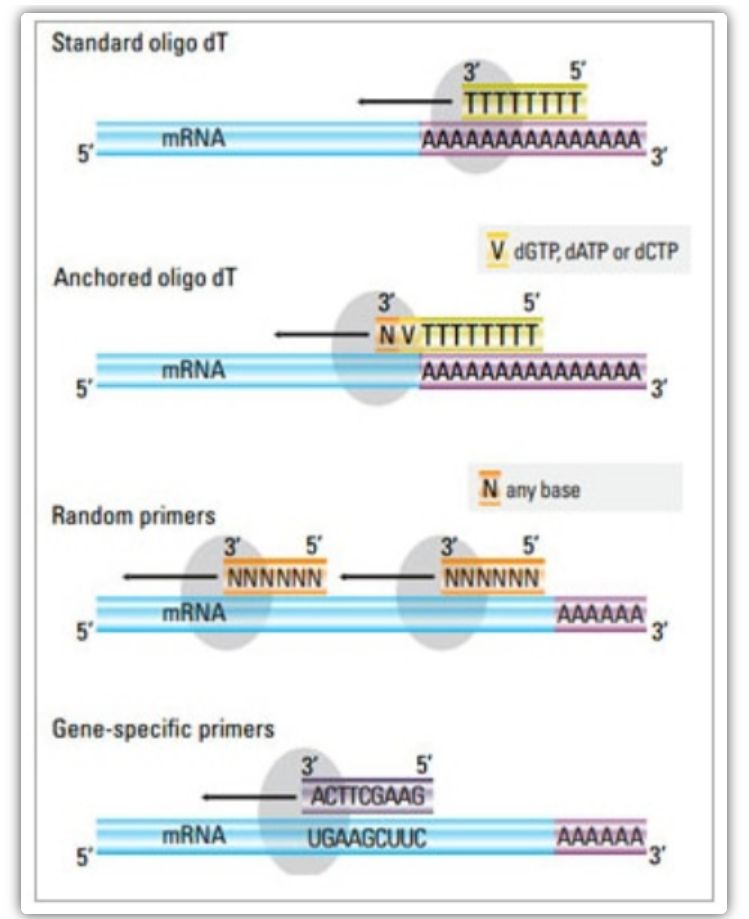
Bei der zweistufigen Methode können drei verschiedene Methoden zum Primen der cDNA-Reaktion verwendet werden: Oligo(dT)-Primer, Zufallsprimer oder sequenzspezifische Primer.Typischerweise werden Oligo(dT)-Primer und Zufallsprimer in Kombination verwendet.Diese Primer verbinden sich mit dem MRNA-Matrizenstrang und bieten der Reversen Transkriptase einen Ausgangspunkt für die Synthese.
| Primerauswahl | Struktur und Funktion | Vorteil | Nachteil |
| Oligo(dT)-Primer (oder verankerter Oligo(dT)-Primer) | Erweitertes Annealing an Thyminreste am Poly(A)-Schwanz der mRNA;Der Anker-Oligo(dT)-Primer enthält ein G, C oder A am 3′-Ende (Ankerstelle). | Synthese von cDNA voller Länge aus mRNA mit Poly(A)-Schwanz
Anwendbar, wenn weniger Ausgangsmaterial verfügbar ist
Die Verankerungsstelle stellt sicher, dass der Oligo(dT)-Primer an den 5′-Poly(A)-Schwanz der mRNA bindet | Nur zur Amplifikation von Genen mit Poly(A)-Schwänzen geeignet
Erhalten Sie von der Priming-Stelle*2 in Poly(A) verkürzte cDNA
Voreingenommen, um an das 3′-Ende zu binden*
*Diese Möglichkeit wird minimiert, wenn verankerte Oligo(dT)-Primer verwendet werden |
| Zufallsprimer
| 6 bis 9 Basen lang, die während der RNA-Transkription an mehrere Stellen anlagern können | Anneal an alle RNAs (tRNA, rRNA und mRNA)
Geeignet für Transkripte mit signifikanter Sekundärstruktur oder wenn weniger Ausgangsmaterial verfügbar ist
Hohe cDNA-Ausbeute | cDNA wird von der gesamten RNA revers transkribiert, was normalerweise unerwünscht ist und das Signal der Ziel-mRNA verdünnen kann
Erhalten Sie verkürzte cDNA |
| sequenzspezifische Primer | Benutzerdefinierte Primer, die auf bestimmte mRNA-Sequenzen abzielen | spezifische cDNA-Bibliothek
Verbessern Sie die Empfindlichkeit
Verwendung von Reverse-qPCR-Primern | Nur auf die Synthese eines einzelnen Zielgens beschränkt |
Umgekehrte Transkriptase
Reverse Transkriptase ist ein Enzym, das RNA zur Synthese von DNA verwendet.Einige Reverse Transkriptasen verfügen über RNase-Aktivität und können nach der Transkription RNA-Stränge in RNA-DNA-Hybridstränge abbauen.Wenn es keine RNase-Enzymaktivität aufweist, kann RNaseH für eine höhere qPCR-Effizienz hinzugefügt werden.Zu den häufig verwendeten Enzymen gehören die Reverse Transkriptase des Moloney-Maus-Leukämievirus und die Reverse Transkriptase des Vogel-Myeloblastomvirus.Für RT-qPCR ist es ideal, eine Reverse Transkriptase mit höherer Thermostabilität zu wählen, damit die cDNA-Synthese bei höheren Temperaturen durchgeführt werden kann, was eine erfolgreiche Transkription von RNAs mit höherer Sekundärstruktur gewährleistet und gleichzeitig ihre volle Aktivität während der Reaktion beibehält, was zu höheren cDNA-Ausbeuten führt.
Verwandte Produkte:
Foreasy M-MLV Reverse Transkriptase
RNase H-Aktivität der Reversen Transkriptase
RNaseH ist in der Lage, RNA-Stränge aus RNA-DNA-Duplexen abzubauen und so eine effiziente Synthese doppelsträngiger DNA zu ermöglichen.Wenn jedoch lange mRNA als Matrize verwendet wird, kann die RNA vorzeitig abgebaut werden, was zu verkürzter cDNA führt.Daher ist es oft von Vorteil, die RNaseH-Aktivität während der cDNA-Klonierung zu minimieren, wenn die Synthese langer Transkripte gewünscht wird.Im Gegensatz dazu sind Reverse Transkriptasen mit RNase H-Aktivität häufig für qPCR-Anwendungen von Vorteil, da sie das Schmelzen von RNA-DNA-Duplexen während des ersten PCR-Zyklus verstärken.
Primer-Design
PCR-Primer, die für den qPCR-Schritt in RT-qPCR verwendet werden, sollten idealerweise so konzipiert sein, dass sie eine Exon-Exon-Verbindung überbrücken, wobei ein Amplifikationsprimer möglicherweise eine tatsächliche Exon-Intron-Grenze überspannen könnte.Da intronhaltige genomische DNA-Sequenzen nicht amplifiziert werden, verringert dieses Design das Risiko falsch positiver Ergebnisse, die durch eine Kontamination der genomischen DNA amplifiziert werden.
Wenn Primer nicht zur Trennung von Exons oder Exon-Exon-Grenzen entwickelt werden können, kann es erforderlich sein, RNA-Proben mit RNase-freier DNase I oder dsDNase zu behandeln, um eine genomische DNA-Kontamination zu entfernen.
RT-qPCR-Kontrolle
In allen RT-qPCR-Experimenten sollte eine Reverse-Transkriptions-Negativkontrolle (-RT-Kontrolle) enthalten sein, um DNA-Kontaminationen (z. B. genomische DNA oder PCR-Produkte aus früheren Reaktionen) nachzuweisen.Diese Kontrolle enthält alle Reaktionskomponenten außer der Reversen Transkriptase.Da bei dieser Kontrolle keine reverse Transkription stattfindet, ist eine Kontamination durch DNA am wahrscheinlichsten, wenn eine PCR-Amplifikation beobachtet wird.
Zeitpunkt der Veröffentlichung: 02.08.2022








