Virales DNA- und RNA-Isolierungskit Virale DNA- und RNA-Extraktions-, Reinigungs- und Vorbereitungskits
Spezifikationen
50 Vorbereitungen, 200 Vorbereitungen
Das Viral RNA Nucleic Acid Purification Extraction Isolation Kit verwendet die von Foregene entwickelte Spin-Säule und Formel, mit der hochreine und qualitativ hochwertige virale RNA effizient aus Proben wie Plasma, Serum, zellfreier Körperflüssigkeit und Zellkulturüberstand extrahiert werden kann.Das Kit fügt speziell lineares Acrylamid hinzu, das problemlos kleine Mengen RNA aus den Proben einfangen kann.Die RNA-Only-Säule kann RNA effizient binden.Das Kit kann eine große Anzahl von Proben gleichzeitig verarbeiten.
Das gesamte Kit enthält keine RNase, sodass die gereinigte RNA nicht abgebaut wird.Puffer viRW1 und Puffer viRW2 können sicherstellen, dass die erhaltene virale Nukleinsäure frei von Proteinen, Nukleasen oder anderen Verunreinigungen ist und direkt für nachgelagerte molekularbiologische Experimente verwendet werden kann.
Kit-Komponenten
| Lineares Acrylamid |
| Puffer DRL |
| Puffer RW1, Puffer RW2 |
| RNase-freies ddH2O |
| DNA/RNA-Säule |
| Anweisungen |
Merkmale und Vorteile
■ Betrieb bei Raumtemperatur (15-25℃) während des gesamten Prozesses, ohne Eisbad und Zentrifugation bei niedriger Temperatur.
■ Komplettes Kit RNase-frei, kein Grund zur Sorge über RNA-Abbau.
■ Hohe Nukleinsäureausbeute: Nur DNA/RNA-Säule und einzigartige Formel können DNA und RNA effizient reinigen.
■ Große Probenverarbeitungskapazität: Es können jeweils bis zu 200 μl Proben verarbeitet werden.
■ Hohe Geschwindigkeit: einfach zu bedienen und innerhalb von 20 Minuten fertig.
■ Sicherheit: Es ist kein organisches Reagenz erforderlich.
■ Hohe Qualität: Die gereinigten RNA-Fragmente sind von hoher Reinheit, frei von Proteinen und anderen Verunreinigungen und können für verschiedene nachgelagerte experimentelle Anwendungen verwendet werden.
Kit-Anwendung
Es eignet sich für die Extraktion und Reinigung viraler Nukleinsäure in Proben wie Plasma, Serum, zellfreier Körperflüssigkeit und Zellkulturüberstand.
Arbeitsablauf
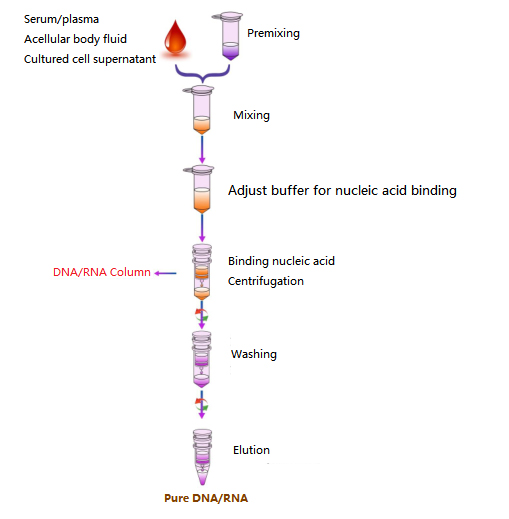
Diagramm
Lagerung und Haltbarkeit
■ Dieses Kit kann 24 Monate lang unter trockenen Bedingungen bei Raumtemperatur (15–25 °C) gelagert werden;Wenn es über einen längeren Zeitraum gelagert werden muss, kann es bei 2–8℃ gelagert werden.
■ Lineare Acrylamidlösung kann 7 Tage lang bei Raumtemperatur gelagert werden;Nachdem Sie das Kit erhalten haben, nehmen Sie es bitte heraus und lagern Sie es bei -20 °C.
■ Nach Zugabe von linearem Acrylamid zum DRL-Puffer kann es bis zu 48 Stunden bei 2–8 °C gelagert werden.Bitte nutzen Sie die fertige Lösung.
Leitfaden zur Problemanalyse
Im Folgenden finden Sie eine Analyse der Probleme, die bei der Extraktion viraler DNA/RNA auftreten können. Ich hoffe, dass sie für Ihre Experimente hilfreich ist.Darüber hinaus steht Ihnen bei anderen experimentellen oder technischen Problemen, die über die Bedienungsanleitung und Problemanalyse hinausgehen, ein spezieller technischer Support zur Verfügung.Wenn Sie Bedarf haben, kontaktieren Sie uns bitte: 028-83360257 oder E-Mail:
Tech@foregene.com.
Keine Nukleinsäureextraktion oder geringe Nukleinsäureausbeute
Normalerweise gibt es viele Faktoren, die die Rückgewinnungseffizienz beeinflussen, wie zum Beispiel: Nukleinsäuregehalt der Probe, Betriebsmethode, Elutionsvolumen usw.
Analyse häufiger Ursachen:
1. Während des Verfahrens wurde ein Eisbad oder eine Zentrifugation bei niedriger Temperatur (4 °C) durchgeführt.
Empfehlung: Während des gesamten Prozesses bei Raumtemperatur (15–25 °C) arbeiten, kein Eisbad und keine Zentrifugation bei niedriger Temperatur durchführen.
2. Die Probe wurde unsachgemäß oder zu lange gelagert.
Empfehlung: Proben bei -80 °C lagern und wiederholtes Einfrieren und Auftauen vermeiden;Versuchen Sie, frisch gesammelte Proben für die Nukleinsäureextraktion zu verwenden.
3. Unzureichende Probenlyse.
Empfehlung: Bitte stellen Sie sicher, dass Probe und Lyse-Arbeitslösung gründlich gemischt und 10 Minuten lang bei Raumtemperatur (15–25 °C) inkubiert werden.
4. Falsche Eluentenzugabe.
Vorschlag: Stellen Sie sicher, dass RNase-freies ddH2O tropfenweise in die Mitte der Membran der Reinigungssäule gegeben wird und lassen Sie es nicht auf den Ring der Reinigungssäule fallen.
5. Dem Puffer RW2 wurde nicht das richtige Volumen an absolutem Ethanol zugesetzt.
Vorschlag: Bitte befolgen Sie die Anweisungen, geben Sie das richtige Volumen an absolutem Ethanol zum Puffer RW2 und mischen Sie es gut, bevor Sie das Kit verwenden.
6. Unangemessenes Probenvolumen.
Vorschlag: Pro 500 µl Puffer DRL werden 200 µl Probe verarbeitet.Eine übermäßige Probenverarbeitung führt zu einer geringeren Nukleinsäureextraktionsausbeute.
7. Unangemessenes Elutionsvolumen oder unvollständige Elution.
Empfehlung: Das Eluentenvolumen der Reinigungssäule beträgt 30-50μl;Wenn der Elutionseffekt nicht zufriedenstellend ist, wird empfohlen, die Zeit bei Raumtemperatur nach Zugabe von vorgewärmtem RNase-freiem ddH2O zu verlängern, beispielsweise um 5–10 Minuten.
8. Nach dem Waschen mit Puffer RW2 verbleibt Ethanol auf der Säule.
Vorschlag: Wenn nach der Zentrifugation mit Puffer RW2 für 2 Minuten noch Ethanol zurückbleibt, kann die Säule nach der Zentrifugation für 5 Minuten bei Raumtemperatur stehen, um restliches Ethanol vollständig zu entfernen.
Die gereinigte Nukleinsäure wird abgebaut
Die Qualität der gereinigten Nukleinsäure hängt von der Konservierung der Probe, der RNase-Kontamination, dem Betrieb und anderen Faktoren ab.Analyse häufiger Ursachen:
1. Die gesammelten Proben wurden nicht rechtzeitig gelagert.
Vorschlag: Wenn die Probe nicht rechtzeitig nach der Entnahme verwendet wird, lagern Sie sie bitte sofort bei -80 °C und niedriger Temperatur.Versuchen Sie für die RNA-Extraktion, frisch gesammelte Proben zu verwenden.
2. Proben sammeln und wiederholt einfrieren und auftauen.
Empfehlung: Vermeiden Sie das Einfrieren und Auftauen (nicht mehr als einmal) während der Probenentnahme und -lagerung, da sonst die Nukleinsäureausbeute verringert wird.
3. RNase wird im Operationssaal eingeführt oder es werden keine Einweghandschuhe, Masken usw. getragen.
Empfehlung: RNA-Extraktionsexperimente werden am besten in einem separaten RNA-Operationsraum durchgeführt und der Labortisch sollte vor dem Experiment gereinigt werden.
Tragen Sie während des Experiments Einweghandschuhe und -masken, um den durch die Einführung von RNase verursachten RNA-Abbau weitestgehend zu vermeiden.
4. Das Reagenz wurde während der Verwendung mit RNase kontaminiert.
Empfehlung: Für entsprechende Experimente durch ein neues Viral DNA/RNA Isolation Kit ersetzen.
5. Die zur RNA-Manipulation verwendeten Zentrifugenröhrchen und Pipettenspitzen sind mit RNase kontaminiert.
Vorschlag: Stellen Sie sicher, dass die für die RNA-Extraktion verwendeten Zentrifugenröhrchen, Pipettenspitzen, Pipetten usw. alle RNase-frei sind.
Gereinigte Nukleinsäure wirkt sich auf nachgelagerte Experimente aus
Durch die Reinigungssäule gereinigte DNA und RNA. Wenn der Salzionen- und Proteingehalt zu hoch ist, wirkt sich dies auf die nachgeschalteten Experimente aus, z. B. PCR-Amplifikation, reverse Transkription usw.
1. Die eluierte DNA und RNA weisen Restsalzionen auf.
Vorschlag: Stellen Sie sicher, dass dem Puffer RW2 das richtige Volumen an absolutem Ethanol zugesetzt wird, und waschen Sie die Reinigungssäule zweimal mit der in der Bedienungsanleitung angegebenen Zentrifugationsgeschwindigkeit;Führen Sie eine Zentrifugation durch, um die Kontamination mit Salzionen zu minimieren.
2. Die eluierte DNA und RNA weisen Ethanolreste auf.
Vorschlag: Nachdem Sie das Waschen mit Puffer RW2 bestätigt haben, führen Sie eine Zentrifugation des leeren Röhrchens mit der in der Bedienungsanleitung angegebenen Zentrifugationsgeschwindigkeit durch;Sollten noch Ethanolrückstände vorhanden sein, können Sie das leere Röhrchen zentrifugieren und anschließend für 5 Minuten bei Raumtemperatur stehen lassen, um die Ethanolrückstände weitestgehend zu entfernen.
Bedienungsanleitung:
Bedienungsanleitung für das virale DNA- und RNA-Isolierungskit