Einige revolutionäre Erfindungen in der Geschichte der Nachweistechnologie sind meiner Meinung nach die Immunmarkierungstechnologie, die auf dem Prinzip der Antigen-Antikörper-spezifischen Bindung basiert, die PCR-Technologie und die Sequenzierungstechnologie.Heute werden wir über die PCR-Technologie sprechen.Entsprechend der Entwicklung der PCR-Technologie wird die PCR-Technologie üblicherweise in drei Generationen eingeteilt: gewöhnliche PCR-Technologie, fluoreszierende quantitative Echtzeit-PCR-Technologie und digitale PCR-Technologie.
CGemeinsame PCR-Technik

KARY MULLIS (1944.12.28-2019.8.7)
Kary Mullis erfand 1983 die Polymerase-Kettenreaktion (Polymerase-Kettenreaktion, PCR). Es heißt, dass er, als er mit seiner Freundin Auto fuhr, plötzlich einen Geistesblitz hatte und an das Prinzip der PCR (über die Vorteile des Autofahrens) dachte.Kary Mullis wurde 1993 mit dem Nobelpreis für Chemie ausgezeichnet. Die New York Times kommentierte: „Sehr originell und bedeutsam, es teilt die Biologie fast in Prä-PCR- und Post-PCR-Ära.“
Das Prinzip der PCR: Unter der Katalyse der DNA-Polymerase wird die Mutterstrang-DNA als Matrize und der spezifische Primer als Ausgangspunkt der Verlängerung verwendet, und die zur Mutterstrang-Matrizen-DNA komplementäre Tochterstrang-DNA wird in vitro durch Denaturierung, Annealing, Verlängerung und andere Schritte kopiert.Es handelt sich um eine In-vitro-Technologie zur DNA-Synthese-Amplifikation, mit der jede Ziel-DNA in vitro schnell und spezifisch amplifiziert werden kann.
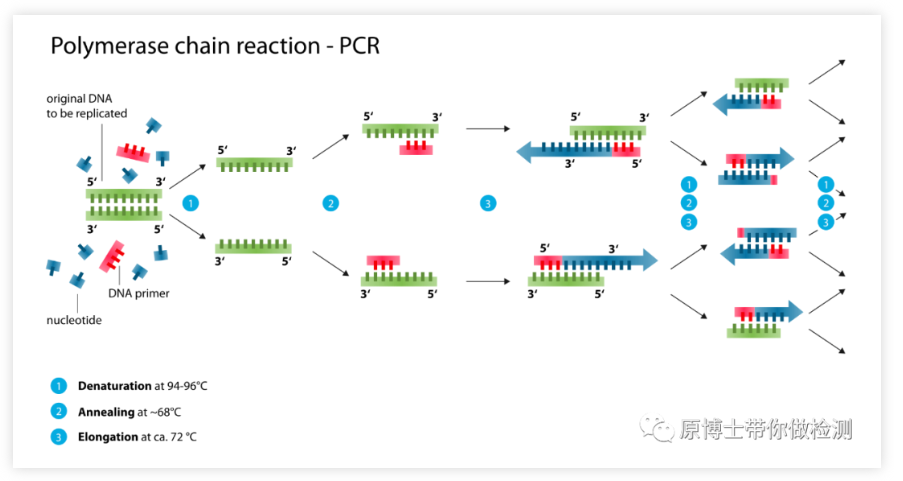
Vorteile der gewöhnlichen PCR
1.Klassische Methode, vollständige internationale und nationale Standards
2.Geringere Kosten für Instrumentenreagenzien
3.PCR-Produkte können für andere molekularbiologische Experimente zurückgewonnen werden
Empfohlenes Foregene-PCR-Gerät: https://www.foreivd.com/foreamp-sn-695-series-thermal-cycler-96-wells-pcr-machine-product/
Verwandte Produkte: https://www.foreivd.com/pcr-herotm-with-dye-product/
Nachteile der gewöhnlichen PCR
1.leicht zu verschmutzen
2.umständliche Bedienung
3.nur qualitative Analyse
4.Mäßige Empfindlichkeit
5.Es liegt eine unspezifische Amplifikation vor, und wenn die unspezifische Bande die gleiche Größe wie die Zielbande hat, kann sie nicht unterschieden werden
CApillarelektrophorese-basierte PCR
Als Reaktion auf die Mängel der gewöhnlichen PCR haben einige Hersteller Geräte eingeführt, die auf dem Prinzip der Kapillarelektrophorese basieren.Der Elektrophoreseschritt nach der PCR-Amplifikation ist in der Kapillare abgeschlossen.Die Empfindlichkeit ist höher, der Unterschied mehrerer Basen kann unterschieden und die Amplifikation von MAERKER berechnet werden.Produktinhalt.Der Nachteil besteht darin, dass das PCR-Produkt noch geöffnet und in das Gerät gegeben werden muss und weiterhin ein großes Kontaminationsrisiko besteht.

CapillarEElektrophorese
2. Echtzeit-fluoreszierende quantitative PCR-Technologie (Quantitative Real-time PCR, qPCR).Die quantitative Fluoreszenz-PCR, auch Real-Time-PCR genannt, ist eine neue quantitative Nukleinsäuretechnologie, die 1995 von PE (Perkin Elmer) entwickelt wurde. Die Entwicklungsgeschichte der quantitativen Fluoreszenz-PCR ist eine Geschichte erschütternder Kämpfe von Giganten wie ABI, Roche und Bio-Rad.Wenn Sie interessiert sind, können Sie es sich ansehen.Diese Technik ist derzeit die ausgereifteste und am weitesten verbreitete semiquantitative PCR-Technik.
Empfohlene qPCR-Maschine:https://www.foreivd.com/mini-real-time-pcr-system-forequant-sf2sf4-product/
Fluoreszenzfarbstoffmethode (SYBR Green I):SYBR Green I ist der am häufigsten verwendete DNA-Bindungsfarbstoff für die quantitative PCR, der unspezifisch an doppelsträngige DNA bindet.Im freien Zustand emittiert SYBR Green eine schwache Fluoreszenz, aber sobald es an doppelsträngige DNA gebunden ist, erhöht sich seine Fluoreszenz um das 1000-fache.Daher ist das gesamte von einer Reaktion emittierte Fluoreszenzsignal proportional zur Menge der vorhandenen doppelsträngigen DNA und nimmt mit der Zunahme des amplifizierten Produkts zu.Da der Farbstoff unspezifisch an doppelsträngige DNA bindet, kann es zu falsch positiven Ergebnissen kommen.
Verwandte Produkte: https://www.foreivd.com/real-time-pcr-easytm-sybr-green-i-kit-product/
Fluoreszenzsondenmethode (Taqman-Technologie): WährendBei der PCR-Amplifikation wird eine spezifische Fluoreszenzsonde gleichzeitig mit einem Primerpaar hinzugefügt.Die Sonde ist ein lineares Oligonukleotid mit einer fluoreszierenden Reportergruppe und einer fluoreszierenden Quenchergruppe, die an beiden Enden markiert sind.Wenn die Sonde intakt ist, wird das von der Reportergruppe emittierte Fluoreszenzsignal von der Quenchergruppe absorbiert und es erfolgt kein Fluoreszenzsignal;Während der PCR-Amplifikation (in der Erweiterungsphase) wird die 5'-3'-Dicer-Aktivität des Taq-Enzyms die Sonde verdauen und abbauen, so dass die Reporter-Fluoreszenzgruppe und die Quencher-Fluoreszenzgruppe getrennt werden, so dass das Fluoreszenzüberwachungssystem das Fluoreszenzsignal empfangen kann, d.Die Taqman-Sondenmethode ist die am häufigsten verwendete Nachweismethode im klinischen Nachweis.
Verwandte Produkte: https://www.foreivd.com/quickeasy%e1%b5%80%e1%b4%b9-real-time-pcr-kit-taqman-product/
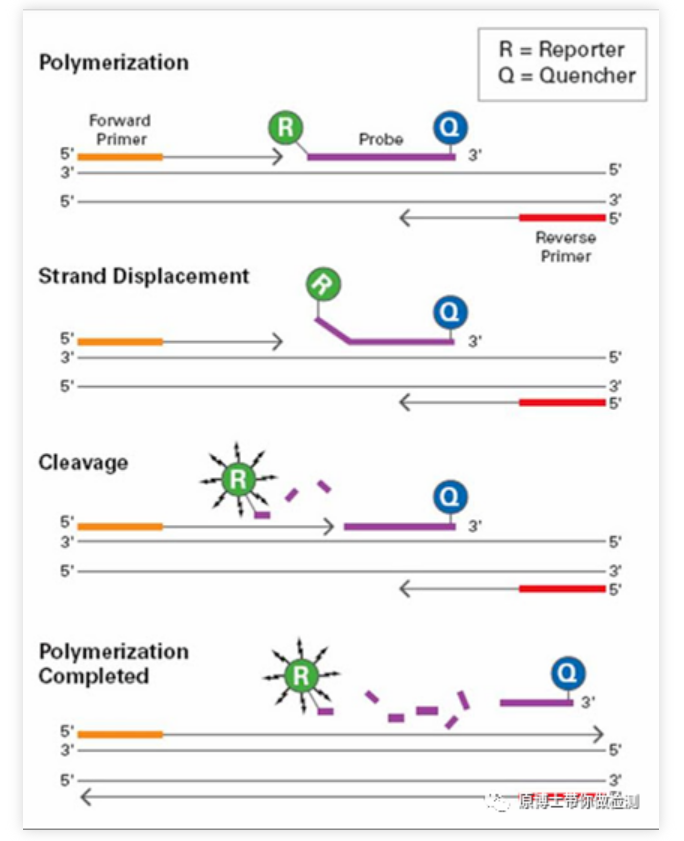
Vorteile von qPCR
1.Die Methode ist ausgereift und die unterstützende Ausrüstung und Reagenzien sind vollständig
2.Mittlere Kosten für Reagenzien
3.Einfach zu verwenden
4.Hohe Nachweisempfindlichkeit und Spezifität
Nachteile von qPCR
Die Mutation des Zielgens führt zu einer Fehlerkennung.
Das Nachweisergebnis einer Vorlage mit niedriger Konzentration kann nicht bestimmt werden.
Bei der Verwendung der Standardkurve für den quantitativen Nachweis tritt ein großer Fehler auf.
3. Digitale PCR-Technologie (digitale PCR, dPCR).
Die digitale PCR ist eine Technik zur absoluten Quantifizierung von Nukleinsäuremolekülen.Im Vergleich zur qPCR kann die digitale PCR direkt die Anzahl der DNA/RNA-Moleküle ablesen, also die absolute Quantifizierung der Nukleinsäuremoleküle in der Ausgangsprobe.Im Jahr 1999 schlugen Bert Vogelstein und Kenneth W. Kinzler offiziell das Konzept der dPCR vor.
Im Jahr 2006 war Fluidigm das erste Unternehmen, das ein kommerzielles dPCR-Gerät auf Chipbasis herstellte.Im Jahr 2009 brachte Life Technologies die dPCR-Systeme OpenArray und QuantStudio 12K Flex auf den Markt.Im Jahr 2013 brachte Life Technologies das QuantStudio 3DdPCR-System auf den Markt, das hochdichte nanoskalige Mikrofluidik-Chiptechnologie nutzt, um Proben gleichmäßig auf 20.000 einzelne Zellen zu verteilen.im Reaktionsbrunnen.
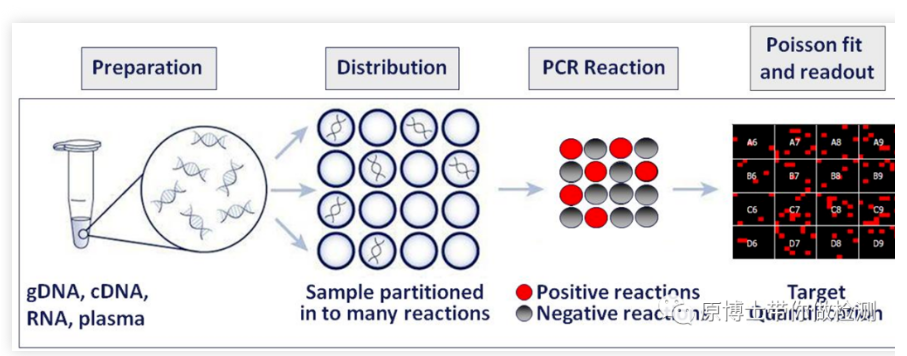
Im Jahr 2011 brachte Bio-Rad das tröpfchenbasierte QX100 dPCR-Gerät auf den Markt, das die Wasser-in-Öl-Technologie nutzt, um die Probe gleichmäßig auf 20.000 Wasser-in-Öl-Tröpfchen zu verteilen, und einen Tröpfchenanalysator zur Analyse der Tröpfchen verwendet.Im Jahr 2012 brachte RainDance das mit Hochdruckgas betriebene RainDrop dPCR-Instrument auf den Markt, um jedes Standardreaktionssystem in eine Reaktionsemulsion aufzuteilen, die 1 Million bis 10 Millionen Mikrotröpfchen auf Picoliter-Ebene enthält.
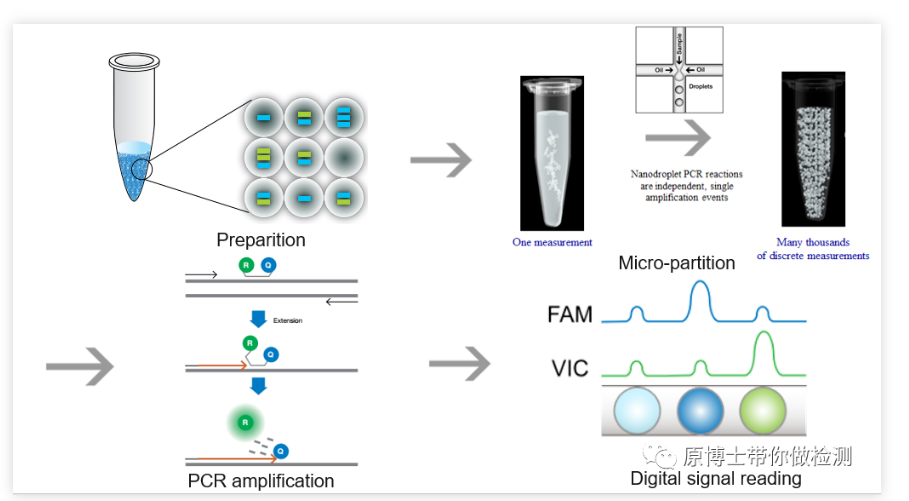
Bisher hat die digitale PCR zwei große Fraktionen gebildet: den Chip-Typ und den Tröpfchen-Typ.Unabhängig von der Art der digitalen PCR sind ihre Kernprinzipien die Begrenzung der Verdünnung, die Endpunkt-PCR und die Poisson-Verteilung.Das Standard-PCR-Reaktionssystem, das Nukleinsäure-Templates enthält, ist gleichmäßig in Zehntausende PCR-Reaktionen unterteilt, die auf Chips oder Mikrotröpfchen verteilt werden, sodass jede Reaktion so viel wie möglich ein Template-Molekül enthält und eine Einzelmolekül-Template-PCR-Reaktion durchgeführt wird.Durch Ablesen der Fluoreszenz wird das Vorhandensein oder Fehlen des Signals gezählt und die absolute Quantifizierung erfolgt nach Kalibrierung der statistischen Poisson-Verteilung.
Im Folgenden sind die Merkmale mehrerer digitaler PCR-Plattformen aufgeführt, die ich verwendet habe:
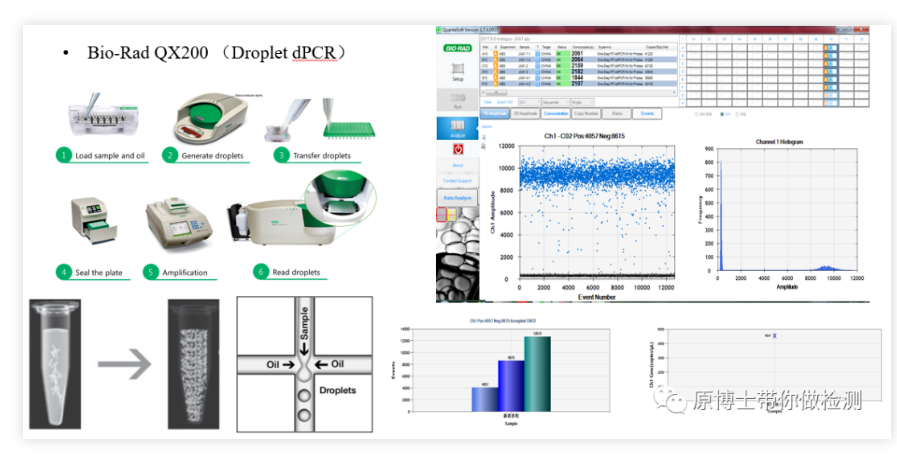
1. Bio-Rad QX200 Tröpfchen-Digital-PCR Bio-RadQX200 ist eine sehr klassische digitale PCR-Plattform, der grundlegende Nachweisprozess: 20.000 Proben werden vom Tröpfchengenerator erzeugt. Wasser-in-Öl-Mikrotröpfchen werden auf einem gewöhnlichen PCR-Gerät verstärkt, und schließlich wird das Fluoreszenzsignal jedes Mikrotröpfchens von einem Mikrotröpfchen-Lesegerät gelesen.Der Vorgang ist komplizierter und das Risiko einer Verschmutzung ist mittel.
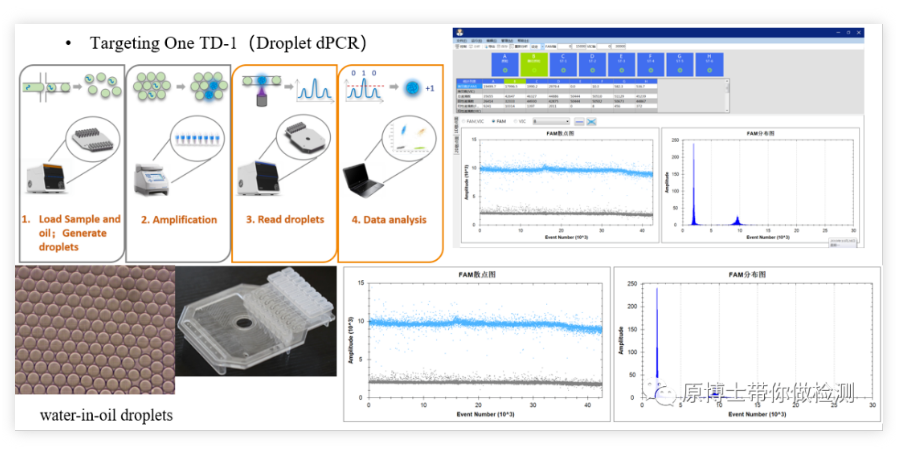
Xinyi TD1 Mikrotröpfchen-Digital-PCRXinyi TD1 ist eine inländische digitale PCR-Plattform, der grundlegende Nachweisprozess: 30.000–50.000 Wasser-in-Öl-Tröpfchen durch einen Tröpfchengenerator erzeugen, auf einem herkömmlichen PCR-Instrument verstärken und schließlich passieren. Der Tröpfchenleser liest das Fluoreszenzsignal jedes Tröpfchens.Sowohl die Tröpfchenerzeugung als auch das Lesen erfolgen bei dieser Plattform in einem speziellen Chip mit geringem Kontaminationsrisiko.
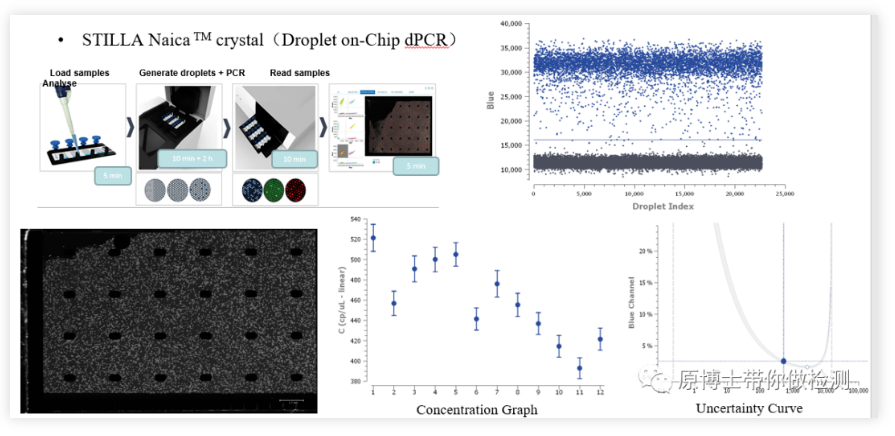
STILLA Naica Mikrotröpfchen-Chip für digitale PCRSTILLA Naica ist eine relativ neue digitale PCR-Plattform.Der grundlegende Erkennungsprozess besteht darin, die Reaktionslösung auf den Chip zu geben, den Chip in das Mikrotröpfchen-Erzeugungs- und -Verstärkungssystem zu geben und 30.000 Mikrotröpfchen zu erzeugen.Auf dem Chip verteilen und die PCR-Amplifikation auf dem Chip abschließen.Dann wird der verstärkte Chip an das Mikrotröpfchen-Leseanalysesystem übertragen und das Fluoreszenzsignal wird durch Aufnehmen von Bildern gelesen.Da der gesamte Prozess in einem geschlossenen Span stattfindet, ist die Gefahr einer Kontamination gering.
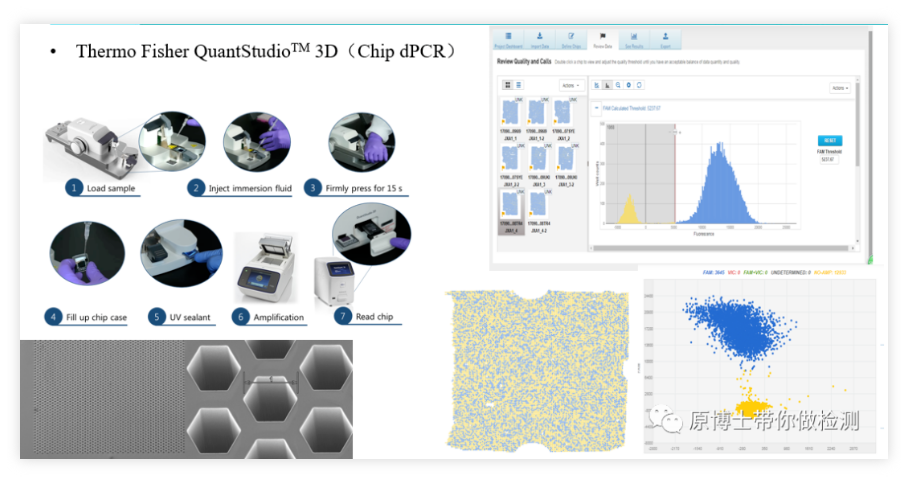
4. Digitale PCR mit ThermoFisher QuantStudio 3D-Chip
ThermoFisher QuantStudio 3D ist eine weitere klassische Chip-basierte digitale PCR-Plattform.Der grundlegende Erkennungsprozess besteht darin, die Reaktionslösung in den Streuer zu geben und die Reaktionslösung durch den Streuer gleichmäßig auf dem Chip mit 20.000 Mikrovertiefungen zu verteilen., setzen Sie den Chip zur Verstärkung auf das PCR-Gerät und stecken Sie schließlich den Chip in das Lesegerät und machen Sie ein Foto, um das Fluoreszenzsignal abzulesen.Der Vorgang ist relativ kompliziert, der gesamte Prozess erfolgt in einem geschlossenen Chip und die Kontaminationsgefahr ist gering.
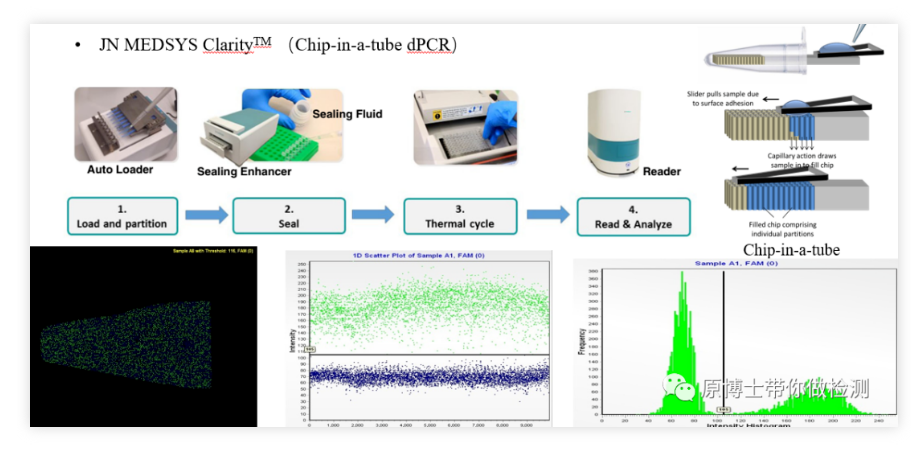
5. JN MEDSYS Clarity Chip digitale PCR
JN MEDSYS Clarity ist eine relativ neue digitale PCR-Plattform vom Chiptyp.Der grundlegende Nachweisprozess besteht darin, die Reaktionslösung in den Applikator zu geben und die Reaktionslösung durch den Applikator gleichmäßig auf 10.000 PCR-Röhrchen zu verteilen, die im PCR-Röhrchen fixiert sind.Auf dem mikroporösen Chip dringt die Reaktionslösung durch Kapillarwirkung in den Chip ein, und das PCR-Röhrchen mit dem Chip wird zur Amplifikation auf das PCR-Gerät gestellt, und schließlich wird der Chip in das Lesegerät eingesetzt, um das Fluoreszenzsignal durch Aufnahme eines Fotos auszulesen.Die Bedienung ist komplizierter.Das Kontaminationsrisiko ist gering.
Die Parameter jeder digitalen PCR-Plattform sind wie folgt zusammengefasst:
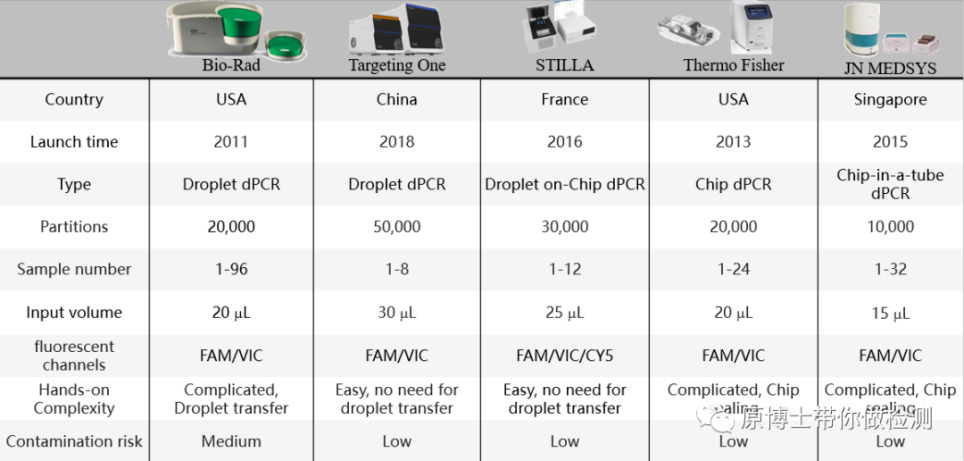
Die Bewertungsindikatoren der digitalen PCR-Plattform sind: die Anzahl der Split-Einheiten, die Anzahl der Fluoreszenzkanäle, die Komplexität der Bedienung und das Kontaminationsrisiko.Aber das Wichtigste ist die Erkennungsgenauigkeit.Eine Möglichkeit zur Bewertung digitaler PCR-Plattformen besteht darin, mehrere digitale PCR-Plattformen zur gegenseitigen Verifizierung zu verwenden, eine andere Möglichkeit besteht darin, Standardsubstanzen mit genauen Werten zu verwenden.
Vorteile von dPCR
1.Absolute Quantifizierung erreichen
2.Höhere Sensitivität und Spezifität
3.Kann Proben mit geringer Kopienzahl erkennen
Nachteile von dPCR1. Teure Ausrüstung und Reagenzien 2. Komplizierter Betrieb und lange Nachweiszeit 3. Enger Nachweisbereich
Gegenwärtig haben die drei Generationen der PCR-Technologie ihre eigenen Vor- und Nachteile, und jede hat ihre eigenen Anwendungsbereiche, und es besteht keine Beziehung dazu, dass eine Generation die andere ersetzt.Die kontinuierliche Weiterentwicklung der Technologie hat der PCR-Technologie neue Dynamik verliehen und ermöglicht es ihr, eine Anwendungsrichtung nach der anderen freizuschalten, wodurch der Nachweis von Nukleinsäuren bequemer und genauer wird.
Quelle: Dr. Yuan nimmt Sie mit zum Test
Empfohlene Produkte:
Zeitpunkt der Veröffentlichung: 18. November 2022











