Die molekulare Diagnosetechnologie verwendet molekularbiologische Methoden, um die Expression und Struktur des genetischen Materials des menschlichen Körpers und verschiedener Krankheitserreger zu erfassen und so den Zweck der Vorhersage und Diagnose von Krankheiten zu erreichen.
In den letzten Jahren ist die klinische Anwendung der Molekulardiagnostik mit der Modernisierung und Weiterentwicklung der Molekulardiagnostiktechnologie immer umfassender und tiefgreifender geworden, und der Markt für Molekulardiagnostik ist in eine Phase rasanter Entwicklung eingetreten.
Der Autor fasst die gängigen molekulardiagnostischen Technologien auf dem Markt zusammen und ist in drei Teile gegliedert: Im ersten Teil wird die PCR-Technologie vorgestellt, im zweiten Teil die isotherme Nukleinsäureamplifikationstechnologie und im zweiten Teil die Sequenzierungstechnologie.
01
Teil I: PCR-Technologie
PCR-Technologie
PCR (Polymerase-Kettenreaktion) ist eine der In-vitro-DNA-Amplifikationstechnologien mit einer mehr als 30-jährigen Geschichte.
Die PCR-Technologie wurde 1983 von Kary Mullis aus Cetus, USA, entwickelt.Mullis meldete 1985 ein PCR-Patent an und veröffentlichte im selben Jahr die erste wissenschaftliche PCR-Arbeit zum Thema Wissenschaft.Mullis erhielt 1993 den Nobelpreis für Chemie.
Grundprinzipien der PCR
PCR kann Ziel-DNA-Fragmente um mehr als eine Million Mal verstärken.Das Prinzip besteht darin, dass bei der Katalyse der DNA-Polymerase die Mutterstrang-DNA als Matrize und ein spezifischer Primer als Ausgangspunkt für die Verlängerung verwendet wird.Es wird in vitro durch Schritte wie Denaturierung, Annealing und Extension repliziert.Der Prozess der Tochterstrang-DNA, die zur Mutterstrang-Matrizen-DNA komplementär ist.
Der Standard-PCR-Prozess ist in drei Schritte unterteilt:
1. Denaturierung: Verwenden Sie hohe Temperaturen, um DNA-Doppelstränge zu trennen.Die Wasserstoffbrückenbindungen zwischen DNA-Doppelsträngen werden bei hohen Temperaturen (93–98 °C) aufgebrochen.
2. Annealing: Nach der Trennung der doppelsträngigen DNA wird die Temperatur gesenkt, damit der Primer an die einzelsträngige DNA binden kann.
3. Verlängerung: Die DNA-Polymerase beginnt mit der Synthese komplementärer Stränge entlang der DNA-Stränge aus den gebundenen Primern, wenn die Temperatur gesenkt wird.Wenn die Verlängerung abgeschlossen ist, ist ein Zyklus abgeschlossen und die Anzahl der DNA-Fragmente verdoppelt sich.
Durch 25- bis 35-maliges Durchführen dieser drei Schritte erhöht sich die Anzahl der DNA-Fragmente exponentiell.
Der Einfallsreichtum der PCR besteht darin, dass unterschiedliche Primer für unterschiedliche Zielgene entwickelt werden können, sodass die Zielgenfragmente in kurzer Zeit amplifiziert werden können.
Bisher lässt sich die PCR in drei Kategorien einteilen, nämlich gewöhnliche PCR, fluoreszierende quantitative PCR und digitale PCR.
Die erste Generation der gewöhnlichen PCR
Verwenden Sie ein gewöhnliches PCR-Amplifikationsgerät, um das Zielgen zu amplifizieren, und verwenden Sie dann eine Agarosegelelektrophorese, um das Produkt nachzuweisen. Es kann nur eine qualitative Analyse durchgeführt werden.
Die Hauptnachteile der PCR der ersten Generation:
-Anfällig für unspezifische Amplifikation und falsch positive Ergebnisse.
-Die Erkennung dauert lange und die Bedienung ist umständlich.
-Es können nur qualitative Tests durchgeführt werden.
Quantitative Fluoreszenz-PCR der zweiten Generation
Die quantitative Fluoreszenz-PCR (Echtzeit-PCR), auch als qPCR bekannt, wird verwendet, um die Ansammlung amplifizierter Produkte durch die Ansammlung von Fluoreszenzsignalen durch Zugabe von Fluoreszenzsonden zu überwachen, die den Fortschritt des Reaktionssystems anzeigen können, und um die Ergebnisse anhand der Fluoreszenzkurve zu beurteilen und mithilfe des Cq-Werts und der Standardkurve zu quantifizieren.
Da die qPCR-Technologie in einem geschlossenen System durchgeführt wird, verringert sich die Wahrscheinlichkeit einer Kontamination und das Fluoreszenzsignal kann für den quantitativen Nachweis überwacht werden. Daher ist sie in der klinischen Praxis am weitesten verbreitet und hat sich zur dominierenden Technologie in der PCR entwickelt.
Die in der quantitativen Echtzeit-Fluoreszenz-PCR verwendeten fluoreszierenden Substanzen können unterteilt werden in: TaqMan-Fluoreszenzsonden, Molecular Beacons und Fluoreszenzfarbstoffe.
1) TaqMan-Fluoreszenzsonde:
Bei der PCR-Amplifikation wird eine spezifische Fluoreszenzsonde hinzugefügt, während gleichzeitig ein Primerpaar hinzugefügt wird.Die Sonde ist ein Oligonukleotid und die beiden Enden sind jeweils mit einer Reporter-Fluoreszenzgruppe und einer Quencher-Fluoreszenzgruppe markiert.
Wenn die Sonde intakt ist, wird das von der Reportergruppe emittierte Fluoreszenzsignal von der Löschgruppe absorbiert;Während der PCR-Amplifikation spaltet und zersetzt die 5′-3′-Exonukleaseaktivität des Taq-Enzyms die Sonde, wodurch die Reporter-Fluoreszenzgruppe und der Quencher entstehen. Die Fluoreszenzgruppe wird getrennt, so dass das Fluoreszenzüberwachungssystem das Fluoreszenzsignal empfangen kann, d.
2) SYBR-Fluoreszenzfarbstoffe:
Im PCR-Reaktionssystem wird ein Überschuss an SYBR-Fluoreszenzfarbstoff zugesetzt.Nachdem der SYBR-Fluoreszenzfarbstoff unspezifisch in den DNA-Doppelstrang eingebaut wurde, sendet er ein Fluoreszenzsignal aus.Das nicht in die Kette eingebaute SYBR-Farbstoffmolekül emittiert kein Fluoreszenzsignal, wodurch sichergestellt wird, dass das Fluoreszenzsignal vollständig mit der Zunahme der PCR-Produkte synchronisiert ist.SYBR bindet nur an doppelsträngige DNA, sodass anhand der Schmelzkurve festgestellt werden kann, ob die PCR-Reaktion spezifisch ist.
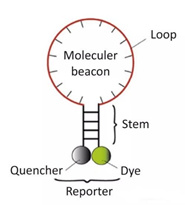
3) Molekulare Beacons
Es handelt sich um eine doppelt markierte Stamm-Schleifen-Oligonukleotidsonde, die an den Enden 5 und 3 eine Haarnadelstruktur von etwa 8 Basen bildet.Die Nukleinsäuresequenzen an beiden Enden sind komplementär gepaart, wodurch die fluoreszierende Gruppe und die Löschgruppe eng beieinander liegen.Schließen Sie es, es wird keine Fluoreszenz erzeugt.
Nachdem das PCR-Produkt erzeugt wurde, wird während des Annealing-Prozesses der mittlere Teil des Molecular Beacon mit einer spezifischen DNA-Sequenz gepaart und das Fluoreszenzgen vom Quencher-Gen getrennt, um Fluoreszenz zu erzeugen.
Die Hauptnachteile der PCR der zweiten Generation:
Es fehlt immer noch an Empfindlichkeit und die Erkennung von Proben mit geringer Kopienzahl ist nicht genau.
Es besteht ein Hintergrundwerteinfluss und das Ergebnis ist störanfällig.
Digitale PCR der dritten Generation
Die digitale PCR (DigitalPCR, dPCR, Dig-PCR) berechnet die Kopienzahl der Zielsequenz durch Endpunktdetektion und kann eine genaue absolute quantitative Detektion ohne Verwendung interner Kontrollen und Standardkurven durchführen.
Die digitale PCR nutzt die Endpunkterkennung und ist nicht vom Ct-Wert (Zyklusschwelle) abhängig, sodass die digitale PCR-Reaktion weniger von der Amplifikationseffizienz beeinflusst wird und die Toleranz gegenüber PCR-Reaktionsinhibitoren bei hoher Genauigkeit und Reproduzierbarkeit verbessert wird.
Aufgrund der Eigenschaften hoher Empfindlichkeit und hoher Genauigkeit wird es nicht leicht durch PCR-Reaktionsinhibitoren beeinträchtigt und kann ohne Standardprodukte eine echte absolute Quantifizierung erreichen, was zu einem Forschungs- und Anwendungs-Hotspot geworden ist.
Entsprechend den unterschiedlichen Formen der Reaktionseinheit kann diese in drei Typen unterteilt werden: Mikrofluidik-, Chip- und Tröpfchensysteme.
Zeitpunkt der Veröffentlichung: 08.07.2021