PCR, mehrere PCR, In-situ-PCR, Reverse PCR, RT-PCR, qPCR(1)–PCR
Wir werden die Konzepte, Schritte und Details verschiedener PCR klären
Ⅰ. PCR
Die Polymerase-Kettenreaktion, auch PCR genannt, ist eine molekularbiologische Technologie, die zur Vergrößerung spezifischer DNA-Fragmente eingesetzt wird.Es kann als eine spezielle DNA-Replikation in vitro angesehen werden.DNA-Polymerase (DNA-Polymerase I) wurde bereits 1955 entdeckt, und das Klenow-Fragment von E. Coli, das experimentellen Wert und praktische Anwendbarkeit hat, wurde Anfang der 1970er Jahre von Dr.Die heute verwendeten Enzyme (Taq-Polymerase genannt) wurden 1976 aus Thermus aquaticus, einem Thermalquellenbakterium, isoliert. Seine Eigenschaft ist, dass es hohen Temperaturen standhält und ein ideales Enzym ist, aber nach den 1980er Jahren wird es häufig verwendet.Das ursprüngliche Konzept des ursprünglichen primitiven Prototyps der PCR ähnelt der Genreparatur und dem Kopieren, die 1971 von Dr. KJell Kleppe vorgeschlagen wurde. Er veröffentlichte die erste einfache und kurzfristige Genkopie (ähnlich den ersten beiden Zyklusreaktionen der PCR).Die heute entwickelte PCR wurde 1983 von Dr. Kary B. Mullis entwickelt. Dr. Mullis war in diesem Jahr für PE-Unternehmen tätig, daher hat PE in der PCR-Branche einen besonderen Status.Dr. Mullis veröffentlichte 1985 zusammen mit Saiki und anderen offiziell die erste verwandte Arbeit. Seitdem wird die PCR täglich tausende Kilometer lang eingesetzt, und man kann sagen, dass die Qualität der verwandten Arbeiten viele andere Forschungsmethoden ungenießbar macht.Anschließend wird die PCR-Technologie in großem Umfang in der biologisch-wissenschaftlichen Forschung und in klinischen Anwendungen eingesetzt und wird zur wichtigsten Technologie der molekularbiologischen Forschung.Mullis gewann 1993 auch den Nobelpreis für Chemie.
PCRPrinzip
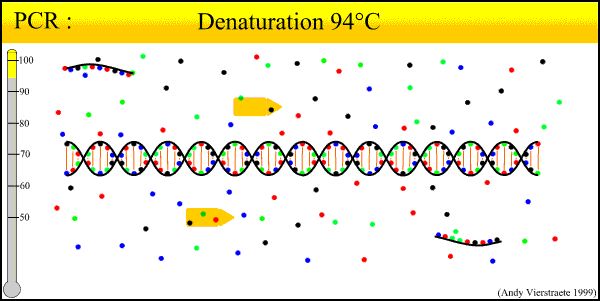
Das Grundprinzip der PCR-Technologie ähnelt dem natürlichen Replikationsprozess der DNA und ihre Spezifität hängt vom Oligonukleotid-Primer ab, der zu beiden Enden der Zielsequenz komplementär ist.Die PCR besteht aus den drei Grundreaktionsschritten Degeneration, Annealing und Verlängerung: ①Degeneration der Matrizen-DNA: Nachdem die Matrizen-DNA für einen bestimmten Zeitraum auf etwa 93 °C erhitzt wurde, wird die duale DNA-Lösung für die durch die PCR-Amplifikation der Matrizen-DNA gebildete Doppelketten-DNA ausgeschieden und zu einer Einzelkette gemacht, sodass sie mit dem Primer kombiniert werden kann, um sich auf die nächste Rundenreaktion vorzubereiten.②Das Annealing (Verbindung) der Matrizen-DNA und des Primers: Nachdem die Matrizen-DNA erhitzt und zu einer einzelnen Kette degeneriert wurde, sinkt die Temperatur auf etwa 55 °C.Die komplementäre Sequenz des Primers und der Template-DNA-Einzelkette.③Die Verlängerung des Primers: DNA-Matrize – die Primerbindung basiert auf der Wirkung der TaqDNA-Polymerase, mit dNTP als Reaktionsrohmaterial.Behalten Sie das Prinzip der Replikation bei, synthetisieren Sie eine neue halbreservierte Kopienkette, die die Template-DNA-Kette ergänzt, und wiederholen Sie den Zyklus Degeneration-Annealing-Extension. Drei Prozesse können mehr „halbreservierte Kopienkette“ erhalten, und diese neue Kette ist wieder verfügbar. Werden Sie eine Vorlage für den nächsten Zyklus.Es dauert 2–4 Minuten, bis die Schleife abgeschlossen ist. Das Zielgen kann in 2–3 Stunden mehrere Millionen Mal amplifiziert werden.
StandardPCRReaktionssystem
| Taq-DNA-Polymerase | 2,5 μl |
| Mg2+ | 1,5 mmol/L |
| 10-facher Amplifikationspuffer | 10μl |
| 4 dNTP-Mischungen | 200μl |
| Template-DNA | 0,1 bis 2 μg |
| Grundierung | 10~100μl |
| Fügen Sie doppelt oder dreifach dampfendes Wasser hinzu | 100 μl |
Fünf Elemente der PCR-Reaktion
An der PCR-Reaktion sind hauptsächlich fünf Arten von Substanzen beteiligt, nämlich Primer, Enzym, dNTP, Template und Puffer (Mg2+ ist erforderlich).[PCR-Verfahren]
Der Standard-PCR-Prozess ist in drei Schritte unterteilt
1. DNA-Degeneration (90 °C–96 °C): Doppelkettige DNA-Matrizen unter thermischer Einwirkung brechen Wasserstoffbrückenbindungen auf und bilden eine einkettige DNA.
2. Annealing (25℃ -65℃): Die Systemtemperatur wird gesenkt, der Primer wird mit der DNA-Matrize kombiniert, um eine lokale Doppelkette zu bilden.
3. Verlängerung (70℃ -75℃): Unter der Wirkung des Taq-Enzyms (ca. 72°C, die beste Aktivität) wird dNTP als Rohmaterial verwendet, das sich vom 5′-Ende des Primers → 3′-Ende erstreckt, Synthese und Matrize ergänzen sich gegenseitig DNA-Kette.
Jeder Zyklus wird denaturiert, angelagert und verlängert, wodurch sich der DNA-Gehalt verdoppelt.Derzeit kann aufgrund der kurzen Amplifikationsfläche ein Teil der PCR in sehr kurzer Zeit repliziert werden, selbst wenn die Taq-Enzymaktivität nicht optimal ist. Daher kann auf zwei Schritte umgestellt werden, d. h. das Annealing und die Verlängerung können gleichzeitig bei 60 °C bis 65 °C durchgeführt werden.Um den Prozess des Hebens und Abkühlens zu reduzieren und die Reaktionsgeschwindigkeit zu verbessern.
Funktionen der PCR-Reaktion
● Hohe Spezifität
Die spezifischen entscheidenden Faktoren der PCR-Antwort sind: ①Die spezifische Kombination des Primers und der Template-DNA.②Das Prinzip der Basenpaarung.③Die Loyalität der TaqDNA-Polymerase-Synthesereaktion.④Die Spezifität und Konservativität des Zielgens.
Die richtige Kombination von Primern und Templates ist der Schlüssel.Die Bindung des Primers und des Templats sowie die Verlängerung der Primerkette basieren auf dem Prinzip der alkalischen Basenpaarung.Die Loyalität der Polymerase-Synthesereaktionen und die hohe Temperaturbeständigkeit der Taq-DNA-Polymerase sorgen dafür, dass die Bindung (Verbindung) der Matrize und des Primers in der Reaktion bei einer höheren Temperatur durchgeführt werden kann.Die Spezifität der Kombination wird deutlich erhöht.Der Clip kann ein hohes Maß an Korrektheit beibehalten.Durch die Auswahl einer genetischen Zielregion mit hoher Konservativität und hoher Konservativität ist ihre Spezifität höher.
● Hohe Empfindlichkeit
Das Produktionsvolumen von PCR-Produkten wird durch einen Index erhöht, der die Startvorlage von Picker (PG=10-12) erweitern kann, um die Mikrocontroller-Ebene auf die Ebene von Mikrogrammen (μg= -6) zu erhöhen.Eine Zielzelle kann aus 1 Million Zellen nachgewiesen werden;beim Nachweis von Viren kann die Empfindlichkeit der PCR 3 RFUs (empty Spots Formed Units) erreichen;Die Mindesterkennungsrate in der Bakterienwissenschaft beträgt 3 Bakterien.
● Einfach und schnell
Die PCR-Reflexion verwendet eine Hochtemperatur-Taq-DNA-Polymerase, die die Reaktionslösung auf einmal hinzufügt, d. h. eine Degenerations-Anneal-Extension-Reaktion an der DNA-Amplifikationslösung und dem Wasserbadtopf.Im Allgemeinen ist die Amplifikationsreaktion in 2 bis 4 Stunden abgeschlossen.Erweiterte Produkte werden im Allgemeinen mit einem elektrischen Schwert analysiert und erfordern keine Isotope, keine radioaktive Verschmutzung und eine einfache Förderung.
● Die Reinheit der Probe ist gering
Eine Trennung von Viren oder Bakterien und Kulturzellen ist nicht erforderlich.Als Verstärker können DNA-Rohprodukte und RNA eingesetzt werden.Der DNA-Amplifikationsnachweis kann direkt an klinischen Proben wie Blut, Körperflüssigkeit, Hustenspülflüssigkeit, Haaren, Zellen und lebendem Gewebe durchgeführt werden.
PCRallgemeine Probleme
● Falsch negativ, keine verstärkten Banden
Zu den wichtigsten Phasen der PCR-Reaktion gehören: ① Vorbereitung der Template-Nukleinsäuren, ② Qualität und Spezifität der Primer, ③ die Qualität der Enzyme ④ PCR-Zyklusbedingungen.Die Ursachenfindung sollte auch für die oben genannten Links analysiert und untersucht werden.
Vorlagen: ① Die Vorlage enthält verschiedene Proteine, ② Die Vorlage enthält einen Taq-Enzym-Inhibitor, ③ Das Protein in der Vorlage wird nicht eliminiert, insbesondere das Gruppenprotein im Chromosom.⑤ Die Degeneration der Deminer-Nukleinsäure ist nicht gründlich.Wenn die Qualität der Enzyme und Primer gut ist, gibt es keine Amplifikationsbande, was höchstwahrscheinlich auf die Verdauungsbehandlung der Proben zurückzuführen ist.Beim Extraktionsprozess der Template-Nukleinsäure stimmt etwas nicht. Um eine wirksame und stabile Verdauungslösung herzustellen, sollte das Verfahren festgelegt und nicht willkürlich geändert werden.
Enzyminaktivierung: Ein neues Enzym oder sowohl alte als auch neue Enzyme sollten zusammen verwendet werden, um zu analysieren, ob die Enzymaktivität verloren geht oder unzureichend ist, was zu falsch negativen Ergebnissen führt.Es ist zu beachten, dass Taq-Enzym oder Ethidiumbromid manchmal vergessen werden.
Primer: Die Qualität des Primers, die Konzentration des Primers und ob die Konzentration der beiden Primer symmetrisch ist.Dies ist ein häufiger Grund für das Scheitern der PCR oder die zunehmende Bande ist nicht ideal und neigt zur Diffusion.Bei einigen Chargennummern gibt es Probleme mit der Qualität der Primer.Die beiden Primer haben eine hohe und eine niedrige Konzentration, was zu einer asymmetrischen Amplifikation mit geringer Effizienz führt.Die Gegenmaßnahmen sind: ① Wählen Sie einen guten Primer, um Einheiten zu synthetisieren.② Die Konzentration des Primers hängt nicht nur vom OD-Wert ab, sondern berücksichtigt auch die ursprüngliche Flüssigkeit des Primers, um eine Agar-Zucker-Gelelektrophorese durchzuführen.Es muss eine Primerstreifenzone vorhanden sein und die Helligkeit der beiden Primer sollte im Allgemeinen konsistent sein.Gürtel, PCR kann zu diesem Zeitpunkt fehlschlagen und sollte mit der Primer-Syntheseeinheit behoben werden.Wenn eine Grundierung hoch ist, ist die Helligkeit niedrig und ihre Konzentration muss beim Verdünnen ausgeglichen werden.③ Die Grundierung sollte in einer hohen Konzentration aufgetragen und gelagert werden, um zu verhindern, dass Teile des Kühlschranks mehrfach einfrieren oder über einen längeren Zeitraum gekühlt werden, was zu einer Verschlechterung und einem Abbau der Grundierung führt.④ Das Design des Primers ist unangemessen, da die Länge des Primers nicht ausreicht und sich zwischen den Primern ein Di-Cluster bildet.
Mg2+-Konzentration: Die Mg2+-Ionenkonzentration hat einen großen Einfluss auf die PCR-Amplifikationseffizienz.Eine zu hohe Konzentration kann die PCR-Amplifikation des anderen Geschlechts beeinträchtigen.Wenn die Konzentration zu niedrig ist, führt die PCR-Amplifikationsleistung sogar dazu, dass die PCR-Amplifikation ohne das Expansionsband fehlschlägt.
Änderung des Reaktionsvolumens: Das bei der PCR-Amplifikation verwendete Volumen beträgt 20 µl, 30 µl und 50 µl oder 100 µl. Das große Volumen der Anwendung für die PCR-Amplifikation wird entsprechend den unterschiedlichen Zwecken der wissenschaftlichen Forschung und klinischen Tests festgelegt.Nach der Herstellung kleiner Volumina, wie z. B. 20 µl, ist es notwendig, beim Erstellen der Größe eine Kordelbedingung zu berücksichtigen, da sonst ein Fehler auftritt.
Physikalische Gründe: Die Transformation ist für die PCR-Amplifikation sehr wichtig.Wenn die Degenerationstemperatur niedrig ist, ist die Degenerationszeit kurz und es kommt wahrscheinlich zu falsch negativen Ergebnissen.Eine zu niedrige Annealing-Temperatur kann zu einer unspezifischen Amplifikation führen und die Effizienz der spezifischen Amplifikation verringern.Beeinträchtigt stark die Kombination von Primern und Templates, um die Effizienz der PCR-Amplifikation zu verringern.Manchmal ist es notwendig, Standardthermometer zu verwenden, um die Variabilität, das Annealing und die verlängerte Temperatur im Verlängerungs- oder wasserlöslichen Kocher zu erfassen, was einer der Gründe für das Scheitern der PCR ist.
Zielsequenzvarianten: Wenn die Zielsequenz auftritt, eine Mutation oder Deletion vorliegt, die Kombination aus Prototyp und Matrize kombiniert wird oder aufgrund des Fehlens einer Zielsequenz der Primer und die Matrize die komplementäre Sequenz verlieren und ihre PCR-Amplifikation nicht erfolgreich sein wird.
● Falsch positiv
Die PCR-Amplifikationsbande scheint mit der Zielsequenzbande übereinzustimmen, und manchmal ist ihre Bande deutlicher und höher.
Das Primerdesign ist nicht geeignet: Die ausgewählte Amplifikationssequenz und die nicht zweckgebundene Amplifikationssequenz sind homolog, sodass bei der PCR-Amplifikation die amplifizierten PCR-Produkte nicht zweckgebundene Sequenzen sind.Die Zielsequenz ist zu kurz oder der Primer ist zu kurz und es besteht die Gefahr falsch positiver Ergebnisse.Muss neu gestaltet werden.
Kreuzkontamination von Zielsequenzen oder Amplifikationsprodukten: Für diese Kontamination gibt es zwei Gründe: Erstens Kreuzkontamination des gesamten Genoms oder großer Segmente, was zu falsch positiven Ergebnissen führt.Diese Art falsch positiver Ergebnisse kann durch die folgenden Methoden behoben werden: Gehen Sie während des Betriebs vorsichtig und vorsichtig vor, um zu verhindern, dass die Zielsequenz in die Probenpistole eingeatmet wird oder aus dem Zentrifugenröhrchen spritzt.Mit Ausnahme von Enzymen und Substanzen, die hohen Temperaturen nicht standhalten, sollten alle Reagenzien oder Geräte mit hohem Druck desinfiziert werden.Die Zentrifugenrohre und Proben sollten gleichzeitig verwendet werden.Bei Bedarf werden das Reaktionsgefäß und das Reagenz vor dem Hinzufügen von Proben ultravioletten Strahlen ausgesetzt, um die vorhandene Nukleinsäure zu zerstören.Zweitens kleine Fragmente in der Luftverschmutzung.Diese kleinen Fragmente sind kürzer als die Zielsequenz, weisen aber eine gewisse Homologie auf.Es kann miteinander gespleißt werden.Nach der Ergänzung der Primer kann das PCR-Produkt expandiert werden, was zu einer falsch positiven Produktion führt.Es kann verwendet werden, um die Nest-PCR-Methode zu reduzieren oder zu eliminieren.
● Es erscheint eine unspezifische Amplifikationsbande
Die nach der PCR-Amplifikation aufgetretenen Banden stimmen nicht mit der erwarteten Größe überein oder sind groß oder klein oder gleichzeitig, oder es gibt gleichzeitig spezifische Amplifikationsbanden und unspezifische Amplifikationsbanden.Die Entstehung unspezifischer Banden ist folgende: Erstens sind die Primer unvollständig komplementär zur Zielsequenz oder die Polymerisation des Primers führt zu einem Di-Cluster.Der zweite Grund ist, dass die Konzentration an MG2+-Ionen zu hoch ist, die Annealing-Temperatur zu niedrig ist und die Anzahl der PCR-Zyklen damit zusammenhängt.Zweitens die Qualität und Menge der Enzyme.Oft sind Enzyme einiger Quellen anfällig für nicht-spezielle Banden und die Enzyme der anderen Quelle treten nicht auf.Manchmal kommt es auch zu einer unspezifischen Verstärkung von Enzymen.Die Gegenmaßnahmen sind: ggf. umgestaltete Attraktivität.Reduzieren Sie die Enzymmenge oder ersetzen Sie das Enzym durch eine andere Quelle.Reduzieren Sie die Anzahl der Primärdaten, erhöhen Sie entsprechend die Anzahl der Vorlagen und verringern Sie die Anzahl der Zyklen.Erhöhen Sie die Glühtemperatur ordnungsgemäß oder verwenden Sie die Zwei-Temperatur-Punkt-Methode (93 °C-Degeneration, Glühen und Strecken bei etwa 65 °C).
● Es erscheinen flockige Werg- oder Schmierklebebänder
Die PCR-Amplifikation scheint manchmal wie ein angewandter oder schalenförmiger oder teppichartiger Gürtel zu sein.Aus diesem Grund ist die dNTP-Konzentration aufgrund der übermäßigen Menge an Enzymen oder der schlechten Qualität des Enzyms zu hoch, die Mg2+-Konzentration ist zu hoch, die Annealing-Temperatur ist zu niedrig und die Anzahl der Zyklen ist zu hoch.Die Gegenmaßnahmen sind: ①Reduzieren Sie die Menge an Enzymen oder ersetzen Sie das Enzym einer anderen Quelle.②Reduzieren Sie die dNTP-Konzentration. ③Reduzieren Sie die Mg2+-Konzentration ordnungsgemäß.④Erhöhen Sie die Anzahl der Vorlagen und reduzieren Sie die Anzahl der Zyklen.
Verwandte Produkte
◮ Höhere Wiedergabetreue: 6-mal höher als bei gewöhnlichem Taq-Enzym;
◮ Schnellere Verstärkungsgeschwindigkeit
◮ Mehr Anpassungsfähigkeit der Vorlagen
◮ Höhere Verstärkungseffizienz
◮ Die Umwelttoleranz ist stärker: Eine Woche lang bei 37 °C gelagert, wodurch eine Aktivität von mehr als 90 % erhalten bleibt.
◮ Es verfügt über 5'→3'-DNA-Polymerase-Aktivität und 5'→3'-Exonuklease-Aktivität, ohne 3'→5'-Exonuklease-Aktivität.
Das einzigartige Reaktionssystem und die hocheffiziente Taq-DNA-Polymerase sorgen dafür, dass die PCR-Reaktion eine höhere Amplifikationseffizienz, Spezifität und Empfindlichkeit aufweist.
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
◮ Mit dem One-Step-Kit können Reverse Transkription und qPCR in zwei Reaktionen im selben Röhrchen durchgeführt werden. Es müssen lediglich Template-RNA, spezifische PCR-Primer und RNase-freies ddH hinzugefügt werden2O.
◮ Das Kit kann virale RNA schnell und effizient quantitativ analysieren oder RNA nachverfolgen.
◮ Das Kit verwendet ein einzigartiges Foregene-Reverse-Transkriptionsreagenz und Foregene HotStar Taq DNA-Polymerase in Kombination mit einem einzigartigen Reaktionssystem, um die Amplifikationseffizienz und Spezifität der Reaktion effektiv zu verbessern.
◮ Das optimierte Reaktionssystem sorgt dafür, dass die Reaktion eine höhere Nachweisempfindlichkeit, eine stärkere thermische Stabilität und eine bessere Toleranz aufweist.
◮ RT-qPCR einfachTMDas (One Step)-SYBR Green I-Kit wird mit einem internen ROX-Referenzfarbstoff geliefert, der zur Eliminierung von Signalhintergrund und Signalfehlern zwischen Wells verwendet werden kann, was für Kunden bequem in verschiedenen Modellen quantitativer PCR-Instrumente zu verwenden ist.
RT EinfachTMII (Master-Premix für Erststrang-cDNA-Synthese fürEchtzeit-PCR)
-Effiziente Fähigkeit zur Entfernung von gDNA, wodurch gDNA in der Vorlage innerhalb von 2 Minuten entfernt werden kann.
-Effizientes Reverse-Transkriptionssystem, es dauert nur 15 Minuten, um die Synthese des ersten cDNA-Strangs abzuschließen.
-Komplexe Vorlagen: Vorlagen mit hohem GC-Gehalt und komplexer Sekundärstruktur können auch mit hoher Effizienz umgekehrt werden.
-Hochempfindliches Reverse-Transkriptionssystem, Vorlagen auf pg-Ebene können auch hochwertige cDNA erhalten.
-Das Reverse-Transkriptionssystem weist eine hohe thermische Stabilität auf, die optimale Reaktionstemperatur liegt bei 42℃ und es weist auch bei 50℃ immer noch eine gute Reverse-Transkriptionsleistung auf.
Zeitpunkt der Veröffentlichung: 18. März 2023