COVID-19 ist eine Infektionskrankheit, die durch das schwere akute respiratorische Syndrom Coronavirus Typ 2 verursacht wird. Wenn eine Person infiziert ist, sind die häufigsten Symptome Fieber, Husten und Kurzatmigkeit.
 Die für den Test verwendeten Proben können durch Nasopharyngealabstriche oder Oropharyngealabstriche entnommen werden.
Die für den Test verwendeten Proben können durch Nasopharyngealabstriche oder Oropharyngealabstriche entnommen werden.
Die Standardmethode zum Nachweis von Coronaviren ist die Polymerase-Kettenreaktion, PCR.Dies ist eine in der Molekularbiologie weit verbreitete Methode.Es kann schnell Millionen bis Milliarden spezifischer DNA-Fragmente kopieren.
 Das neue Coronavirus enthält ein sehr langes einzelsträngiges RNA-Genom.Um diese Viren mittels PCR nachzuweisen, müssen RNA-Moleküle durch Reverse Transkriptase in ihre komplementären DNA-Sequenzen umgewandelt werden. Anschließend kann die neu synthetisierte DNA durch Standard-PCR-Verfahren, die allgemein als RT-PCR bekannt sind, amplifiziert werden.
Das neue Coronavirus enthält ein sehr langes einzelsträngiges RNA-Genom.Um diese Viren mittels PCR nachzuweisen, müssen RNA-Moleküle durch Reverse Transkriptase in ihre komplementären DNA-Sequenzen umgewandelt werden. Anschließend kann die neu synthetisierte DNA durch Standard-PCR-Verfahren, die allgemein als RT-PCR bekannt sind, amplifiziert werden.
RT-PCR-Prozess
RNA-Extraktion
Zur Durchführung dieser Methode sollte grundsätzlich virale RNA extrahiert werden.Für eine bequeme, schnelle und effektive Trennung können verschiedene RNA-Reinigungskits verwendet werden.
Um virale RNA mit einem kommerziellen Kit zu extrahieren, geben Sie die Probe zunächst in ein Mikrozentrifugenröhrchen und mischen Sie sie dann mit dem Lysepuffer.Dieser Puffer ist stark denaturiert und besteht üblicherweise aus Phenol und Guanidinisothiocyanat.Darüber hinaus sind im Lysepuffer üblicherweise RNase-Inhibitoren vorhanden, um die Isolierung intakter viraler RNA sicherzustellen.
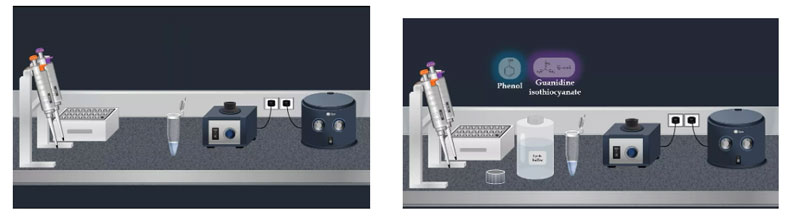 Nach Zugabe des Lysepuffers das Mischrohr pulsieren und bei Raumtemperatur inkubieren.Anschließend wird das Virus unter stark denaturierenden Bedingungen des Lysepuffers lysiert.
Nach Zugabe des Lysepuffers das Mischrohr pulsieren und bei Raumtemperatur inkubieren.Anschließend wird das Virus unter stark denaturierenden Bedingungen des Lysepuffers lysiert.
 Nachdem die Probe lysiert ist, wird ein Zentrifugenröhrchen für den Reinigungsvorgang verwendet.Die Probe wird in das Zentrifugenröhrchen geladen und anschließend zentrifugiert.
Nachdem die Probe lysiert ist, wird ein Zentrifugenröhrchen für den Reinigungsvorgang verwendet.Die Probe wird in das Zentrifugenröhrchen geladen und anschließend zentrifugiert.
 Bei diesem Verfahren handelt es sich um eine Festphasenextraktionsmethode, bei der die stationäre Phase aus einer Kieselgelmatrix besteht.
Bei diesem Verfahren handelt es sich um eine Festphasenextraktionsmethode, bei der die stationäre Phase aus einer Kieselgelmatrix besteht.
 Unter optimalen Salz- und pH-Bedingungen binden RNA-Moleküle an die Silica-Membran.
Unter optimalen Salz- und pH-Bedingungen binden RNA-Moleküle an die Silica-Membran.
 Gleichzeitig werden Eiweiß und andere Verunreinigungen entfernt.
Gleichzeitig werden Eiweiß und andere Verunreinigungen entfernt.
 Nach der Zentrifugation das Zentrifugenröhrchen in ein sauberes Sammelröhrchen geben, das Filtrat verwerfen und dann Waschpuffer hinzufügen.
Nach der Zentrifugation das Zentrifugenröhrchen in ein sauberes Sammelröhrchen geben, das Filtrat verwerfen und dann Waschpuffer hinzufügen.
 Stellen Sie das Röhrchen erneut in die Zentrifuge, um den Waschpuffer durch die Membran zu drücken.Dadurch werden alle verbleibenden Verunreinigungen von der Membran entfernt und nur die RNA bleibt an das Kieselgel gebunden.
Stellen Sie das Röhrchen erneut in die Zentrifuge, um den Waschpuffer durch die Membran zu drücken.Dadurch werden alle verbleibenden Verunreinigungen von der Membran entfernt und nur die RNA bleibt an das Kieselgel gebunden.
 Nachdem die Probe gewaschen wurde, geben Sie das Röhrchen in ein sauberes Mikrozentrifugenröhrchen und geben Sie den Elutionspuffer hinzu.
Nachdem die Probe gewaschen wurde, geben Sie das Röhrchen in ein sauberes Mikrozentrifugenröhrchen und geben Sie den Elutionspuffer hinzu.
 Anschließend wird es zentrifugiert, um den Elutionspuffer durch die Membran zu drücken.Der Elutionspuffer entfernt virale RNA von der Spin-Säule und erhält gereinigte RNA, die frei von Proteinen, Inhibitoren und anderen Verunreinigungen ist.
Anschließend wird es zentrifugiert, um den Elutionspuffer durch die Membran zu drücken.Der Elutionspuffer entfernt virale RNA von der Spin-Säule und erhält gereinigte RNA, die frei von Proteinen, Inhibitoren und anderen Verunreinigungen ist.
Gemischtes Konzentrat
Nach der Extraktion der viralen RNA besteht der nächste Schritt darin, die Reaktionsmischung für die PCR-Amplifikation vorzubereiten.In diesem Schritt wird Konzentrat verwendet.Bei dieser konzentrierten Lösung handelt es sich um eine vorgemischte konzentrierte Lösung, die aus einer Vormischung, Reverse Transkriptase, Nukleotiden, Vorwärtsprimer, Rückwärtsprimer, TaqMan-Sonde und DNA-Polymerase besteht.
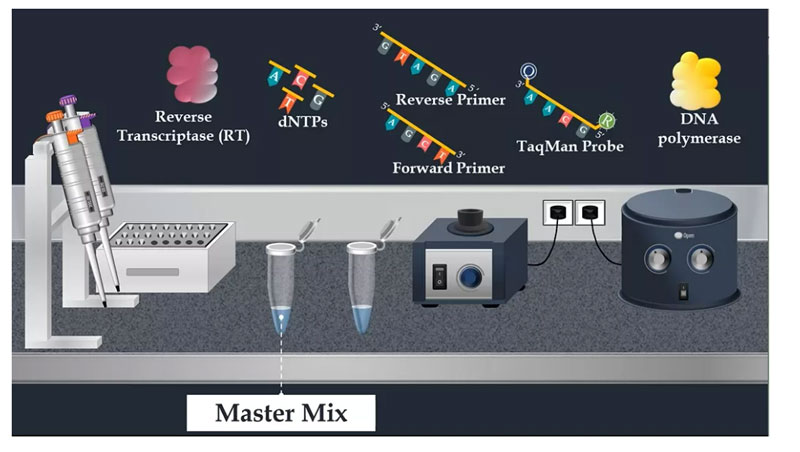 Um diese Reaktionsmischung zu vervollständigen, wird schließlich die RNA-Matrize hinzugefügt.Die Röhrchen werden durch Pulsvortexen gemischt und dann wird die Reaktionsmischung in die PCR-Platte geladen.Die PCR-Platte enthält in der Regel 96 Wells und kann mehrere Proben gleichzeitig analysieren.
Um diese Reaktionsmischung zu vervollständigen, wird schließlich die RNA-Matrize hinzugefügt.Die Röhrchen werden durch Pulsvortexen gemischt und dann wird die Reaktionsmischung in die PCR-Platte geladen.Die PCR-Platte enthält in der Regel 96 Wells und kann mehrere Proben gleichzeitig analysieren.
PCR-Amplifikation
Als nächstes legen Sie die Platte in das PCR-Gerät, das im Wesentlichen ein Thermocycler ist.
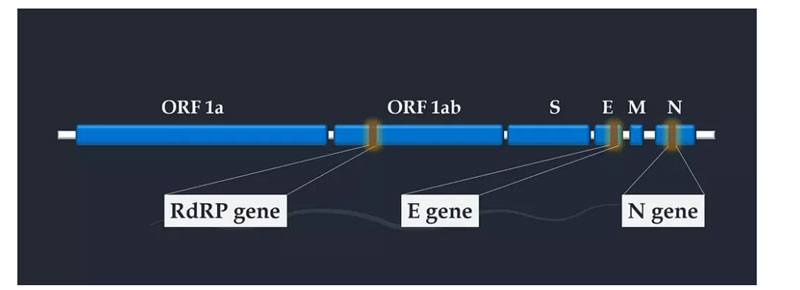
 Echtzeit-RT-PCR dient zum Nachweis des 2019 neuartigen Coronavirus durch Amplifikation der Zielsequenz im RdrRP-Gen, E-Gen und N-Gen.Die Wahl des Zielgens hängt von der Primer- und Sondensequenz ab.
Echtzeit-RT-PCR dient zum Nachweis des 2019 neuartigen Coronavirus durch Amplifikation der Zielsequenz im RdrRP-Gen, E-Gen und N-Gen.Die Wahl des Zielgens hängt von der Primer- und Sondensequenz ab.
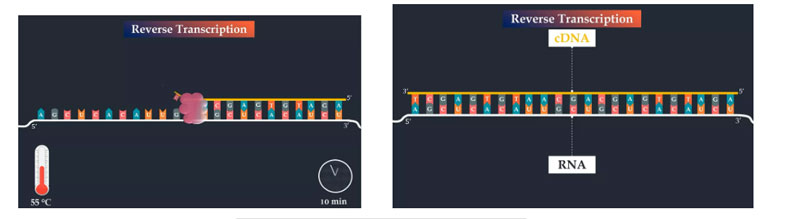
 Der erste Schritt der RT-PCR ist die reverse Transkription.Der erste Strang komplementärer DNA wird synthetisiert, was durch den PCR-Reverse-Primer initiiert wird, der an den komplementären Teil des viralen RNA-Genoms bindet.Dann fügt die Reverse Transkriptase DNA-Nukleotide an das 3′-Ende des Primers hinzu, um DNA zu synthetisieren, die zur viralen RNA komplementär ist.Die Temperatur und Dauer dieses Schritts hängen von den verwendeten Primern, der Ziel-RNA und der Reversen Transkriptase ab.
Der erste Schritt der RT-PCR ist die reverse Transkription.Der erste Strang komplementärer DNA wird synthetisiert, was durch den PCR-Reverse-Primer initiiert wird, der an den komplementären Teil des viralen RNA-Genoms bindet.Dann fügt die Reverse Transkriptase DNA-Nukleotide an das 3′-Ende des Primers hinzu, um DNA zu synthetisieren, die zur viralen RNA komplementär ist.Die Temperatur und Dauer dieses Schritts hängen von den verwendeten Primern, der Ziel-RNA und der Reversen Transkriptase ab.
 Als nächstes wird ein erster Denaturierungsschritt angewendet, der zur Denaturierung des RNA-DNA-Hybrids führt.Dieser Schritt ist notwendig, um die DNA-Polymerase zu aktivieren.Gleichzeitig wird die Reverse Transkriptase inaktiviert.
Als nächstes wird ein erster Denaturierungsschritt angewendet, der zur Denaturierung des RNA-DNA-Hybrids führt.Dieser Schritt ist notwendig, um die DNA-Polymerase zu aktivieren.Gleichzeitig wird die Reverse Transkriptase inaktiviert.
 Die PCR besteht aus einer Reihe thermischer Zyklen.Jeder Zyklus besteht aus Denaturierungs-, Annealing- und Verlängerungsschritten.
Die PCR besteht aus einer Reihe thermischer Zyklen.Jeder Zyklus besteht aus Denaturierungs-, Annealing- und Verlängerungsschritten.
 Beim Denaturierungsschritt wird die Reaktionskammer auf 95 Grad Celsius erhitzt und zur Denaturierung der doppelsträngigen DNA-Matrize verwendet.
Beim Denaturierungsschritt wird die Reaktionskammer auf 95 Grad Celsius erhitzt und zur Denaturierung der doppelsträngigen DNA-Matrize verwendet.
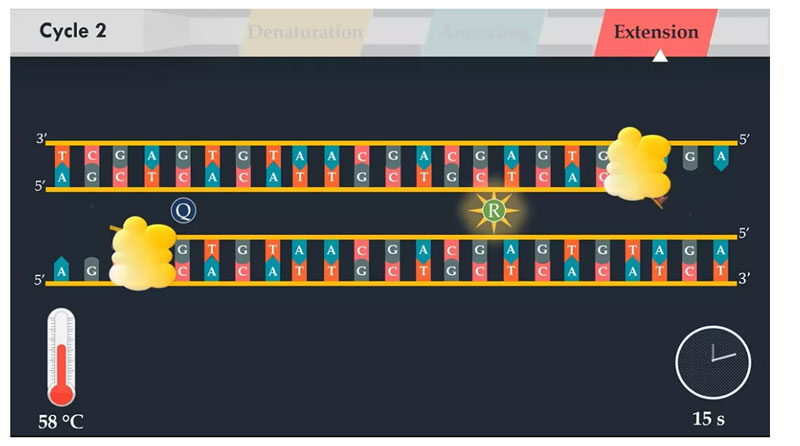
 Im nächsten Schritt wird die Reaktionstemperatur auf 58 Grad Celsius gesenkt, sodass sich der Vorwärtsprimer an den komplementären Teil seiner einzelsträngigen DNA-Matrize anlagern kann.Die Glühtemperatur hängt direkt von der Länge und Zusammensetzung des Primers ab.
Im nächsten Schritt wird die Reaktionstemperatur auf 58 Grad Celsius gesenkt, sodass sich der Vorwärtsprimer an den komplementären Teil seiner einzelsträngigen DNA-Matrize anlagern kann.Die Glühtemperatur hängt direkt von der Länge und Zusammensetzung des Primers ab.
 Im Verlängerungsschritt synthetisiert die DNA-Polymerase einen neuen DNA-Strang, der zum DNA-Matrizenstrang komplementär ist.Durch Zugabe freier, zum Templat komplementärer Kerne in 5′- bis 3′-Richtung aus dem Reaktionsgemisch.Die Temperatur dieses Schritts hängt von der verwendeten DNA-Polymerase ab.
Im Verlängerungsschritt synthetisiert die DNA-Polymerase einen neuen DNA-Strang, der zum DNA-Matrizenstrang komplementär ist.Durch Zugabe freier, zum Templat komplementärer Kerne in 5′- bis 3′-Richtung aus dem Reaktionsgemisch.Die Temperatur dieses Schritts hängt von der verwendeten DNA-Polymerase ab.
 Nach dem ersten Zyklus wird ein doppelsträngiges DNA-Ziel erhalten.
Nach dem ersten Zyklus wird ein doppelsträngiges DNA-Ziel erhalten.
 Dann treten Sie in den zweiten Zyklus ein.Die doppelsträngige DNA wird denaturiert, um zwei einzelsträngige DNA-Moleküle zu erzeugen.
Dann treten Sie in den zweiten Zyklus ein.Die doppelsträngige DNA wird denaturiert, um zwei einzelsträngige DNA-Moleküle zu erzeugen.
 Im nächsten Schritt wird die Reaktionstemperatur gesenkt, die Primer an jede einzelsträngige DNA-Matrize angelagert und die Taq-man-Sonde an den komplementären Teil der Ziel-DNA angelagert.
Im nächsten Schritt wird die Reaktionstemperatur gesenkt, die Primer an jede einzelsträngige DNA-Matrize angelagert und die Taq-man-Sonde an den komplementären Teil der Ziel-DNA angelagert.
 Die TaqMan-Sonde besteht aus einem Fluorophor, der kovalent an das 5'-Ende der Oligonukleotidsonde gebunden ist.Bei Anregung durch die Lichtquelle des Cyclers emittiert der Fluorophor Fluoreszenz.Darüber hinaus besteht die Sonde aus einem Quencher am 3′-Ende.Die Nähe des Reportergens zum Quencher verhindert den Nachweis von Fluoreszenz.
Die TaqMan-Sonde besteht aus einem Fluorophor, der kovalent an das 5'-Ende der Oligonukleotidsonde gebunden ist.Bei Anregung durch die Lichtquelle des Cyclers emittiert der Fluorophor Fluoreszenz.Darüber hinaus besteht die Sonde aus einem Quencher am 3′-Ende.Die Nähe des Reportergens zum Quencher verhindert den Nachweis von Fluoreszenz.
 Im Verlängerungsschritt synthetisiert die DNA-Polymerase einen neuen Strang.Wenn die Polymerase die TaqMan-Sonde erreicht, spaltet ihre endogene 5′-Nukleaseaktivität die Sonde und trennt so den Farbstoff vom Quencher.
Im Verlängerungsschritt synthetisiert die DNA-Polymerase einen neuen Strang.Wenn die Polymerase die TaqMan-Sonde erreicht, spaltet ihre endogene 5′-Nukleaseaktivität die Sonde und trennt so den Farbstoff vom Quencher.
 Mit jedem PCR-Zyklus werden mehr Farbstoffmoleküle freigesetzt, was zu einem Anstieg der Fluoreszenzintensität proportional zur Anzahl der synthetisierten Amplifikate führt.
Mit jedem PCR-Zyklus werden mehr Farbstoffmoleküle freigesetzt, was zu einem Anstieg der Fluoreszenzintensität proportional zur Anzahl der synthetisierten Amplifikate führt.
 Diese Methode ermöglicht die Schätzung der Anzahl einer bestimmten Sequenz, die in der Probe vorhanden ist.Die Anzahl der doppelsträngigen DNA-Fragmente verdoppelt sich in jedem Zyklus.Daher kann die PCR zur Analyse sehr kleiner Proben eingesetzt werden.
Diese Methode ermöglicht die Schätzung der Anzahl einer bestimmten Sequenz, die in der Probe vorhanden ist.Die Anzahl der doppelsträngigen DNA-Fragmente verdoppelt sich in jedem Zyklus.Daher kann die PCR zur Analyse sehr kleiner Proben eingesetzt werden.
 Zur Messung von Fluoreszenzsignalen, Wolfram-Halogenlampen, Anregungsfiltern, Reflektoren, Linsen, Emissionsfiltern und CCD-Kameras mit ladungsgekoppelten Geräten.
Zur Messung von Fluoreszenzsignalen, Wolfram-Halogenlampen, Anregungsfiltern, Reflektoren, Linsen, Emissionsfiltern und CCD-Kameras mit ladungsgekoppelten Geräten.
SCHRITT 4 Erkennen
Zur Messung von Fluoreszenzsignalen, Wolfram-Halogenlampen, Anregungsfiltern, Reflektoren, Linsen, Emissionsfiltern und CCD-Kameras mit ladungsgekoppelten Geräten.
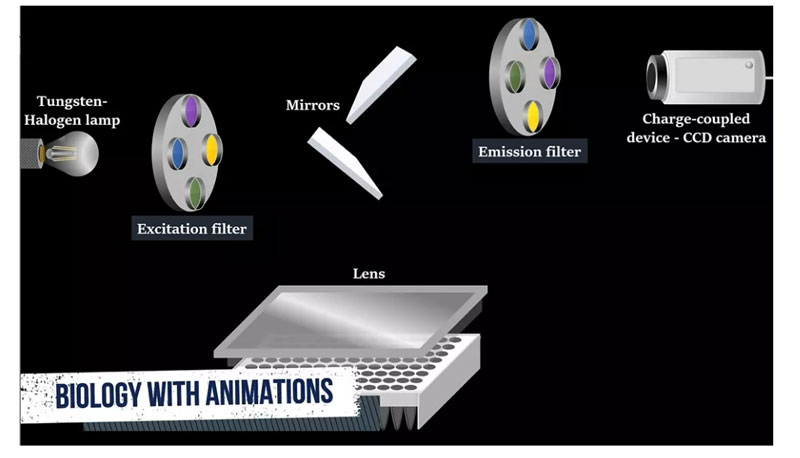 Das gefilterte Licht der Lampe wird vom Reflektor reflektiert, durchläuft die Kondensorlinse und wird auf die Mitte jedes Lochs fokussiert.Anschließend wird die vom Loch emittierte Fluoreszenz vom Spiegel reflektiert, durchläuft den Emissionsfilter und wird von der CCD-Kamera erfasst.In jedem PCR-Zyklus kann das selbsterregte Fluorophorlicht vom CCD erfasst werden.
Das gefilterte Licht der Lampe wird vom Reflektor reflektiert, durchläuft die Kondensorlinse und wird auf die Mitte jedes Lochs fokussiert.Anschließend wird die vom Loch emittierte Fluoreszenz vom Spiegel reflektiert, durchläuft den Emissionsfilter und wird von der CCD-Kamera erfasst.In jedem PCR-Zyklus kann das selbsterregte Fluorophorlicht vom CCD erfasst werden.
 Es wandelt das erfasste Licht in digitale Daten um.Diese Methode wird als Echtzeit-PCR bezeichnet und ermöglicht die Echtzeitüberwachung des Fortschritts der PCR-Reaktion.
Es wandelt das erfasste Licht in digitale Daten um.Diese Methode wird als Echtzeit-PCR bezeichnet und ermöglicht die Echtzeitüberwachung des Fortschritts der PCR-Reaktion.
Zeitpunkt der Veröffentlichung: 19. Juli 2021













