Überblick
Schnelle Identifizierung transgener Pflanzen
Text/Tong Yucheng
Experimenteller Betrieb/Han Ying
Herausgeber/Wen Youjun
Wörter/1600+
Empfohlene Lesezeit: 8–10 Minuten
Schnelle Identifizierung transgener Pflanzen
Als Neuling im Labor ist es keine gute Aufgabe, positive Pflanzen aus einer Reihe von Pflanzen mit einer niedrigen Umwandlungsrate herauszufiltern.Zunächst muss aus einer großen Anzahl von Proben einzeln DNA extrahiert werden, anschließend werden die fremden Gene per PCR nachgewiesen.Bei den Ergebnissen handelt es sich jedoch häufig um Leerzeichen und Streifen mit gelegentlich ein paar Elementen, es ist jedoch unmöglich festzustellen, ob es sich um Fehlerkennungen oder Fehlerkennungen handelt..Ist es sehr hilflos, solchen experimentellen Prozessen und Ergebnissen gegenüberzutreten?Machen Sie sich keine Sorgen, Bruder bringt Ihnen bei, wie Sie transgene positive Pflanzen einfach und genau aussortieren.
Schritt 1: Designerkennungsprimer
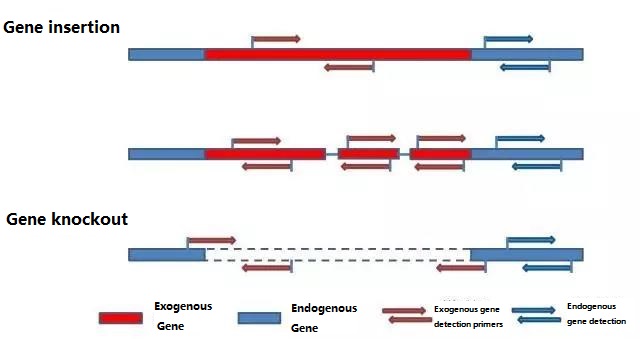
Bestimmen Sie das nachzuweisende endogene und exogene Gen entsprechend der zu testenden Probe und wählen Sie eine repräsentative 100-500-bp-Sequenz im Gen für das Primer-Design aus.Gute Primer können die Genauigkeit der Nachweisergebnisse sicherstellen und die Nachweiszeit verkürzen (siehe Anhang für häufig verwendete Nachweisprimer).
Notiz:
Die neu entwickelten Primer müssen die Reaktionsbedingungen optimieren und die Genauigkeit, Präzision und Nachweisgrenze des Nachweises überprüfen, bevor ein groß angelegter Nachweis durchgeführt wird.
Schritt 2:Entwickeln Sie ein Versuchsprotokoll

Positivkontrolle: Verwenden Sie die gereinigte DNA, die das Zielfragment enthält, als Vorlage, um zu bestimmen, ob das PCR-Reaktionssystem und die Bedingungen normal sind.
Negativ-/Leerkontrolle: Verwenden Sie eine DNA-Vorlage oder ddH2O, das das Zielfragment nicht als Vorlage enthält, um festzustellen, ob eine Kontaminationsquelle im PCR-System vorhanden ist.
Interne Referenzkontrolle: Verwenden Sie die Primer/Sonden-Kombination des endogenen Gens der zu testenden Probe, um zu bewerten, ob die Vorlage durch PCR nachgewiesen werden kann.
Notiz:
Für jeden Test sollten Positiv-, Negativ-/Blindkontrollen und interne Kontrollkontrollen festgelegt werden, um die Gültigkeit der Versuchsergebnisse zu bewerten.
Schritt 3: Versuchsvorbereitung

Beobachten Sie vor der Anwendung, ob die Lösung gleichmäßig vermischt ist.Wenn Niederschlag festgestellt wird, muss dieser vor der Verwendung gemäß den Anweisungen aufgelöst und gemischt werden.Die 2×PCR-Mischung muss vor der Verwendung wiederholt mit einer Mikropipette pipettiert und gemischt werden, um eine ungleichmäßige Ionenverteilung zu vermeiden.
Notiz:
Nehmen Sie die Anweisungen heraus, lesen Sie sie sorgfältig durch und treffen Sie die Vorbereitungen vor dem Experiment in strikter Übereinstimmung mit den Anweisungen.
Schritt 4: PCR-Reaktionssystem vorbereiten
Mischen Sie gemäß dem Versuchsprotokoll die Primer H2O, 2×PCR mischen, zentrifugieren und auf jedes Reaktionsgefäß verteilen.
Notiz:
Für groß angelegte Tests oder Langzeittests wird die Verwendung eines PCR-Reaktionssystems mit UNG-Enzym empfohlen, mit dem eine Aerosolkontamination durch PCR-Produkte wirksam vermieden werden kann.
Schritt 5: Reaktionsvorlage hinzufügen

Durch die Verwendung der Direct-PCR-Technologie ist kein langwieriger Nukleinsäure-Reinigungsprozess erforderlich.Die Probenvorlage kann innerhalb von 10 Minuten vorbereitet und dem entsprechenden PCR-Reaktionssystem hinzugefügt werden.
Notiz:
Die Lysemethode hat einen besseren Nachweiseffekt und das erhaltene Produkt kann für mehrere Nachweisreaktionen verwendet werden.
5.1: Direkte PCR von Blättern
Schneiden Sie entsprechend der Größe des Bildes im Handbuch das Blattgewebe mit einem Durchmesser von 2–3 mm ab und legen Sie es in das PCR-Reaktionssystem.
Hinweis: Stellen Sie sicher, dass die Blattfragmente vollständig in die PCR-Reaktionslösung eingetaucht sind, und fügen Sie nicht zu viel Blattgewebe hinzu.
5.2: Blattlysemethode
Schneiden Sie das Blattgewebe mit einem Durchmesser von 5–7 mm ab und geben Sie es in ein Zentrifugenröhrchen.Wenn Sie reife Blätter wählen, vermeiden Sie bitte die Verwendung des Gewebes der Hauptader des Blattes.Pipettieren Sie 50 µl Puffer-P1-Lysat in ein Zentrifugenröhrchen, um sicherzustellen, dass das Lysat das Blattgewebe vollständig umhüllen kann, legen Sie es in einen Thermocycler oder ein Metallbad und lysieren Sie es 5–10 Minuten lang bei 95 °C.


50 µl Puffer P2-Neutralisierungslösung hinzufügen und gut vermischen.Das resultierende Lysat kann als Vorlage verwendet und dem PCR-Reaktionssystem hinzugefügt werden.
Hinweis: Die Menge der Vorlage sollte zwischen 5 und 10 % des PCR-Systems betragen und 20 % nicht überschreiten (fügen Sie beispielsweise in einem 20-μl-PCR-System 1–2 μl Lysepuffer hinzu, nicht mehr als 4 μl).
Schritt 6: PCR-Reaktion

Nachdem Sie das PCR-Reaktionsröhrchen zentrifugiert haben, legen Sie es zur Amplifikation in ein PCR-Gerät.
Notiz:
Die Reaktion verwendet eine nicht gereinigte Matrize zur Amplifikation, sodass die Anzahl der Amplifikationszyklen 5–10 Zyklen höher ist als bei Verwendung einer gereinigten DNA-Matrize.
Schritt 7: Elektrophorese-Erkennung und Ergebnisanalyse

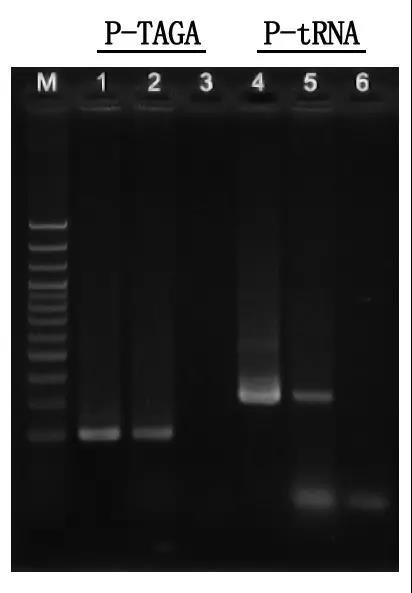
M:100bp DNA-Leiter
1\4: Gereinigte DNA-Methode
2\5: Direkte PCR-Methode
3\6: Leerkontrolle
Qualitätskontrolle:
Die Testergebnisse der verschiedenen im Experiment festgelegten Kontrollen sollten die folgenden Bedingungen erfüllen.Andernfalls sollte die Ursache des Problems analysiert und der Test nach Beseitigung des Problems erneut durchgeführt werden.
Tabelle 1. Normale Testergebnisse verschiedener Kontrollgruppen

*Wenn das Plasmid als Positivkontrolle verwendet wird, kann das Ergebnis des endogenen Gentests negativ sein
Ergebnisurteil:
A. Das Testergebnis des endogenen Gens der Probe ist negativ, was darauf hinweist, dass die für den normalen PCR-Nachweis geeignete DNA nicht aus der Probe extrahiert werden kann oder die extrahierte DNA PCR-Reaktionsinhibitoren enthält und die DNA erneut extrahiert werden sollte.
B. Das Testergebnis des endogenen Gens der Probe ist positiv und das Testergebnis des exogenen Gens ist negativ, was darauf hinweist, dass die für den gewöhnlichen PCR-Nachweis geeignete DNA aus der Probe extrahiert wurde und man daraus schließen kann, dass das XXX-Gen in der Probe nicht nachgewiesen wird.
C. Das Testergebnis des endogenen Gens der Probe ist positiv, und das Testergebnis des exogenen Gens ist positiv, was darauf hinweist, dass die für den gewöhnlichen PCR-Nachweis geeignete DNA aus der Probe extrahiert wurde und die Proben-DNA das XXX-Gen enthält.Es können weiterhin Bestätigungsexperimente durchgeführt werden.
Schritt 8: Entwerfen Sie Erkennungsprimer

Wischen Sie nach dem Experiment den Versuchsbereich mit 2 %iger Natriumhypochloritlösung und 70 %iger Ethanollösung ab, um eine Umweltverschmutzung zu vermeiden.
Anhang
Tabelle 2. Häufig verwendete Primer für den allgemeinen PCR-Nachweis gentechnisch veränderter Pflanzen
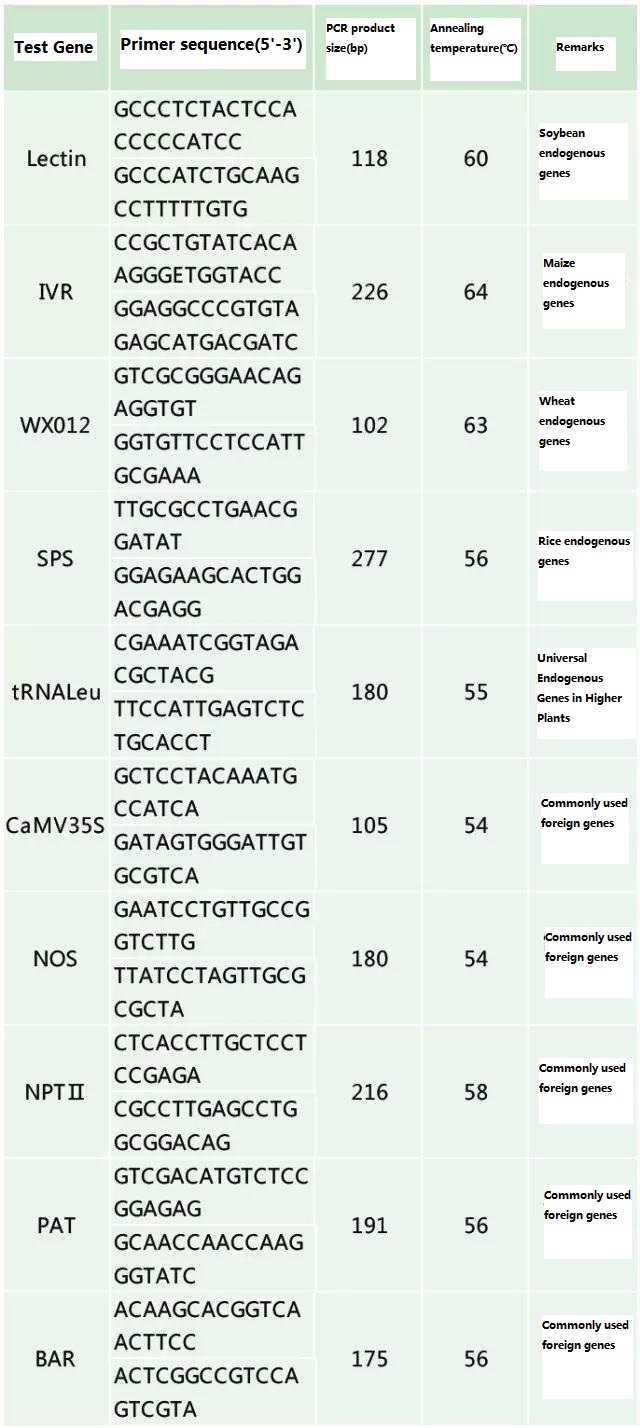
Referenzdokument:
SN/T 1202-2010, Qualitative PCR-Nachweismethode für gentechnisch veränderte Pflanzeninhaltsstoffe in Lebensmitteln.
Bekanntmachung des Landwirtschaftsministeriums 1485-5-2010, Prüfung der Inhaltsstoffe gentechnisch veränderter Pflanzen und ihrer Produkte – Reis M12 und seine Derivate.
Zeitpunkt der Veröffentlichung: 09.06.2021








