一、Erhöhen Sie die Empfindlichkeit des Reaktionssystems:
1. Hochwertige RNA isolieren:
Eine erfolgreiche cDNA-Synthese basiert auf hochwertiger RNA.Hochwertige RNA sollte mindestens in voller Länge und frei von Reverse-Transkriptase-Inhibitoren wie EDTA oder SDS sein.Die Qualität der RNA bestimmt die maximale Menge an Sequenzinformationen, die Sie in cDNA transkribieren können.Eine gängige RNA-Reinigungsmethode ist eine einstufige Methode unter Verwendung von Guanidinisothiocyanat/Säurephenol.Um eine Kontamination durch Spuren von RNase zu verhindern, muss RNA, die aus RNase-reichen Proben (z. B. Bauchspeicheldrüse) isoliert wurde, in Formaldehyd gelagert werden, um hochwertige RNA zu erhalten, insbesondere für die Langzeitlagerung.Die aus Rattenleber extrahierte RNA war nach einwöchiger Lagerung in Wasser im Wesentlichen abgebaut, während die aus Rattenmilz extrahierte RNA nach dreijähriger Lagerung in Wasser stabil blieb.Darüber hinaus reagieren Transkripte, die länger als 4 kb sind, empfindlicher auf den Abbau durch Spuren-RNasen als kleine Transkripte.Um die Stabilität gelagerter RNA-Proben zu erhöhen, kann RNA in entionisiertem Formamid gelöst und bei -70 °C gelagert werden.Formamid, das zur Konservierung von RNA verwendet wird, muss frei von RNA-abbauenden Ablagerungen sein.RNA aus der Bauchspeicheldrüse kann in Formamid mindestens ein Jahr lang konserviert werden.Wenn Sie die Verwendung von RNA vorbereiten, können Sie die folgende Methode zur Ausfällung von RNA verwenden: 0,2 M NaCl und das 4-fache Volumen Ethanol hinzufügen, 3–5 Minuten bei Raumtemperatur stehen lassen und 5 Minuten bei 10.000 × g zentrifugieren.
2. Verwenden Sie die RNaseH-inaktive (RNaseH-) Reverse Transkriptase:
RNase-Inhibitoren werden häufig zu Reverse-Transkriptionsreaktionen hinzugefügt, um die Länge und Ausbeute der cDNA-Synthese zu erhöhen.RNase-Inhibitoren sollten während der Erststrang-Synthesereaktion in Gegenwart eines Puffers und eines Reduktionsmittels (z. B. DTT) zugesetzt werden, da der Prozess vor der cDNA-Synthese den Inhibitor denaturiert und dadurch gebundene RNase freisetzt, die RNA abbauen kann.Protein-RNase-Inhibitoren verhindern nur den Abbau von RNA durch RNase A, B, C und nicht die RNase auf der Haut. Achten Sie daher darauf, dass Sie trotz der Verwendung dieser Inhibitoren keine RNase über Ihre Finger einführen.
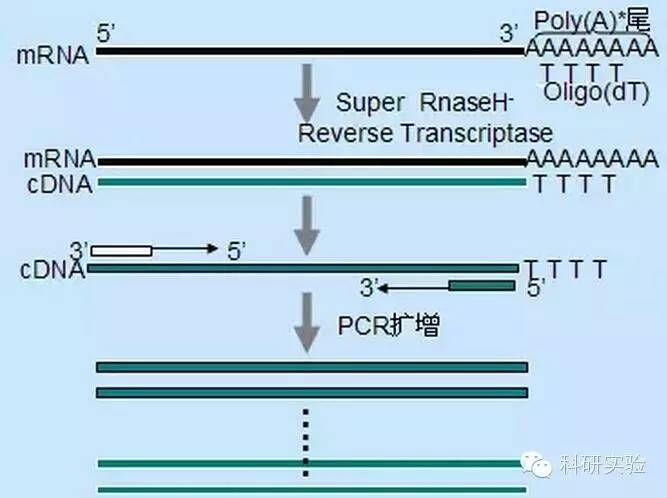
Reverse Transkriptase katalysiert die Umwandlung von RNA in cDNA.Sowohl M-MLV als auch AMV verfügen zusätzlich zu ihrer eigenen Polymeraseaktivität über eine endogene RNaseH-Aktivität.RNaseH-Aktivität und Polymeraseaktivität konkurrieren miteinander um den Hybridstrang, der zwischen der RNA-Matrize und dem DNA-Primer oder dem cDNA-Verlängerungsstrang gebildet wird, und bauen den RNA-Strang im RNA:DNA-Komplex ab.Die durch die RNaseH-Aktivität abgebaute RNA-Matrize kann nicht länger als wirksames Substrat für die cDNA-Synthese dienen, was die Ausbeute und Länge der cDNA-Synthese verringert.Daher wäre es von Vorteil, die RNaseH-Aktivität der Reversen Transkriptase zu eliminieren oder stark zu reduzieren.。
SuperScript Ⅱ Reverse Transkriptase, RNaseH-MMLV Reverse Transkriptase und ThermoScript Reverse Transkriptase, RNaseH-AMV, können mehr cDNA in voller Länge als MMLV und AMV erhalten.Die RT-PCR-Empfindlichkeit wird durch die Menge der cDNA-Synthese beeinflusst.ThermoScript ist viel empfindlicher als AMV.Die Größe von RT-PCR-Produkten wird durch die Fähigkeit der Reversen Transkriptase zur Synthese von cDNA begrenzt, insbesondere bei der Klonierung größerer cDNAs.Im Vergleich zu MMLV steigerte SuperScripⅡ die Ausbeute an langen RT-PCR-Produkten deutlich.Die RNaseH-reverse Transkriptase weist außerdem eine erhöhte Thermostabilität auf, sodass die Reaktion bei Temperaturen über den normalen 37–42 °C durchgeführt werden kann.Unter den empfohlenen Synthesebedingungen verwenden Sie Oligo(dT)-Primer und 10 μCi [α-P]dCTP.Die Gesamtausbeute des ersten Strangs wurde mithilfe der TCA-Fällungsmethode berechnet.Die cDNA voller Länge wurde unter Verwendung größensortierter Banden analysiert, die auf einem alkalischen Agarosegel ausgeschnitten und gezählt wurden.
3. Erhöhen Sie die Inkubationstemperatur für die umgekehrte Transkription:
Eine höhere Inkubationstemperatur trägt zur Öffnung der RNA-Sekundärstruktur bei und erhöht so die Ausbeute der Reaktion.Bei den meisten RNA-Vorlagen werden die meisten Sekundärstrukturen durch Inkubation der RNA und Primer bei 65 °C ohne Puffer oder Salz und anschließendes schnelles Abkühlen auf Eis entfernt und die Bindung der Primer ermöglicht.Allerdings weisen einige Template auch nach Hitzedenaturierung noch Sekundärstrukturen auf.Die Amplifikation dieser schwierigen Vorlagen kann mit ThermoScript Reverse Transcriptase durchgeführt werden und die Reverse-Transkriptionsreaktion bei einer höheren Temperatur durchgeführt werden, um die Amplifikation zu verbessern.Höhere Inkubationstemperaturen können auch die Spezifität erhöhen, insbesondere wenn genspezifische Primer (GSP) für die cDNA-Synthese verwendet werden (siehe Kapitel 3).Stellen Sie bei Verwendung von GSP sicher, dass die Tm der Primer mit der erwarteten Inkubationstemperatur übereinstimmt.Verwenden Sie keine Oligo(dT)- und Zufallsprimer über 60 °C.Zufällige Primer erfordern eine 10-minütige Inkubation bei 25 °C, bevor sie auf 60 °C erhöht werden.Zusätzlich zur Verwendung einer höheren Reverse-Transkriptionstemperatur kann die Spezifität auch verbessert werden, indem das RNA/Primer-Gemisch direkt von der 65 °C-Denaturierungstemperatur auf die Reverse-Transkriptions-Inkubationstemperatur übertragen und ein vorgewärmtes 2x-Reaktionsgemisch hinzugefügt wird (cDNA-Hot-Start-Synthese).Dieser Ansatz trägt dazu bei, die intermolekulare Basenpaarung zu verhindern, die bei niedrigeren Temperaturen auftritt.Der für die RT-PCR erforderliche mehrfache Temperaturwechsel kann durch den Einsatz eines Thermocyclers vereinfacht werden.
Die thermostabile Tth-Polymerase fungiert in Gegenwart von Mg2+ als DNA-Polymerase und in Gegenwart von Mn2+ als RNA-Polymerase.Es kann bei einer maximalen Temperatur von 65°C warm gehalten werden.Das Vorhandensein von Mn2+ während der PCR verringert jedoch die Wiedergabetreue, wodurch die Tth-Polymerase weniger für eine hochpräzise Amplifikation, wie z. B. das Klonen von cDNA, geeignet ist.Darüber hinaus weist Tth eine geringe Reverse-Transkriptions-Effizienz auf, was die Empfindlichkeit verringert, und da Reverse-Transkription und PCR mit einem einzigen Enzym durchgeführt werden können, können Kontrollreaktionen ohne Reverse-Transkription nicht zum Vergleich von cDNA-Amplifikationsprodukten mit kontaminierender genomischer DNA verwendet werden.Die Amplifikationsprodukte wurden getrennt.
4. Zusatzstoffe, die die umgekehrte Transkription fördern:
Der Erststrang-Synthesereaktion werden Zusatzstoffe wie Glycerin und DMSO zugesetzt, die die Stabilität des Nukleinsäure-Doppelstrangs verringern und die Sekundärstruktur der RNA auflösen können.Bis zu 20 % Glycerin oder 10 % DMSO können hinzugefügt werden, ohne die SuperScript II- oder MMLV-Aktivität zu beeinträchtigen.AMV verträgt auch bis zu 20 % Glycerin ohne Aktivitätsverlust.Um die Empfindlichkeit der RT-PCR in der SuperScriptⅡ-Reverse-Transkriptionsreaktion zu maximieren, können 10 % Glycerin hinzugefügt und bei 45 °C inkubiert werden.Wenn 1/10 des Reverse-Transkriptions-Reaktionsprodukts der PCR zugesetzt wird, beträgt die Glycerinkonzentration in der Amplifikationsreaktion 0,4 %, was nicht ausreicht, um die PCR zu hemmen.
5. RNaseH-Behandlung:
Die Behandlung von cDNA-Synthesereaktionen mit RNaseH vor der PCR kann die Empfindlichkeit erhöhen.Bei einigen Templates wird angenommen, dass RNA in der cDNA-Synthesereaktion die Bindung von Amplifikationsprodukten verhindert. In diesem Fall kann eine RNaseH-Behandlung die Empfindlichkeit erhöhen.Im Allgemeinen ist eine RNaseH-Behandlung erforderlich, wenn längere cDNA-Zielvorlagen voller Länge amplifiziert werden, wie z. B. tuberöse Scherosis II mit geringer Kopiezahl.Bei dieser schwierigen Matrize verstärkte die RNaseH-Behandlung das von SuperScript II oder AMV-synthetisierter cDNA erzeugte Signal.Bei den meisten RT-PCR-Reaktionen ist die RNaseH-Behandlung optional, da der PCR-Denaturierungsschritt bei 95 °C im Allgemeinen die RNA im RNA:DNA-Komplex hydrolysiert.
6. Verbesserung der Methode zum Nachweis kleiner RNA:
Eine besondere Herausforderung stellt die RT-PCR dar, wenn nur geringe Mengen an RNA verfügbar sind.Glykogen, das während der RNA-Isolierung als Träger hinzugefügt wird, trägt dazu bei, die Ausbeute kleiner Proben zu erhöhen.RNase-freies Glykogen kann gleichzeitig mit der Zugabe von Trizol hinzugefügt werden.Glykogen ist wasserlöslich und kann mit RNA in der wässrigen Phase gehalten werden, um die anschließende Fällung zu unterstützen.Für Proben mit weniger als 50 mg Gewebe oder 106 kultivierten Zellen beträgt die empfohlene Konzentration an RNase-freiem Glykogen 250 μg/ml.
Die Zugabe von acetyliertem BSA zur Reverse-Transkriptionsreaktion mit SuperScript II kann die Empfindlichkeit erhöhen, und bei kleinen RNA-Mengen kann die Reduzierung der SuperScript II-Menge und die Zugabe von 40 Einheiten RNaseOut-Nukleaseinhibitor den Nachweisgrad erhöhen.Wenn im RNA-Isolierungsprozess Glykogen verwendet wird, wird dennoch empfohlen, BSA oder RNase-Inhibitor hinzuzufügen, wenn SuperScript II für die Reverse-Transkriptionsreaktion verwendet wird.
二、Erhöhen Sie die RT-PCR-Spezifität
1. CND-Asynthese:
Die Erststrang-cDNA-Synthese kann mit drei verschiedenen Methoden initiiert werden, deren relative Spezifität die Menge und Art der synthetisierten cDNA beeinflusst.
Die Random-Primer-Methode war die am wenigsten spezifische der drei Methoden.Primer verbinden sich an mehreren Stellen im gesamten Transkript und erzeugen so kurze cDNAs mit Teillänge.Diese Methode wird häufig verwendet, um 5′-Endsequenzen und cDNA aus RNA-Matrizen mit Regionen mit Sekundärstruktur oder mit Terminationsstellen zu erhalten, die durch Reverse Transkriptase nicht repliziert werden können.Um die längste cDNA zu erhalten, muss das Verhältnis von Primern zu RNA in jeder RNA-Probe empirisch bestimmt werden.Die Ausgangskonzentration der Zufallsprimer lag zwischen 50 und 250 ng pro 20 μl Reaktion.Da es sich bei cDNA, die mithilfe von Zufallsprimern aus Gesamt-RNA synthetisiert wird, hauptsächlich um ribosomale RNA handelt, wird im Allgemeinen Poly(A)+RNA als Matrize gewählt.
Oligo(dT)-Primer sind spezifischer als Zufallsprimer.Es hybridisiert mit dem Poly(A)-Schwanz, der sich am 3′-Ende der meisten eukaryotischen mRNAs befindet.Da Poly(A)+-RNA etwa 1 bis 2 % der Gesamt-RNA ausmacht, ist die Menge und Komplexität der cDNA viel geringer als bei Zufallsprimern.Aufgrund seiner hohen Spezifität erfordert Oligo(dT) im Allgemeinen keine Optimierung des Verhältnisses von RNA zu Primern und der Poly(A)+-Selektion.Es wird empfohlen, 0,5 μg Oligo(dT) pro 20 μl Reaktionssystem zu verwenden.oligo(dT)12-18 ist für die meisten RT-PCR geeignet.Das ThermoScript RT-PCR-System bietet Oligo(dT)20 aufgrund seiner besseren thermischen Stabilität für höhere Inkubationstemperaturen.
Genspezifische Primer (GSP) sind die spezifischsten Primer für den Reverse-Transkriptionsschritt.GSP ist ein Antisense-Oligonukleotid, das im Gegensatz zu Zufallsprimern oder Oligo(dT), die sich an alle RNAs anlagern, spezifisch an die RNA-Zielsequenz hybridisieren kann.Für das Design von GSP in Reverse-Transkriptions-Reaktionen gelten die gleichen Regeln wie für das Design von PCR-Primern.Das GSP kann die gleiche Sequenz wie der Amplifikationsprimer haben, der sich an das 3′-äußerste Ende der mRNA anlagert, oder das GSP kann so konzipiert sein, dass es stromabwärts des Reverse-Amplifikationsprimers anlagert.Für einige amplifizierte Probanden muss für eine erfolgreiche RT-PCR mehr als ein Antisense-Primer entwickelt werden, da die Sekundärstruktur der Ziel-RNA die Primerbindung verhindern kann.Es wird empfohlen, 1 pmol Antisense-GSP in einer 20 μl Erststrang-Synthesereaktion zu verwenden.
2. Erhöhen Sie die Inkubationstemperatur für die umgekehrte Transkription:
Um den vollen Nutzen aus der GSP-Spezifität zu ziehen, sollte eine Reverse Transkriptase mit höherer Thermostabilität verwendet werden.Thermostabile Reverse Transkriptasen können bei höheren Temperaturen inkubiert werden, um die Reaktionsstringenz zu erhöhen.Wenn beispielsweise ein GSP bei 55 °C anlagert, wird die Spezifität des GSP nicht vollständig ausgenutzt, wenn AMV oder M-MLV für die reverse Transkription bei einer niedrigen Stringenz von 37 °C verwendet wird.SuperScript II und ThermoScript können jedoch bei 50 °C oder höher umgesetzt werden, wodurch unspezifische Produkte, die bei niedrigeren Temperaturen entstehen, eliminiert werden.Für maximale Spezifität kann der RNA/Primer-Mix direkt von der 65 °C Denaturierungstemperatur auf die Reverse-Transkriptions-Inkubationstemperatur überführt und zu einem vorgewärmten 2x-Reaktionsmix hinzugefügt werden (Heißstart der cDNA-Synthese).Dadurch wird eine intermolekulare Basenpaarung bei niedrigen Temperaturen verhindert.Die für die RT-PCR erforderlichen mehrfachen Temperaturübergänge können durch den Einsatz eines Thermocyclers vereinfacht werden.
3. Reduziert die Kontamination genomischer DNA:
Eine potenzielle Schwierigkeit bei der RT-PCR ist die Kontamination der genomischen DNA in der RNA.Durch die Verwendung einer guten RNA-Isolierungsmethode, wie z. B. Trizol-Reagenz, wird die Menge an genomischer DNA reduziert, die das RNA-Präparat verunreinigt.Um Produkte zu vermeiden, die aus genomischer DNA stammen, kann RNA vor der reversen Transkription mit DNase I in Amplifikationsqualität behandelt werden, um kontaminierende DNA zu entfernen.Der DNase I-Verdau wurde durch 10-minütige Inkubation der Proben in 2,0 mM EDTA bei 65 °C beendet.EDTA kann Magnesiumionen chelatisieren und so die Magnesiumionen-abhängige RNA-Hydrolyse bei hohen Temperaturen verhindern.
Um amplifizierte cDNA von kontaminierenden genomischen DNA-Amplifikationsprodukten zu trennen, können Primer entworfen werden, die jeweils an separate Exons anlagern.Aus cDNA abgeleitete PCR-Produkte sind kürzer als solche, die aus kontaminierter genomischer DNA stammen.Darüber hinaus wurde an jeder RNA-Matrize ein Kontrollexperiment ohne reverse Transkription durchgeführt, um festzustellen, ob ein bestimmtes Fragment von genomischer DNA oder cDNA stammte.Das ohne Reverse Transkription erhaltene PCR-Produkt stammt aus dem Genom.
Zeitpunkt der Veröffentlichung: 16. Mai 2023








