Ⅰ. Erhöhen Sie die Empfindlichkeit des Reaktionssystems:
1. Hochwertige RNA trennen:
Eine erfolgreiche cDNA-Synthese basiert auf hochwertiger RNA.Hochwertige RNA sollte mindestens eine Gesamtlänge gewährleisten und enthält keine Inhibitoren, die keine Aufnahmeenzyme wie EDTA oder SDS enthalten.Die Qualität der RNA bestimmt den maximalen Wert der Sequenzinformationen, die Sie in die cDNA übertragen können.Die allgemeine RNA-Reinigungsmethode ist eine Stufenmethode unter Verwendung von Isocyanat/Acidophenol.Um die Verschmutzung durch RNase zu verhindern, muss die RNA, die aus einer RNase-reichen Probe (z. B. Bauchspeicheldrüse) abgetrennt wurde, in Formaldehyd gelagert werden, um hochwertige RNA zu erhalten, was umso mehr für die Langzeitlagerung gilt.Die aus der Rattenleber extrahierte RNA war nach einer Woche Lagerung in Wasser im Wesentlichen abgebaut, während die aus der Rattenmilz extrahierte RNA nach drei Jahren Lagerung in Wasser stabil blieb.Darüber hinaus reagieren Transkripte, die größer als 4 kb sind, empfindlicher auf den Spurenabbau von RNase als kleine Transkripte.Um die Stabilität der gelagerten RNA-Probe zu erhöhen, kann die RNA in einem Methalamin von Ionen gelöst und bei -70 °C gelagert werden.Thylid, das zur Rettung von RNA verwendet wird, darf kein sonstiges Objekt enthalten, das RNA abbaut.RNA, die aus der Bauchspeicheldrüse stammt, kann in Methalmamin mindestens ein Jahr lang aufbewahrt werden.Wenn Sie bereit sind, RNA zu verwenden, können Sie die folgenden Methoden zur Ausfällung von RNA verwenden: NaCl zu 0,2 m und das 4-fache Volumen Ethanol hinzufügen, 3–5 Minuten bei Raumtemperatur stehen lassen und 5 Minuten bei 10.000 × g zentrifugieren.
2. Verwenden Sie Reverse Transkriptase ohne RNaseH-Aktivität (RNaseH-):
RNase-Inhibitoren werden häufig zu Reverse-Transkriptionsreaktionen hinzugefügt, um die Länge und Ausbeute der cDNA-Synthese zu erhöhen.Der RNase-Inhibitor wird in der ersten Kettensynthesereaktion in Gegenwart von Puffern und Reduktionsmitteln wie DTT hinzugefügt, da der Prozess vor der cDNA-Synthese den Inhibitor denaturiert und dadurch gebundene RNasen freisetzt, die die RNA abbauen.Protein-RNase-Inhibitoren verhindern nur den Abbau von RNA durch RNase A, B, C und verhindern nicht die Bildung von RNasen auf der Haut. Daher sollte trotz der Verwendung dieser Inhibitoren darauf geachtet werden, keine RNasen über die Finger einzuführen.
Reverse Transkriptase katalysiert die Umwandlung von RNA in cDNA.Sowohl M-MLV als auch AMV verfügen zusätzlich zu ihrer eigenen Polymeraseaktivität über eine endogene RNaseH-Aktivität.Die RNaseH-Aktivität konkurriert mit der Polymeraseaktivität um heterozygote Stränge, die zwischen RNA-Matrizen und DNA-Primern oder cDNA-Verlängerungssträngen gebildet werden, und baut RNA:RNA-Stränge in DNA-Komplexen ab.Durch RNaseH-Aktivität abgebaute RNA-Templates können nicht mehr als wirksame Substrate für die cDNA-Synthese verwendet werden, was die Ausbeute und Länge der cDNA-Synthese verringert.Daher wäre es von großem Nutzen, die RNaseH-Aktivität der Reversen Transkriptase zu eliminieren oder stark zu reduzieren.
SuperScriptⅡ Reverse Transkriptase, MMLV Reverse Transkriptase von RNaseH- und ThermoScript Reverse Transkriptase, AMV von RNaseH- ergaben mehr cDNA voller Länge als MMLV und AMV.Die RT-PCR-Empfindlichkeit wird durch die Menge der synthetisierten cDNA beeinflusst.ThermoScript ist viel empfindlicher als AMV.Die Größe von RT-PCR-Produkten wird durch die Fähigkeit der Reversen Transkriptase zur Synthese von cDNA begrenzt, insbesondere bei der Klonierung größerer Cdnas.Im Vergleich zu MMLV steigerte SuperScripⅡ die Ausbeute an langen RT-PCR-Produkten deutlich.Die Reverse Transkriptase von RNaseH- erhöht auch die thermische Stabilität, sodass die Reaktion bei höheren Temperaturen als normal von 37–42 °C durchgeführt werden kann.Unter den vorgeschlagenen Synthesebedingungen wurden Oligo(dT)-Primer und 10μCi [alpha-p]dCTP verwendet.Die Gesamtproduktion der ersten Kette wurde mit der TCA-Fällungsmethode berechnet.Die cDNA voller Länge wurde mittels größensortierter Streifenentfernung und Zählung in einem alkalischen Agarosegel analysiert.
3. Erhöhen Sie die Wärmekonservierungstemperatur der reversen Transkription:
Eine höhere Haltetemperatur trägt dazu bei, die Sekundärstruktur der RNA zu öffnen und die Ausbeute der Reaktion zu erhöhen.Bei den meisten RNA-Vorlagen werden die meisten Sekundärstrukturen entfernt, wenn die RNA und der Primer ohne Puffer oder Salz bei 65 °C gehalten und dann schnell auf Eis abgekühlt werden, und die Primer können binden.Einige Template weisen jedoch auch nach thermischer Denaturierung noch eine Sekundärstruktur auf.Die Amplifikation dieser schwierigen Vorlagen kann mithilfe der ThermoScript-Reverse-Transkriptase und durch die Durchführung der Reverse-Transkriptase-Reaktion bei höheren Temperaturen durchgeführt werden, um die Amplifikation zu verbessern.Höhere Haltetemperaturen können auch die Spezifität erhöhen, insbesondere wenn die cDNA-Synthese mithilfe genspezifischer Primer (GSPS) durchgeführt wird (siehe Kapitel 3).Stellen Sie bei Verwendung von GSP sicher, dass der Tm-Wert des Primers mit der erwarteten Haltetemperatur übereinstimmt.Verwenden Sie keine Oligo(dT)- und Zufallsprimer über 60℃.Zufallsprimer müssen 10 Minuten lang bei 25 °C gehalten werden, bevor sie auf 60 °C erhöht werden.Zusätzlich zur Verwendung höherer Reverse-Transkriptionstemperaturen kann die Spezifität verbessert werden, indem die RNA/Primer-Mischung direkt von der Denaturierungstemperatur von 65 °C auf die Reverse-Transkriptions-Haltetemperatur übertragen und eine vorgewärmte 2x-Reaktionsmischung hinzugefügt wird (thermische cDNA-Initiationssynthese).Dieser Ansatz trägt dazu bei, die intermolekulare Basenpaarung zu verhindern, die bei niedrigeren Temperaturen auftritt.Die Verwendung eines PCR-Instruments vereinfacht die vielen Temperaturschalter, die für die RT-PCR erforderlich sind.
Die hitzestabilisierte Tth-Polymerase fungiert in Gegenwart von Mg2+ als DNA-Polymerase und in Gegenwart von Mn2+ als RNA-Polymerase.Es kann Wärme bis zu 65℃ halten.Das Vorhandensein von Mn2+ während der PCR verringert jedoch die Wiedergabetreue, wodurch die Tth-Polymerase für eine hochpräzise Amplifikation, wie z. B. die cDNA-Klonierung, weniger geeignet ist.Darüber hinaus ist Tth bei der reversen Transkription weniger effizient, was die Empfindlichkeit verringert, und da ein einzelnes Enzym reverse Transkription und PCR durchführen kann, können Kontrollreaktionen ohne reverse Transkription nicht verwendet werden, um amplifizierte cDNA-Produkte von denen kontaminierter genomischer DNA zu unterscheiden.
4. Zusatzstoff, der die umgekehrte Transkription fördert:
Die Zugabe von Zusatzstoffen, darunter Glycerin und DMSO, zur ersten Kettensynthesereaktion kann die Stabilität des Nukleinsäure-Doppelstrangs verringern und die RNA-Sekundärstruktur aufwickeln.Bis zu 20 % Glycerin oder 10 % DMSO können hinzugefügt werden, ohne die Aktivität von SuperScriptⅡ oder MMLV zu beeinträchtigen.AMV kann auch bis zu 20 % Glycerin vertragen, ohne die Aktivität zu verringern.Um die Empfindlichkeit der RT-PCR in der SuperScriptⅡ-Reverse-Transkriptionsreaktion zu maximieren, können 10 % Glycerin hinzugefügt und bei 45 °C isoliert werden.Wenn 1/10 des Retrotranskriptionsreaktionsprodukts zur PCR hinzugefügt wird, beträgt die Glycerinkonzentration in der Amplifikationsreaktion 0,4 %, was nicht ausreicht, um die PCR zu hemmen.
5. RNaseH-Verarbeitung:
Die Empfindlichkeit kann verbessert werden, indem cDNA-Synthesereaktionen vor der PCR mit RNaseH behandelt werden.Bei einigen Templates wird angenommen, dass die RNA in der cDNA-Synthesereaktion die Bindung amplifizierter Produkte verhindert. In diesem Fall kann die RNaseH-Behandlung die Empfindlichkeit erhöhen.Im Allgemeinen ist eine RNaseH-Behandlung für die Amplifikation einer relativ langen cDNA-Zielvorlage voller Länge erforderlich, wie z. B. tuberöse ScherosisⅡ mit geringer Kopie.Für diese schwierige Matrize verstärkte RNaseH das Signal, das von der von SuperScriptⅡ oder AMV synthetisierten cDNA erzeugt wurde.Bei den meisten RT-PCR-Reaktionen ist die RNaseH-Behandlung optional, da der 95℃-isolierte PCR-Denaturierungsschritt typischerweise die RNA aus dem RNA:DNA-Komplex hydrolysiert.
6. Verbesserte Methoden zum Nachweis kleiner RNA-Mengen:
Eine besondere Herausforderung stellt die RT-PCR dar, wenn nur geringe Mengen an RNA verfügbar sind.Die Zugabe von Glykogen als Träger während der RNA-Trennung trägt dazu bei, die Ausbeute kleiner Proben zu erhöhen.Gleichzeitig mit Trizol kann ein RNase-freies Glykogen hinzugefügt werden.Glykogen ist wasserlöslich und kann zusammen mit der RNA in der Wasserphase verbleiben, um die anschließende Ausfällung zu unterstützen.Die empfohlene Konzentration an RNase-freiem Glykogen beträgt 250 μg/ml für Proben mit weniger als 50 mg Gewebe oder 106 kultivierten Zellen.
Die Zugabe von acetyliertem BSA zu Reverse-Transkriptionsreaktionen mit SuperScriptⅡ kann die Empfindlichkeit erhöhen, und bei kleinen RNA-Mengen kann die Reduzierung der SuperScriptⅡ-Menge und die Zugabe von 40 Einheiten RnaseOut-Nukleaseinhibitor den Nachweisgrad verbessern.Wenn Glykogen bei der RNA-Trennung verwendet wird, wird weiterhin die Zugabe von BSA- oder RNase-Inhibitoren zu Reverse-Transkriptionsreaktionen mit SuperScriptⅡ empfohlen.
Ⅱ. Erhöhen Sie die Spezifität der RT-PCR
1. cNDA-Synthese:
Drei verschiedene Methoden können verwendet werden, um die Erststrang-cDNA-Synthese zu initiieren, und die relative Spezifität jeder Methode beeinflusst die Menge und Art der synthetisierten cDNA.
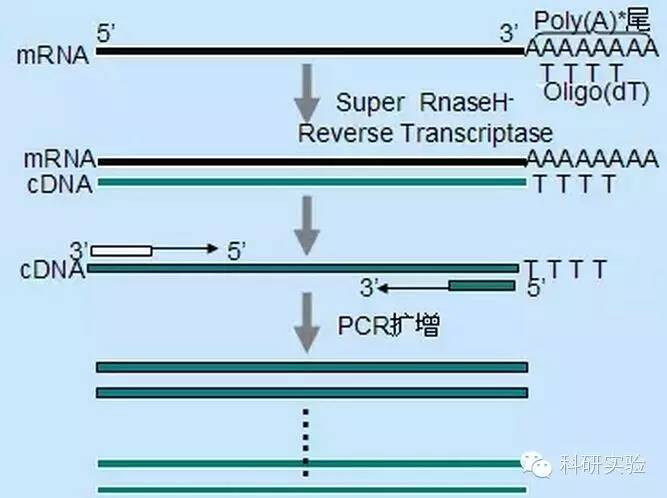
Die Random-Primer-Methode ist die am wenigsten spezifische der drei Methoden.Primer werden an mehreren Stellen im gesamten Transkript angelagert, um kurze cDNA mit Teillänge zu erzeugen.Diese Methode wird häufig verwendet, um 5′-terminale Sequenzen und cDNA aus RNA-Vorlagen mit sekundären Strukturregionen oder mit Terminationsstellen zu erhalten, die die Reverse Transkriptase nicht replizieren kann.Um die längste cDNA zu erhalten, muss das Verhältnis von Primern zu RNA in jeder RNA-Probe empirisch bestimmt werden.Die anfängliche Konzentration an Zufallsprimern liegt zwischen 50 und 250 ng pro 20 μl Reaktionssystem.Da es sich bei der aus Gesamt-RNA unter Verwendung von Zufallsprimern synthetisierten cDNA hauptsächlich um ribosomale RNA handelt, wird im Allgemeinen Poly(A)+RNA als Matrize ausgewählt.
Die Oligo(dT)-Initiation ist spezifischer als Zufallsprimer.Es hybridisiert mit dem Poly(A)-Schwanz, der sich in den meisten eukaryontischen Zellen am 3′-Ende der mRNA befindet.Da Poly(A)+RNA etwa 1 bis 2 % der gesamten RNA ausmacht, ist die Menge und Komplexität der cDNA viel geringer, als wenn Zufallsprimer verwendet würden.Aufgrund seiner hohen Spezifität erfordert Oligo(dT) im Allgemeinen keine Optimierung des RNA-Primer-Verhältnisses und der Poly(A)+-Selektion.Es wird empfohlen, 0,5 μg Oligo(dT) pro 20 μl Reaktionssystem zu verwenden.oligo(dT)12-18 ist für die meisten RT-PCR geeignet.Das ThermoScript RT-PCR-System bietet Oligo(dT)20 aufgrund seiner guten thermischen Stabilität und ist für höhere Haltetemperaturen geeignet.
Genspezifische Primer (GSP) sind die besten spezifischen Primer für den Reverse-Transkriptionsschritt.GSP ist ein Antisense-Oligonukleosid, das spezifisch mit RNA-Zielsequenzen hybridisieren kann, anstatt alle Rnas wie Zufallsprimer oder Oligo(dT) zu verbinden.Die zum Design von PCR-Primern verwendeten Regeln gelten auch für das Design der Reverse-Transkriptionsreaktion GSP.GSP kann die gleiche Sequenz haben wie der Amplifikationsprimer, der am Ende von mRNA3′ angelagert ist, oder GSP kann so konzipiert sein, dass es stromabwärts mit dem umgekehrten Amplifikationsprimer angelagert wird.Für einige amplifizierte Objekte ist es für eine erfolgreiche RT-PCR erforderlich, mehr als einen Antisense-Primer zu entwerfen, da die Sekundärstruktur der Ziel-RNA möglicherweise die Bindung des Primers verhindert.Es wird empfohlen, 1 pmol Antisense-GSP im ersten Kettensynthesereaktionssystem von 20 μl zu verwenden.
2. Erhöhen Sie die Wärmekonservierungstemperatur der reversen Transkription:
Um die GSP-Spezifität voll auszunutzen, sollte Reverse Transkriptase mit hoher thermischer Stabilität verwendet werden.Hitzestabile Reverse Transkriptase kann bei höheren Temperaturen isoliert werden, um die Reaktionsgenauigkeit zu erhöhen.Wenn beispielsweise ein GSP bei 55 °C angelagert wird, wird die Spezifität von GSP nicht vollständig ausgenutzt, wenn die reverse Transkription bei 37 °C mit geringer Genauigkeit unter Verwendung von AMV oder M-MLV durchgeführt wird.SuperScripⅡ und ThermoScript können jedoch bei 50℃ oder höher reagieren, wodurch unspezifische Produkte ausgeschlossen werden, die bei niedrigeren Temperaturen entstehen.Für maximale Spezifität kann die RNA/Primer-Mischung durch Zugabe einer vorgewärmten 2-fach-Reaktionsmischung (thermische Initiierung der cDNA-Synthese) direkt von der Denaturierungstemperatur von 65 °C auf die Haltetemperatur für die reverse Transkription überführt werden.Dadurch wird die Basenpaarung zwischen Molekülen bei niedrigen Temperaturen verhindert.Die Verwendung eines PCR-Instruments vereinfacht die vielen Temperaturübergänge, die für die RT-PCR erforderlich sind.
3. Reduzieren Sie die Kontamination genomischer DNA:
Eine mögliche Schwierigkeit bei der RT-PCR besteht darin, dass RNA die genomische DNA kontaminiert.Der Einsatz besserer RNA-Trennmethoden, wie z. B. Trizol-Reagenz, reduziert die Kontamination genomischer DNA in RNA-Präparaten.Um Produkte zu vermeiden, die aus genomischer DNA entstehen, kann die RNA vor der reversen Transkription mit DnasⅠ in Amplifikationsqualität behandelt werden, um kontaminierte DNA zu entfernen.Die Proben wurden 10 Minuten lang bei 65 °C in 2,0 mM EDTA gehalten, um den DNaseⅠ-Verdau zu beenden.EDTA chelatisiert Magnesiumionen, um die Magnesiumionen-abhängige RNA-Hydrolyse zu verhindern, die bei hohen Temperaturen auftritt.
Um amplifizierte cDNA vom Genom-DNA-Amplifikationsprodukt zu trennen, können Primer entworfen werden, die sich separat mit dem abgetrennten Exon verbinden.Aus cDNA abgeleitete PCR-Produkte sind kürzer als solche, die aus kontaminierter genomischer DNA stammen.An jeder RNA-Matrize wird außerdem ein kontrolliertes Experiment ohne reverse Transkription durchgeführt, um festzustellen, ob ein bestimmtes Fragment aus genomischer DNA oder cDNA stammt.PCR-Produkte, die ohne reverse Transkription erhalten werden, werden aus dem Genom abgeleitet.
Ähnliches Produkt
RT-PCR einfachᵀᴹIch (einen Schritt)
-Das One-Step-Kit ermöglicht die Durchführung von Reverse Transkription und PCR im selben Röhrchen.Es müssen lediglich Template-RNA, spezifische PCR-Primer und RNase-freies ddH hinzugefügt werden2O.
-Die quantitative Analyse von RNA in Echtzeit kann schnell und genau durchgeführt werden.
-Das Kit verwendet ein einzigartiges Foregene-Reverse-Transkriptionsreagenz und Foregene HotStar Taq DNA-Polymerase in Kombination mit einem einzigartigen Reaktionssystem, um die Amplifikationseffizienz und Spezifität der Reaktion effektiv zu verbessern.
-Das optimierte Reaktionssystem sorgt dafür, dass die Reaktion eine höhere Nachweisempfindlichkeit, eine stärkere thermische Stabilität und eine bessere Toleranz aufweist.
-Effiziente Fähigkeit zur Entfernung von gDNA, wodurch gDNA in der Vorlage innerhalb von 2 Minuten entfernt werden kann.
-Effizientes Reverse-Transkriptionssystem, es dauert nur 15 Minuten, um die Synthese des ersten cDNA-Strangs abzuschließen.
-Komplexe Vorlagen: Vorlagen mit hohem GC-Gehalt und komplexer Sekundärstruktur können auch mit hoher Effizienz umgekehrt werden.
-Hochempfindliches Reverse-Transkriptionssystem, Vorlagen auf pg-Ebene können auch hochwertige cDNA erhalten.
-Das Reverse-Transkriptionssystem weist eine hohe thermische Stabilität auf, die optimale Reaktionstemperatur liegt bei 42℃ und es weist auch bei 50℃ immer noch eine gute Reverse-Transkriptionsleistung auf.
Zeitpunkt der Veröffentlichung: 07.03.2023