1. Grundkenntnisse (wenn Sie den experimentellen Teil sehen möchten, übertragen Sie ihn bitte direkt auf den zweiten Teil)
Als abgeleitete Reaktion der herkömmlichen PCR überwacht die Echtzeit-PCR hauptsächlich die Änderung der Menge des Amplifikationsprodukts in jedem Zyklus der PCR-Amplifikationsreaktion in Echtzeit durch die Änderung des Fluoreszenzsignals und analysiert die Ausgangsvorlage quantitativ durch die Beziehung zwischen dem ct-Wert und der Standardkurve.
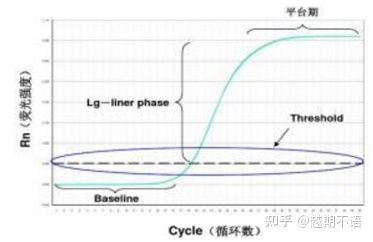
Die spezifischen Daten der RT-PCR sindGrundlinie, FluoreszenzschwelleUndCt-Wert.
| Grundlinie: | Der Fluoreszenzwert des 3.-15. Zyklus ist die Grundlinie (Basislinie), die durch gelegentliche Fehler der Messung verursacht wird. |
| Schwelle (Schwelle): | Bezieht sich auf die Fluoreszenz-Nachweisgrenze, die an einer geeigneten Position im exponentiellen Wachstumsbereich der Amplifikationskurve eingestellt ist, im Allgemeinen das Zehnfache der Standardabweichung der Basislinie. |
| CT-Wert: | Dies ist die Anzahl der PCR-Zyklen, bei denen der Fluoreszenzwert in jedem Reaktionsgefäß den Schwellenwert erreicht. Der Ct-Wert ist umgekehrt proportional zur Menge der anfänglichen Vorlage. |
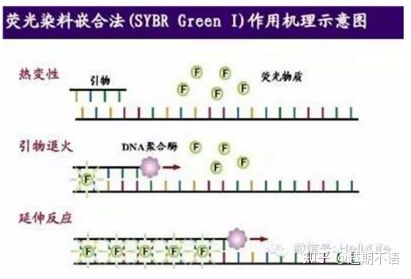
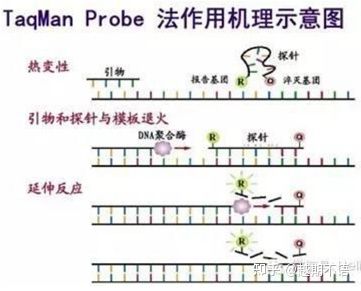
Gängige Markierungsmethoden für RT-PCR:
| Methode | Vorteil | Mangel | Geltungsbereich |
| SYBR GrünⅠ | Breite Anwendbarkeit, empfindlich, günstig und bequem | Die Primeranforderungen sind hoch und neigen zu unspezifischen Banden | Es eignet sich für die quantitative Analyse verschiedener Zielgene, die Erforschung der Genexpression und die Erforschung transgener rekombinanter Tiere und Pflanzen. |
| TaqMan | Gute Spezifität und hohe Wiederholgenauigkeit | Der Preis ist hoch und nur für bestimmte Ziele geeignet. | Erkennung von Krankheitserregern, Erforschung von Medikamentenresistenzgenen, Beurteilung der Wirksamkeit von Medikamenten, Diagnose genetischer Krankheiten. |
| molekulares Leuchtfeuer | Hohe Spezifität, Fluoreszenz, geringer Hintergrund | Der Preis ist hoch, es ist nur für einen bestimmten Zweck geeignet, das Design ist schwierig und der Preis ist hoch. | Spezifische Genanalyse, SNP-Analyse |
2. Experimentelle Schritte
2.1 Über die experimentelle Gruppierung- Es müssen mehrere Vertiefungen in der Gruppe vorhanden sein und es müssen biologische Wiederholungen vorhanden sein.
| ① | Leerkontrolle | Wird verwendet, um den Zellwachstumsstatus in Experimenten zu ermitteln |
| ② | Negativkontroll-siRNA (unspezifische siRNA-Sequenz) | Demonstrieren Sie die Spezifität der RNAi-Wirkung.siRNA kann bei einer Konzentration von 200 nM eine unspezifische Stressreaktion auslösen. |
| ③ | Transfektionsreagenzkontrolle | Schließen Sie die Toxizität des Transfektionsreagenzes für die Zellen oder die Auswirkung auf die Expression des Zielgens aus |
| ④ | siRNA gegen Zielgen | Unterdrücken Sie die Expression des Zielgens |
| ⑤ (optional) | positive siRNA | Wird zur Fehlerbehebung bei experimentellen System- und Betriebsproblemen verwendet |
| ⑥ (optional) | Fluoreszenzkontroll-siRNA | Die Effizienz der Zelltransfektion kann mit einem Mikroskop beobachtet werden |
2.2 Prinzipien des Primerdesigns
| Verstärkte Fragmentgröße | Vorzugsweise bei 100–150 bp |
| Primerlänge | 18-25bp |
| GC-Inhalt | 30 %–70 %, vorzugsweise 45 %–55 % |
| Tm-Wert | 58-60℃ |
| Reihenfolge | Vermeiden Sie T/C-Kontinuität;A/G kontinuierlich |
| 3 Endsequenz | Vermeiden Sie GC-reiche oder AT-reiche Produkte;die terminale Base ist vorzugsweise G oder C;Am besten meidet man T |
| Komplementarität | Vermeiden Sie komplementäre Sequenzen von mehr als 3 Basen innerhalb des Primers oder zwischen zwei Primern |
| Spezifität | Verwenden Sie die Explosionssuche, um die Primerspezifität zu bestätigen |
①SiRNA ist artspezifisch und die Sequenzen verschiedener Arten sind unterschiedlich.
②SiRNA ist in gefriergetrocknetem Pulver verpackt, das bei Raumtemperatur 2–4 Wochen stabil gelagert werden kann.
2.3 Werkzeuge oder Reagenzien, die im Voraus vorbereitet werden müssen
| Primer (interne Referenz) | Einschließlich Vorwärts- und Rückwärtsgang zwei |
| Primer (Zielgen) | Einschließlich Vorwärts- und Rückwärtsgang zwei |
| Ziel-Si-RNA (3 Streifen) | Im Allgemeinen synthetisiert das Unternehmen drei Streifen und wählt dann mittels RT-PCR einen der drei aus |
| Transfektionskit | Lipo2000 usw. |
| RNA-Schnellextraktionskit | Zur RNA-Extraktion nach der Transfektion |
| Rapid Reverse Transkriptionskit | für die cDNA-Synthese |
| PCR-Amplifikationskit | 2×Super SYBR Grün qPCR-Mastermix |
2.4 Bezüglich der Probleme, auf die in den spezifischen experimentellen Schritten geachtet werden muss:
①siRNA-Transfektionsprozess
1. Zum Ausplattieren können Sie zwischen einer 24-Well-Platte, einer 12-Well-Platte oder einer 6-Well-Platte wählen (die vorgeschlagene durchschnittliche RNA-Konzentration in jedem Well einer 24-Well-Platte beträgt etwa 100–300 ng/ul), und die optimale Transfektionsdichte der Zellen beträgt etwa 60–80 %
2. Die Transfektionsschritte und spezifischen Anforderungen richten sich strikt nach den Anweisungen.
3. Nach der Transfektion können innerhalb von 24–72 Stunden Proben für den mRNA-Nachweis (RT-PCR) oder den Proteinnachweis innerhalb von 48–96 Stunden (WB) entnommen werden.
② RNA-Extraktionsprozess
1. Verhindern Sie eine Kontamination durch exogene Enzyme.Dazu gehört vor allem das strikte Tragen von Masken und Handschuhen;Verwendung sterilisierter Pipettenspitzen und EP-Röhrchen;Das im Experiment verwendete Wasser muss RNase-frei sein.
2. Es wird empfohlen, zweimal so viel wie im Schnellextraktionsset empfohlen durchzuführen, um die Reinheit und Ausbeute deutlich zu verbessern.
3. Die Abfallflüssigkeit darf die RNA-Säule nicht berühren.
③ RNA-Quantifizierung
Nachdem die RNA extrahiert wurde, kann sie direkt mit Nanodrop quantifiziert werden, und der Mindestwert kann bis zu 10 ng/ul betragen.
④Reverse Transkriptionsprozess
1. Aufgrund der hohen Empfindlichkeit der RT-qPCR sollten für jede Probe mindestens 3 parallele Vertiefungen erstellt werden, um zu verhindern, dass die nachfolgenden Ct-Werte zu unterschiedlich sind oder die SD für eine statistische Analyse zu groß wird.
2. Den Mastermix nicht wiederholt einfrieren und auftauen.
3. Jedes Rohr/Loch muss durch eine neue Spitze ersetzt werden!Verwenden Sie zum Hinzufügen von Proben nicht ständig dieselbe Pipettenspitze!
4. Der nach Zugabe der Probe auf der 96-Well-Platte angebrachte Film muss mit einer Platte geglättet werden.Am besten zentrifugieren Sie es, bevor Sie es in die Maschine stellen, damit die Flüssigkeit an der Röhrchenwand nach unten fließen und Luftblasen entfernen kann.
⑤Gemeinsame Kurvenanalyse
| Keine Periode logarithmischen Wachstums | Möglicherweise hohe Templatkonzentration |
| Kein CT-Wert | Falsche Schritte zur Erkennung von Fluoreszenzsignalen; Abbau von Primern oder Sonden – ihre Integrität kann durch PAGE-Elektrophorese nachgewiesen werden; unzureichende Menge an Vorlage; Abbau von Templaten – Vermeidung der Einführung von Verunreinigungen und wiederholtem Einfrieren und Auftauen bei der Probenvorbereitung; |
| Ct>38 | Geringe Verstärkungseffizienz;PCR-Produkt ist zu lang;Verschiedene Reaktionskomponenten werden abgebaut |
| Lineare Verstärkungskurve | Die Sonden können durch wiederholte Gefrier-Tau-Zyklen oder längere Lichteinwirkung teilweise beschädigt werden |
| Besonders groß ist der Unterschied bei doppelten Löchern | Die Reaktionslösung ist nicht vollständig geschmolzen oder die Reaktionslösung ist nicht vermischt;Das Thermalbad des PCR-Geräts ist durch fluoreszierende Substanzen verunreinigt |
2.5 Über Datenanalyse
Die Datenanalyse von qPCR kann in relative Quantifizierung und absolute Quantifizierung unterteilt werden.Beispielsweise wurden Zellen in der Behandlungsgruppe mit Zellen in der Kontrollgruppe verglichen.
Wie oft sich die mRNA des X-Gens ändert, ist eine relative Quantifizierung;in einer bestimmten Anzahl von Zellen die mRNA des X-Gens
Wie viele Exemplare es gibt, das ist eine absolute Quantifizierung.Normalerweise verwenden wir im Labor am häufigsten die relative quantitative Methode.Normalerweise,die 2-ΔΔct-Methodewird am häufigsten in Experimenten verwendet, daher wird hier nur diese Methode im Detail vorgestellt.
2-ΔΔct-Methode: Das erhaltene Ergebnis ist der Unterschied in der Expression des Zielgens in der Versuchsgruppe im Vergleich zum Zielgen in der Kontrollgruppe.Es ist erforderlich, dass die Amplifikationseffizienz sowohl des Zielgens als auch des internen Referenzgens nahezu 100 % beträgt und die relative Abweichung 5 % nicht überschreiten darf.
Die Berechnungsmethode ist wie folgt:
Δct-Kontrollgruppe = ct-Wert des Zielgens in der Kontrollgruppe – ct-Wert des internen Referenzgens in der Kontrollgruppe
Δct Experimentalgruppe = ct-Wert des Zielgens in der Experimentalgruppe – ct-Wert des internen Referenzgens in der Experimentalgruppe
ΔΔct = Δct Versuchsgruppe – Δct Kontrollgruppe
Berechnen Sie abschließend das Vielfache der Differenz im Ausdrucksniveau:
Change Fold=2-ΔΔct (entsprechend der Excel-Funktion ist POWER)
Verwandte Produkte:
Zeitpunkt der Veröffentlichung: 20. Mai 2023