Kürzlich habe ich etwas Erstaunliches entdeckt!Viele fortgeschrittene Experimentierprofis um ihn herum kennen einige sehr grundlegende experimentelle Wissenspunkte nicht einmal.
Können Sie beispielsweise die folgenden Fragen beantworten?
Gibt es einen Unterschied zwischen OD260 und A260?Was bedeutet jedes?
OD ist die Abkürzung für optische Dichte (optische Dichte), A ist die Abkürzung für Absorption (Absorption), die beiden Konzepte sind eigentlich gleich, „optische Dichte“ ist „Absorption“, aber „optische Dichte“ entspricht den meisten nationalen Standards und ist standardisierter.
Normalerweise messen wir den OD-Wert bei 260 nm, um die Nukleinsäurekonzentration zu berechnen. Was bedeutet also 1OD?
Nukleinsäure hat einen maximalen Absorptionspeak bei einer Wellenlänge von 260 nm und enthält sowohl DNA als auch RNA sowie fragmentierte Nukleinsäurefragmente (das ist der entscheidende Punkt).
Der bei einer Wellenlänge von 260 nm gemessene OD-Wert wurde als OD260 aufgezeichnet.Wenn die Probe rein ist, kann der OD260-Wert die Konzentration der Nukleinsäureprobe berechnen.
1 OD260=50 μg/ml dsDNA (doppelsträngige DNA)
=37 μg/ml ssDNA (einzelsträngige DNA)
=40 μg/ml RNA
=30 μg/ml dNTPs (Oligonukleotide)
Gibt es einen Zusammenhang und Unterschied zwischen RT-PCR, Realtime-PCR und QPCR?
RT-PCR ist die Abkürzung für Reverse Transcription PCR
Echtzeit-PCR = qPCR, kurz für Quantitative Real Time PCR
Obwohl Real-Time-PCR (quantitative Echtzeit-Fluoreszenz-PCR) und Reverse-Transkriptions-PCR (Reverse-Transkriptions-PCR) beide als RT-PCR abgekürzt zu werden scheinen.Aber die internationale Konvention lautet: RT-PCR bezieht sich speziell auf die Reverse-Transkriptions-PCR.
Welche nt, bp und kb werden üblicherweise zur Beschreibung der Länge von DNA/RNA in der Biologie verwendet?
nt = Nukleotid
bp = Basenpaar Basenpaar
kb = Kilobase
Natürlich würde man sagen, dass viele Leute sich nicht um diese kleinen Details kümmern!Jeder macht das, und niemand wird Sie fragen, was es ist.Sie wissen, dass das unnötig ist, oder?
Nein, nein, nein, das muss man unbedingt wissen!wegen welchem?
Weil Sie einen Artikel veröffentlichen möchten!Bruder!Egal, ob Sie einen Abschluss anstreben oder wissenschaftliche Forschungsleistungen anstreben, Sie müssen sich darauf verlassen, dass Artikel sprechen!
Die Nukleinsäureextraktion sollte das einfachste und grundlegendste Experiment sein.Die Qualität der Nukleinsäureextraktion bestimmt direkt die Ergebnisse nachfolgender Experimente.
Obwohl ich es schon oft gesagt habe, gibt es immer noch viele Freunde, denen es egal ist.Dieses Mal habe ich beschlossen, den Artikel zu verlassen!

Mindestinformationen für die Veröffentlichung quantitativer Echtzeit-PCR-Experimente, kurz MIQE genanntist eine Reihe internationaler Richtlinien für quantitative Fluoreszenzexperimente, die Mindeststandards für die experimentellen Informationen vorschlagen, die für die Bewertung quantitativer Fluoreszenz-PCR-Experimente und die Veröffentlichung von Artikeln erforderlich sind.Durch die vom Experimentator bereitgestellten experimentellen Bedingungen und Analysemethoden können die Gutachter die Gültigkeit des experimentellen Schemas des Forschers besser bewerten.
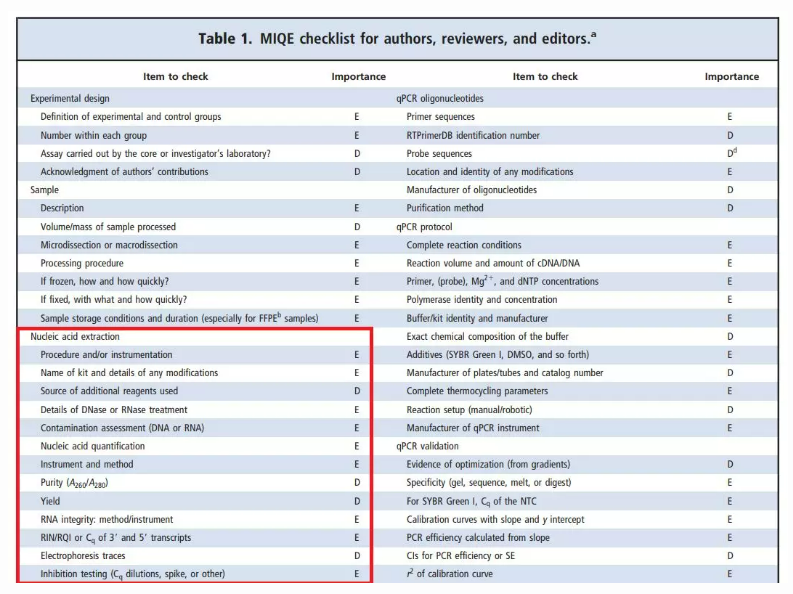
Es ist ersichtlich, dass im Bereich der Nukleinsäureextraktion die folgenden Nachweiselemente vorgeschlagen wurden:
„E“ weist auf Informationen hin, die bereitgestellt werden müssen, und „D“ weist auf Informationen hin, die bei Bedarf bereitgestellt werden sollten.
Das Formular ist sehr kompliziert, eigentlich möchte ich sagen, dass jeder damit beginnen muss
Reinheit (D), Ausbeute (D), Integrität (E) und Konsistenz (E) zur Bewertung von Nukleinsäuren in diesen vier Aspekten.
Sprechen Sie gemäß den experimentellen Gewohnheiten zunächst über die Bewertungsmethoden für Reinheit und Konzentration.
Die OD-Messung ist die beliebteste und einfachste Nachweismethode für Experimentatoren.Auf das Prinzip möchte ich hier nicht näher eingehen.Viele Labore nutzen mittlerweile Ultramikrospektrophotometer, um Nukleinsäureproben direkt quantitativ zu analysieren.Während der Absorptionswert angezeigt wird, gibt das Programm direkt den Konzentrationswert (Nukleinsäure, Protein und Fluoreszenzfarbstoff) und die zugehörigen Verhältnisse an.Was die Analyse des OD-Werts betrifft, speichern Sie dieses Bild, dann ist alles in Ordnung.
Liste der universellen OD-Wert-Lösungen
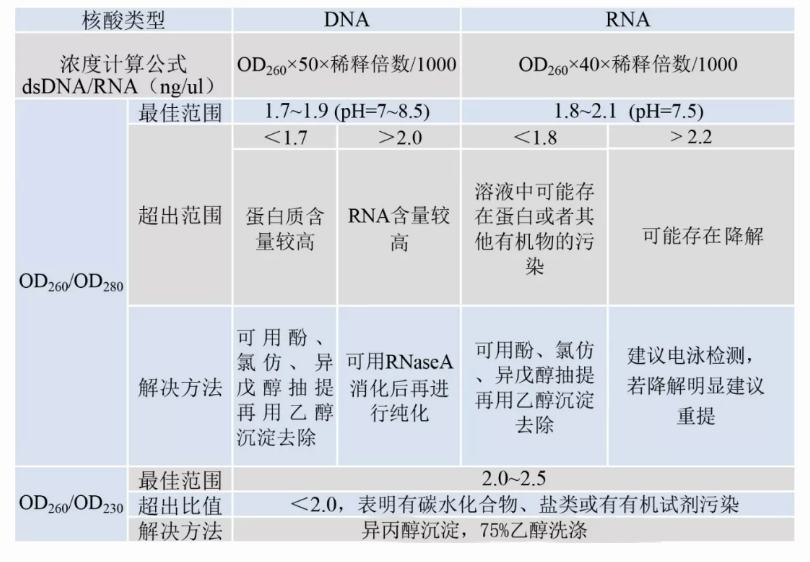 Es gibt jedoch einige Vorbehalte, die gesondert für Sie erläutert werden müssen.
Es gibt jedoch einige Vorbehalte, die gesondert für Sie erläutert werden müssen.
(Schließlich weiß ich, dass Sie diejenigen sein müssen, die sparen und warten, bis Sie sie brauchen!)
Anmerkung 1 Ausrüstung
Der OD-Wert wird durch unterschiedliche Geräte beeinflusst.Solange die OD260 innerhalb eines bestimmten Bereichs liegt, sind die Werte von OD230 und OD280 aussagekräftig.Beispielsweise beträgt der Absorptionsbereich des herkömmlichen Eppendorf D30 bei 260 nm 0 bis 3 A und der des NanoDrop One von Thermo liegt bei 260 nm.Der Absorptionsbereich liegt zwischen 0,5 und 62,5 A.
Anmerkung 2Verdünnungsreagenz
Der OD-Wert kann durch die Verdünnung verschiedener Reagenzien beeinflusst werden.Zum Beispiel der OD260/280-Wert gereinigter RNA im pH-Wert7,5 10 mM TrisDer Puffer liegt zwischen 1,9 und 2,1, während inneutrale wässrige LösungDas Verhältnis wird niedriger sein, vielleicht nur 1,8-2,0, aber das bedeutet nicht, dass sich die Qualität der RNA ändert.
Notiz 3Reststoffe
Das Vorhandensein von Reststoffen beeinträchtigt die Genauigkeit der Messung der Nukleinsäurekonzentration. Daher müssen Protein-, Phenol-, Polysaccharid- und Polyphenolrückstände in Nukleinsäureproben so weit wie möglich vermieden werden.
Tatsächlich ist die Extraktion mit organischen Reagenzien jedoch eine alte Methode.In kommerziellen Kits kann der Extraktionseffekt durch eine Adsorptionssäule auf Kieselsäurebasis in Kombination mit Zentrifugation erzielt werden, wodurch giftige und schädliche organische Reagenzien vermieden werden, die schwer zu entfernen sind usw. Das Problem, wie zDas Nukleinsäure-Extraktionskit von Foregene verwendet während des gesamten Vorgangs keine DNase/RNase und keine giftigen organischen Reagenzien, schnell und sicher, und dasWirkung istGut(habe versehentlich gesagt, dass es kahl sei, aber ich weiß, dass du es wissen willst).
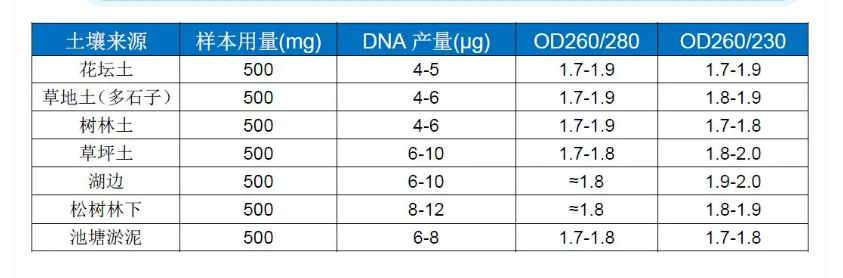
Beispiel 1: Ausbeute und Reinheit der genomischen DNA-Extraktion
Das Foregene Soil DNA Isolation Kit (DE-05511) behandelt Bodenproben aus verschiedenen Quellen. Die Menge und Reinheit der erhaltenen genomischen DNA ist in der folgenden Tabelle aufgeführt:
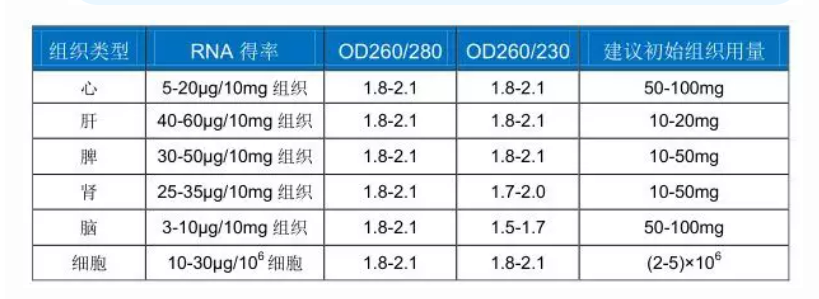
 Beispiel 2: Ausbeute und Reinheit der Gewebe-RNA-Extraktion
Beispiel 2: Ausbeute und Reinheit der Gewebe-RNA-Extraktion
Das Animal Total RNA Isolation Kit (RE-03012) verarbeitete verschiedene Gewebeproben. Die Menge und Reinheit der erhaltenen RNA sind in der folgenden Tabelle aufgeführt (für Mausgewebe):
 Denken Sie jedoch nicht, dass Sie mit dem OD-Wert fertig sind.Haben Sie einen Überblick über die wichtigsten Punkte, die ich vorne für Sie gezeichnet habe?
Denken Sie jedoch nicht, dass Sie mit dem OD-Wert fertig sind.Haben Sie einen Überblick über die wichtigsten Punkte, die ich vorne für Sie gezeichnet habe?
Notiz
Fragmentierte Nukleinsäuremoleküle werden ebenfalls in die Extinktion einbezogen.Unter der Annahme, dass die RNA genomische DNA-Reste enthält, scheint Ihr OD-Wert sehr hoch zu sein, die tatsächliche RNA-Konzentration kann jedoch nicht bestimmt werden.Ob es sich bei Ihrer RNA um einen Abbau handelt, ist nicht klar. Daher benötigen wir noch eine umfassende Bewertungsmethode, um eine genauere Beurteilung zu ermöglichen, nämlich die in MIQE erwähnte Bewertung der Nukleinsäureintegrität.
Zeitpunkt der Veröffentlichung: 13. Januar 2022








