Der mRNA-Impfstoff von Pfizer gegen COVID hat die Leidenschaft für die Verwendung von Ribonukleinsäure (RNA) als therapeutisches Ziel neu entfacht.Die gezielte Bekämpfung von RNA mit kleinen Molekülen ist jedoch äußerst schwierig.
RNA besteht nur aus vier Bausteinen: Adenin (A), Cytosin (C), Guanin (G) und Uracil (U), das das in der DNA vorkommende Thymin (T) ersetzt.Dies macht die Arzneimittelselektivität zu einem nahezu unüberwindbaren Hindernis.Im Gegensatz dazu gibt es 22 natürliche Aminosäuren, aus denen Proteine bestehen, was erklärt, warum die meisten Protein-Targeting-Medikamente eine relativ gute Selektivität aufweisen.
Struktur und Funktion der RNA
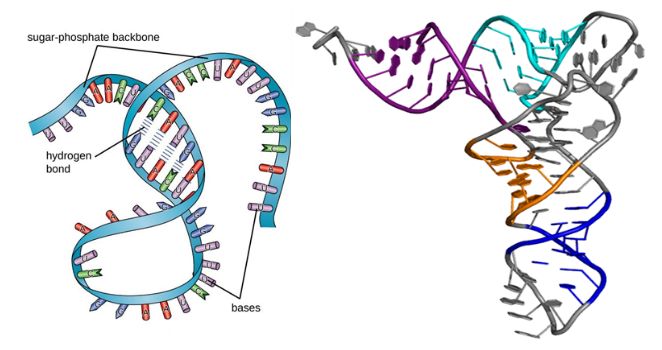
RNA-Moleküle haben wie Proteine Sekundär- und Tertiärstrukturen, wie in der Abbildung unten dargestellt.Obwohl es sich um einkettige Makromoleküle handelt, nimmt ihre Sekundärstruktur Gestalt an, wenn durch Basenpaarung Ausbuchtungen, Schleifen und Helices entstehen.Anschließend führt die dreidimensionale Faltung zur Tertiärstruktur der RNA, die für ihre Stabilität und Funktion wesentlich ist.
Abbildung 1. Struktur der RNA
Es gibt drei Arten von RNA:
- Messenger-RNA (mRNA)transkribiert genetische Informationen aus der DNA und wird als Basensequenz auf das Ribosom übertragen;l
- Ribosomale RNA (rRNA)ist Teil der proteinsynthetisierenden Organellen, den sogenannten Ribosomen, die in das Zytoplasma exportiert werden und dabei helfen, Informationen in mRNA in Proteine zu übersetzen;
- Transfer-RNA (tRNA)ist die Verbindung zwischen mRNA und der Aminosäurekette, aus der das Protein besteht.
Die gezielte Verwendung von RNA als therapeutisches Ziel ist sehr attraktiv.Es wurde festgestellt, dass nur 1,5 % unseres Genoms letztendlich in Protein übersetzt werden, während 70–90 % in RNA transkribiert werden.RNA-Moleküle sind die wichtigsten für alle lebenden Organismen.Laut Francis Cricks „zentralem Dogma“ besteht die wichtigste Rolle der RNA darin, genetische Informationen von der DNA in Proteine zu übersetzen.Darüber hinaus haben RNA-Moleküle noch weitere Funktionen, darunter:
- Wirken als Adaptermoleküle bei der Proteinsynthese;l
- Dient als Bote zwischen der DNA und dem Ribosom;l
- Sie sind Träger der genetischen Information in allen lebenden Zellen;l
- Förderung der ribosomalen Auswahl der richtigen Aminosäuren, die für die Synthese neuer Proteine notwendig sindin vivo.
Antibiotika
Obwohl der Wirkmechanismus vieler Antibiotika bereits in den 1940er-Jahren entdeckt wurde, konnte der Wirkungsmechanismus erst Ende der 1980er-Jahre aufgeklärt werden.Es wurde festgestellt, dass ein großer Teil der Antibiotika durch Bindung an bakterielle Ribosomen diese daran hindert, entsprechende Proteine zu bilden, wodurch die Bakterien abgetötet werden.
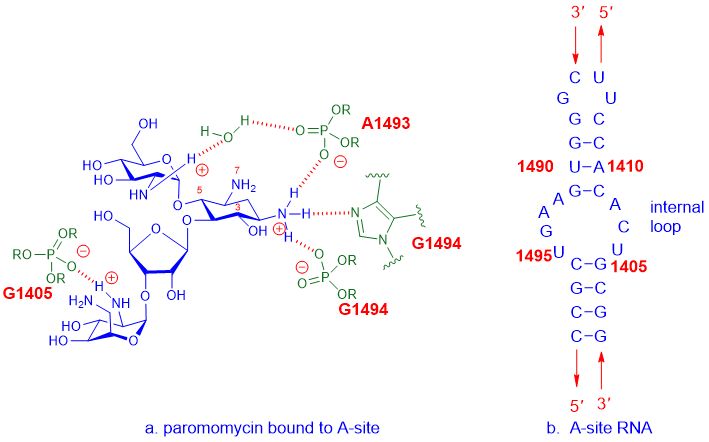
Beispielsweise binden Aminoglykosid-Antibiotika an die A-Stelle der 16S-rRNA, die Teil der 30S-Ribosomen-Untereinheit ist, und stören dann die Proteinsynthese, um das Bakterienwachstum zu beeinträchtigen, was letztendlich zum Zelltod führt.Die A-Stelle bezieht sich auf die Aminoacylstelle, auch bekannt als tRNA-Akzeptorstelle.Die detaillierte Wechselwirkung zwischen Aminoglykosid-Medikamenten, wie zParomomycin, und die A-Seite vonE coliDie RNA ist unten dargestellt.
Abbildung 2. Die Wechselwirkung zwischen Paromomycin und der A-Stelle vonE coliRNA
Leider weisen viele A-Stellen-Inhibitoren, einschließlich Aminoglykosid-Medikamente, Sicherheitsprobleme wie Nephrotoxizität, dosisabhängige und spezifische irreversible Ototoxizität auf.Diese Toxizitäten sind das Ergebnis einer mangelnden Selektivität von Aminoglykosid-Arzneimitteln zur Erkennung kleiner RNA-Moleküle.
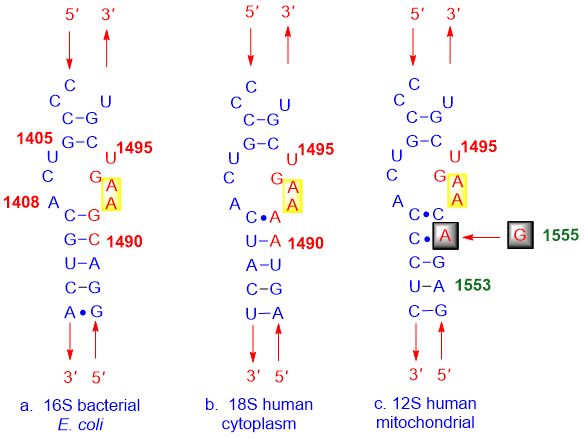
Wie in der Abbildung unten gezeigt: (a) sind die Strukturen der Bakterien, (b) die menschliche Zellmembran und (c) die menschliche mitochondriale A-Stelle sehr ähnlich, sodass A-Stellen-Inhibitoren an sie alle binden.
Abbildung 3. Die nicht-selektive Bindung des A-Stellen-Inhibitors
Tetracyclin-Antibiotika hemmen auch die A-Stelle der rRNA.Sie hemmen selektiv die bakterielle Proteinsynthese, indem sie reversibel an eine helikale Region (H34) der mit Mg komplexierten 30S-Untereinheit binden2+.
Andererseits binden Makrolid-Antibiotika in der Nähe der Austrittsstelle (E-Stelle) des bakteriellen Ribosomentunnels für entstehende Peptide (NPET) und blockieren diesen teilweise, wodurch die bakterielle Proteinsynthese gehemmt wird.Schließlich sind Oxazolidinon-Antibiotika wie zLinezolid(Zyvox) binden an einen tiefen Spalt in der bakteriellen ribosomalen 50S-Untereinheit, der von 23S-rRNA-Nukleotiden umgeben ist.
Antisense-Oligonukleotide (ASO)
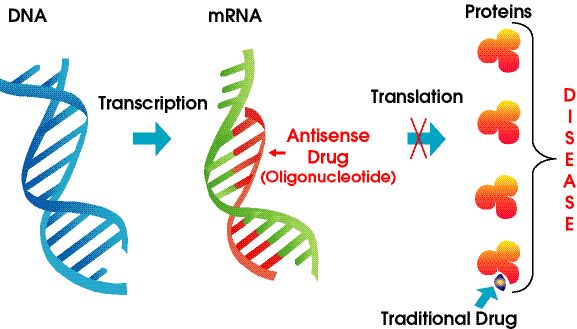
Bei den Antisense-Medikamenten handelt es sich um chemisch modifizierte Nukleinsäurepolymere, die auf RNA abzielen.Sie sind auf die Watson-Crick-Basenpaarung angewiesen, um an die Ziel-mRNA zu binden, was zu Gen-Stilllegung, sterischer Blockade oder Spleißveränderung führt.ASOs können mit Prä-RNAs im Zellkern und reifen mRNAs im Zytoplasma interagieren.Sie können auf Exons, Introns und untranslatierte Regionen (UTRs) abzielen.Bisher wurden mehr als ein Dutzend ASO-Medikamente von der FDA zugelassen.
Abbildung 4. Antisense-Technologie
Niedermolekulare Medikamente, die auf RNA abzielen
Im Jahr 2015 berichtete Novartis, dass sie einen SMN2-Spleißregulator namens Branaplam entdeckt hatten, der die Assoziation von U1-prä-mRNA verstärkt und SMA-Mäuse rettet.
Andererseits wurde Risdiplam (Evrysdi) von PTC/Roche im Jahr 2020 von der FDA für die Behandlung von SMA zugelassen.Wie Branaplam reguliert auch Risdiplam das Spleißen relevanter SMN2-Gene, um funktionelle SMN-Proteine zu produzieren.
RNA-Degrader
RBM steht für RNA-bindendes Motivprotein.Im Wesentlichen handelt es sich bei Indolsulfonamid um einen molekularen Klebstoff.Es rekrutiert RBM39 selektiv für die CRL4-DCAF15 E3-Ubiquitin-Ligase und fördert so die Polyubiquitinierung und den Proteinabbau von RBM39.Genetische Depletion oder Sulfonamid-vermittelter Abbau von RBM39 führt zu erheblichen genomweiten Spleißanomalien, die letztendlich zum Zelltod führen.
RNA-PROTACs werden entwickelt, um RNA-bindende Proteine (RBPs) abzubauen.PROTAC verwendet einen Linker, um den E3-Ligase-Liganden mit dem RNA-Liganden zu verbinden, der an RNA und RBPs bindet.Da RBP Strukturdomänen enthält, die an spezifische Oligonukleotidsequenzen binden können, verwendet RNA-PROTAC eine Oligonukleotidsequenz als Ligand für das Protein of Interest (POI).Das Endergebnis ist der Abbau von RBPs.
Kürzlich hat Professor Matthew Disney von der Scripps Institution of Oceanography die RNA erfundenRibonuklease-Targeting-Chimären (RiboTACs).RiboTAC ist ein heterofunktionelles Molekül, das einen RNase-L-Liganden und einen RNA-Liganden mit einem Linker verbindet.Es kann endogene RNase L gezielt für bestimmte RNA-Ziele rekrutieren und dann mithilfe des zellulären Nukleinsäureabbaumechanismus (RNase L) erfolgreich RNA eliminieren.
Da Forscher mehr über die Wechselwirkung zwischen kleinen Molekülen und RNA-Zielen erfahren, werden in Zukunft weitere Medikamente auf den Markt kommen, die diese Methode nutzen.
Zeitpunkt der Veröffentlichung: 02.08.2023