Die drei Buchstaben SNP sind in der Erforschung der Populationsgenetik allgegenwärtig.Unabhängig von der Erforschung menschlicher Krankheiten, der Positionierung von Pflanzenmerkmalen, der Evolution von Tieren und der Molekularökologie werden SNPs als Grundlage benötigt.Wenn Sie jedoch nicht über ein tiefes Verständnis der modernen Genetik auf der Grundlage der Hochdurchsatzsequenzierung verfügen und diese drei Buchstaben als „der vertrauteste Fremde“ betrachten, können Sie die anschließende Forschung nicht durchführen.Bevor wir also mit der weiteren Forschung beginnen, werfen wir einen Blick darauf, was SNP ist.
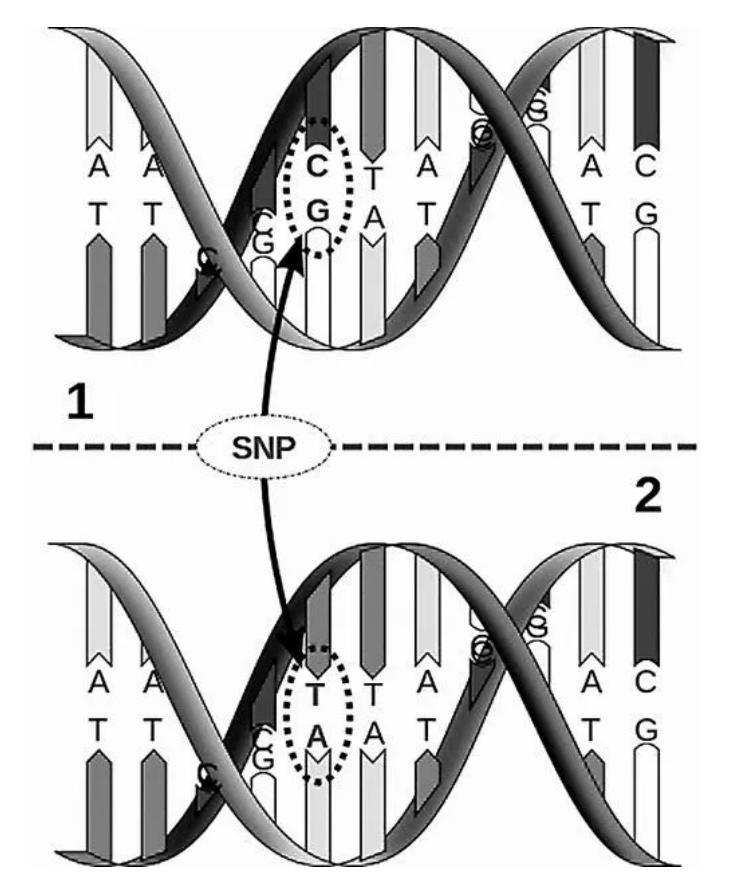
SNP (Single Nucleotide Polymorphism), wie wir aus dem vollständigen englischen Namen ersehen können, bezieht sich auf die Variation oder den Polymorphismus einzelner Nukleotide.Es hat auch einen separaten Namen, der SNV (Single Nucleotide Variation) heißt.In einigen Humanstudien werden nur solche mit einer Bevölkerungshäufigkeit von mehr als 1 % als SNPs bezeichnet, aber im Großen und Ganzen können die beiden gemischt werden.Wir können also sagen, dass sich SNP, Single Nucleotide Polymorphism, auf die Mutation bezieht, bei der ein Nukleotid im Genom durch ein anderes Nukleotid ersetzt wird.In der folgenden Abbildung wird beispielsweise ein AT-Basenpaar durch ein GC-Basenpaar ersetzt, bei dem es sich um eine SNP-Stelle handelt.
Bild
Unabhängig davon, ob es sich um einen „Einzelnukleotid-Polymorphismus“ oder eine „Einzelnukleotid-Variation“ handelt, ist es relativ gesehen, dass SNP-Daten eine Genom-Resequenzierung als Grundlage benötigen, das heißt, die Sequenzierungsdaten werden sequenziert, nachdem das Genom des Individuums sequenziert wurde.Im Vergleich zum Genom wird die Stelle, die sich vom Genom unterscheidet, als SNP-Stelle erkannt.
In Bezug auf SNPs auf Gemüse,das Plant Direct PCR-Kitkann zur schnellen Erkennung verwendet werden.
Hinsichtlich der Mutationstypen umfasst SNP Transition und Transversion.Unter Übergang versteht man den Ersatz von Purinen durch Purine oder von Pyrimidinen durch Pyrimidine.Unter Transversion versteht man die gegenseitige Substitution zwischen Purinen und Pyrimidinen.Die Häufigkeit des Auftretens wird unterschiedlich sein und die Wahrscheinlichkeit eines Übergangs wird höher sein als die einer Transversion.
Je nachdem, wo der SNP auftritt, haben verschiedene SNPs unterschiedliche Auswirkungen auf das Genom.SNPs, die in der intergenen Region vorkommen, also der Region zwischen Genen im Genom, beeinträchtigen möglicherweise nicht die Funktion des Genoms, und Mutationen im Intron oder in der vorgelagerten Promotorregion des Gens können einen gewissen Einfluss auf das Gen haben;Die Mutationen, die in den Exon-Regionen von Genen auftreten, haben je nachdem, ob sie Veränderungen in den kodierten Aminosäuren verursachen, unterschiedliche Auswirkungen auf die Genfunktionen.(Selbst wenn zwei SNPs Unterschiede in den Aminosäuren verursachen, haben sie natürlich unterschiedliche Auswirkungen auf die Struktur des Proteins, und letztendlich können die Auswirkungen auf den biologischen Phänotyp ganz unterschiedlich sein.)
Allerdings ist die Anzahl der SNPs, die an Genorten auftreten, in der Regel deutlich geringer als die an Nicht-Genorten, da ein SNP, der die Funktion eines Gens beeinflusst, in der Regel einen negativen Einfluss auf das Überleben des Individuums hat, was dazu führt, dass das Individuum, das diesen SNP trägt, in der Gruppe unter ihnen eliminiert wird.
Natürlich existieren die Chromosomen bei diploiden Organismen paarweise, aber es ist unmöglich, dass ein Chromosomenpaar für jede Base genau gleich ist.Daher erscheinen einige SNPs auch heterozygot, das heißt, es gibt zwei Basen an dieser Position auf dem Chromosom.In einer Gruppe werden die SNP-Genotypen verschiedener Individuen zusammengefasst, was die Grundlage für die meisten nachfolgenden Analysen bildet.In Kombination mit Merkmalen kann beurteilt werden, ob SNP als molekularer Marker mit Merkmalen verknüpft ist, der QTL (Quantitative Trait Locus) des Merkmals kann beurteilt werden und GWAS (Genomweite Assoziationsstudie) oder eine genetische Kartenkonstruktion können durchgeführt werden;SNP kann als molekularer Marker zur Beurteilung der evolutionären Beziehung zwischen Individuen verwendet werden;Sie können funktionelle SNPs untersuchen und krankheitsbedingte Mutationen untersuchen.Sie können SNP-Allelfrequenzänderungen oder heterozygote Raten und andere Indikatoren verwenden, um die ausgewählten Regionen im Genom usw. zu bestimmen, kombiniert mit der Gegenwart. Mit der Entwicklung der Hochdurchsatzsequenzierung können Hunderttausende oder mehr SNP-Sites aus einem Satz von Sequenzierungsdaten erhalten werden.Man kann sagen, dass SNP mittlerweile zum Eckpfeiler der populationsgenetischen Forschung geworden ist.
Natürlich handelt es sich bei den Veränderungen der Basen im Genom nicht immer um den Austausch einer Base durch eine andere (obwohl dies am häufigsten vorkommt).Es ist auch möglich, dass eine oder mehrere Basen fehlen, oder zwei Basen.In der Mitte wurden mehrere weitere Sockel eingefügt.Dieser kleine Bereich von Einfügungen und Löschungen wird zusammenfassend als InDel (Einfügung und Löschung) bezeichnet, was sich speziell auf die Einfügung und Löschung kurzer Fragmente (einer oder mehrerer Basen) bezieht.InDel, das an der Stelle eines Gens auftritt, kann sich auch auf die Funktion des Gens auswirken, sodass InDel manchmal auch eine wichtige Rolle in der Forschung spielen kann.Aber insgesamt ist der Status von SNP als Eckpfeiler der Populationsgenetik immer noch unerschütterlich.
Zeitpunkt der Veröffentlichung: 27. August 2021