Bei qPCR-Experimenten ist auch das Primerdesign ein sehr wichtiges Bindeglied.Ob die Primer geeignet sind oder nicht, hängt eng damit zusammen, ob die Amplifikationseffizienz den Standard erreicht, ob die amplifizierten Produkte spezifisch sind und ob die experimentellen Ergebnisse verfügbar sind.
Wie kann man also die Spezifität von qPCR-Primern verbessern?Hohe Verstärkungseffizienz?
Heute zeigen wir Ihnen, wie Sie gemeinsam qPCR-Primer entwerfen und das Design von qPCR-Primern zu einer effizienten Wissenskompetenz in Experimenten werden lassen.
Achten Sie beim Entwerfen von qPCR-Primern normalerweise auf die folgenden Punkte: Primer sollten so weit wie möglich über Introns hinweg entworfen werden, die Produktlänge sollte 100–300 bp betragen, der Tm-Wert sollte so nahe wie möglich bei 60 ° C liegen und die Upstream- und Downstream-Primer sollten so nahe wie möglich beieinander liegen und das Ende des Primers sollte G oder C usw. sein. Warten Sie.
1. Design von Primern, die Introns überspannen
Bei der Entwicklung von qPCR-Primern kann die Wahl von intronübergreifenden Primern verhindern, dass die gDNA-Matrize amplifiziert wird, und die Produkte stammen alle aus der Amplifikation von cDNA, wodurch der Einfluss einer gDNA-Kontamination eliminiert wird.
2. Primerlänge
Die Primerlänge liegt im Allgemeinen zwischen 18 und 30 nt, und die Länge des Amplifikationsprodukts sollte so weit wie möglich zwischen 100 und 300 bp kontrolliert werden.
Wenn der Primer zu kurz ist, führt er zu einer unspezifischen Amplifikation, und wenn er zu lang ist, bildet er leicht eine Sekundärstruktur (z. B. eine Haarnadelstruktur).Wenn das Amplifikationsprodukt zu lang ist, ist es nicht für die Polymerasereaktion geeignet, was die Effizienz der PCR-Amplifikation beeinträchtigt.
3. GC-Gehalt und Tm-Wert
Der GC-Gehalt der Primer sollte zwischen 40 % und 60 % liegen.Ist er zu hoch oder zu niedrig, ist er nicht förderlich für die Auslösung der Reaktion.Der GC-Gehalt der Vorwärts- und Rückwärtsprimer sollte nahezu gleich sein, um den gleichen Tm-Wert und die gleiche Annealing-Temperatur zu erhalten.
Der Tm-Wert sollte so weit wie möglich zwischen 55 und 65 °C liegen, im Allgemeinen bei etwa 60 °C, und der Tm-Wert von stromaufwärts und stromabwärts sollte so nahe wie möglich beieinander liegen, vorzugsweise nicht mehr als 4 °C.
4. Vermeiden Sie die Auswahl von A am 3′-Ende des Primers
Wenn das 3′-Ende des Primers nicht übereinstimmt, gibt es große Unterschiede in der Syntheseeffizienz verschiedener Basen.Wenn die letzte Base A ist, kann sie auch im Falle einer Fehlpaarung die Kettensynthese initiieren, und wenn die letzte Base T ist, ist die Effizienz der Fehlpaarungsinduktion stark verringert.Vermeiden Sie daher die Auswahl von A am 3′-Ende des Primers und wählen Sie besser T.
Wenn es sich um einen Sondenprimer handelt, kann das 5′-Ende der Sonde nicht G sein, denn selbst wenn eine einzelne G-Base mit der FAM-Fluoreszenzreportergruppe verbunden ist, kann G auch das von der FAM-Gruppe emittierte Fluoreszenzsignal löschen, was zu falsch negativen Ergebnissen führt.Erscheinen.
5. Basisverteilung
Die Verteilung der vier Basen im Primer ist vorzugsweise zufällig, wobei mehr als drei aufeinanderfolgende G oder C am 3'-Ende und mehr als drei aufeinanderfolgende vermieden werdenG oder C lassen sich in der GC-reichen Sequenzregion leicht paaren.
6. Der Primer-Designbereich sollte komplexe Sekundärstrukturen vermeiden.
Die durch den Einzelstrang des Amplifikationsprodukts gebildete Sekundärstruktur beeinflusst den reibungslosen Ablauf der PCR.Versuchen Sie, diese Region beim Design von Primern zu vermeiden, indem Sie im Voraus vorhersagen, ob in der Zielsequenz eine Sekundärstruktur vorhanden ist.
7. Bei den Primern selbst und zwischen den Primern sollte versucht werden, aufeinanderfolgende komplementäre Basen zu vermeiden.
Es kann keine aufeinanderfolgende 4-Basen-Komplementarität zwischen dem Primer selbst und dem Primer geben.Der Primer selbst sollte keine komplementäre Sequenz aufweisen, da er sich sonst zu einer Haarnadelstruktur faltet, was sich auf die Annealing-Kombination von Primer und Matrize auswirkt.
Komplementäre Sequenzen können zwischen Upstream- und Downstream-Primern nicht existieren.Durch die Komplementarität zwischen Primern entstehen Primerdimere, die die PCR-Effizienz verringern und sogar die quantitative Genauigkeit beeinträchtigen.Wenn die Primer-Dimer- und Haarnadelstrukturen unvermeidbar sind, sollte der △G-Wert nicht zu hoch sein (sollte unter 4,5 kcal/mol liegen).
8. Die Primer verstärken das zielspezifische Produkt.
Das ultimative Ziel des qPCR-Nachweises besteht darin, die Häufigkeit des Zielgens zu verstehen.Wenn eine unspezifische Amplifikation auftritt, ist die Quantifizierung ungenau.Daher müssen die Primer nach dem Entwurf von BLAST getestet und die Spezifität der Produkte in der Sequenzdatenbank verglichen werden.
Als nächstes nehmen wir das menschliche GAS6-Gen (Growth Arrest Specific 6) als Beispiel, um qPCR-Primer zu entwerfen.
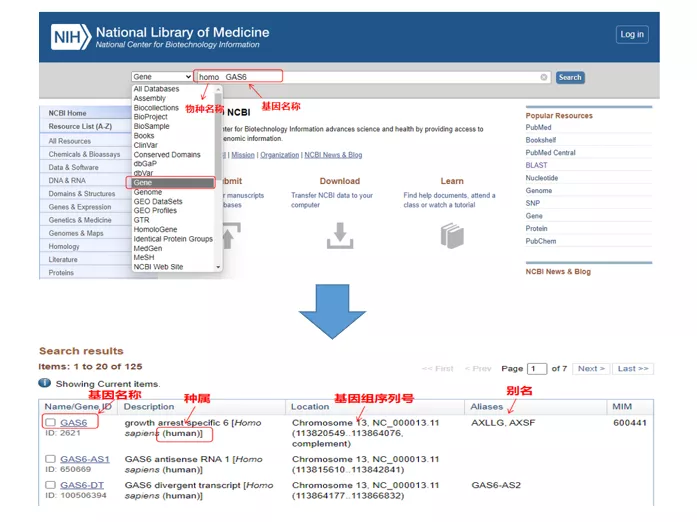
01 Abfragegen
Homo GAS6über NCBI.Hier sollten wir darauf achten, den Gennamen und die Art zu vergleichen, um sicherzustellen, dass sie konsistent sind.
 02 Finden Sie die Gensequenz
02 Finden Sie die Gensequenz
(1) Wenn es sich bei der Zielsequenz um genomische DNA handelt, wählen Sie die erste aus, bei der es sich um die genomische DNA-Sequenz des Gens handelt.
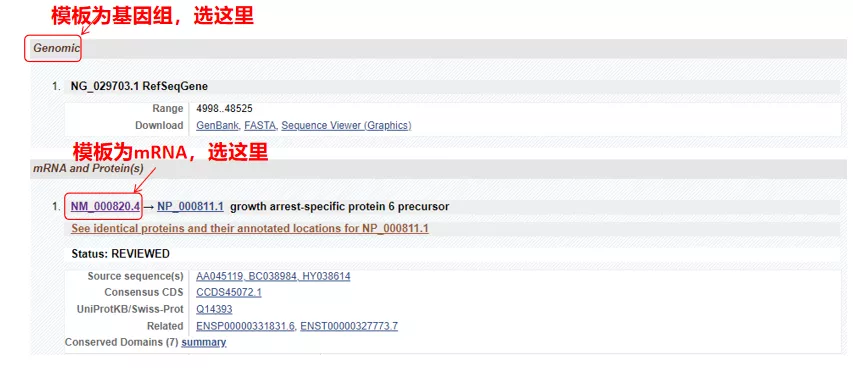 (2) Wenn die Zielsequenz mRNA ist, wählen Sie die zweite aus.Klicken Sie nach der Eingabe in der Tabelle unten auf „CDS“.Die braune Hintergrundsequenz ist die kodierende Sequenz des Gens.
(2) Wenn die Zielsequenz mRNA ist, wählen Sie die zweite aus.Klicken Sie nach der Eingabe in der Tabelle unten auf „CDS“.Die braune Hintergrundsequenz ist die kodierende Sequenz des Gens.
 03 Designgrundierungen
03 Designgrundierungen
Geben Sie die Primer-BLAST-Schnittstelle ein
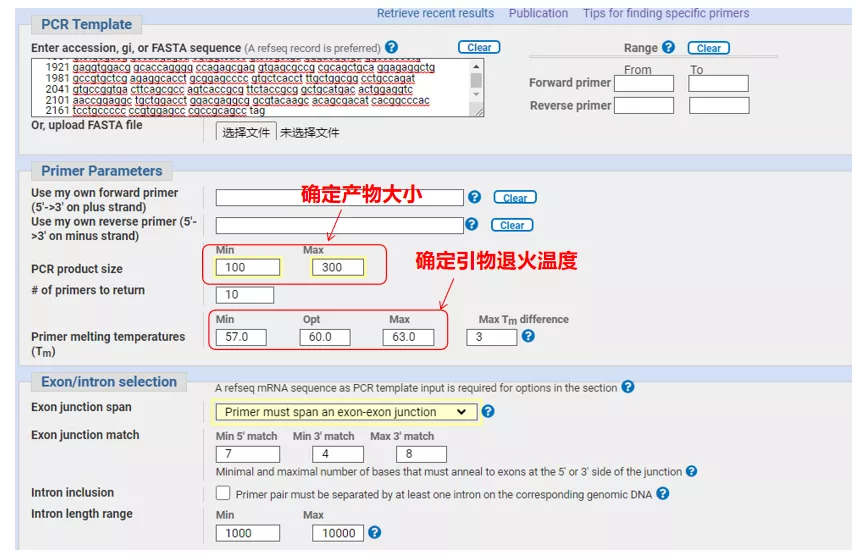
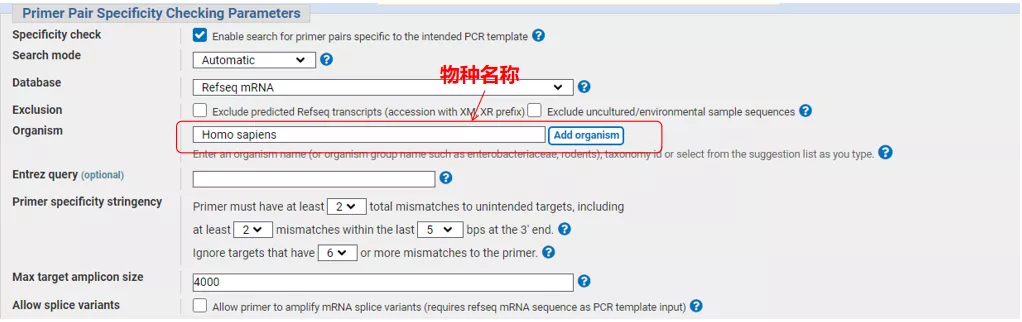
 Geben Sie oben links die Gensequenznummer oder die Sequenz im Fasta-Format ein und geben Sie die relevanten Parameter ein.
Geben Sie oben links die Gensequenznummer oder die Sequenz im Fasta-Format ein und geben Sie die relevanten Parameter ein.
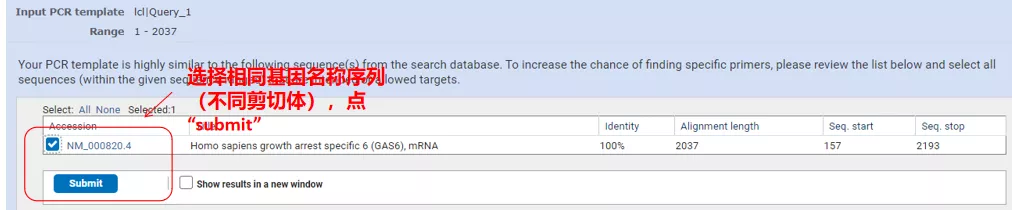
Klicken Sie auf „Primer abrufen“ und NCBI wird angezeigt, um Ihnen mitzuteilen, dass diese Parameterauswahl auf andere Spleißvarianten ausgeweitet wird.Wir können die verschiedenen Spleißvarianten überprüfen und sie einreichen, um das entsprechende Primerpaar zu erhalten (wie in der Abbildung unten gezeigt).Die Ausführung dieses Vorgangs kann mehrere zehn Sekunden dauern.
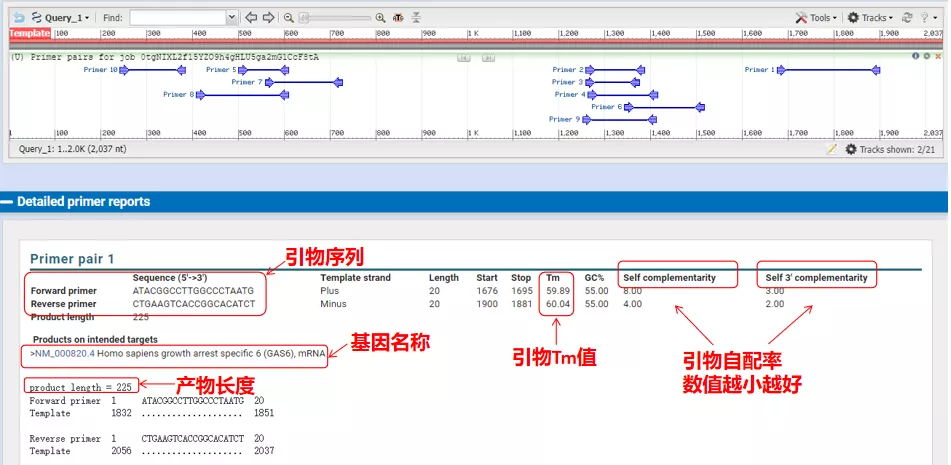 Die Annealing-Temperaturen dieser Primerpaare liegen alle bei etwa 60 °C.Wählen Sie je nach Zweck des Experiments Primer mit mäßiger Länge, guter Spezifität und geringer Selbstkomplementierung der Primer für das Experiment, und die Erfolgsquote ist recht hoch!
Die Annealing-Temperaturen dieser Primerpaare liegen alle bei etwa 60 °C.Wählen Sie je nach Zweck des Experiments Primer mit mäßiger Länge, guter Spezifität und geringer Selbstkomplementierung der Primer für das Experiment, und die Erfolgsquote ist recht hoch!
04Überprüfung der Primerspezifität
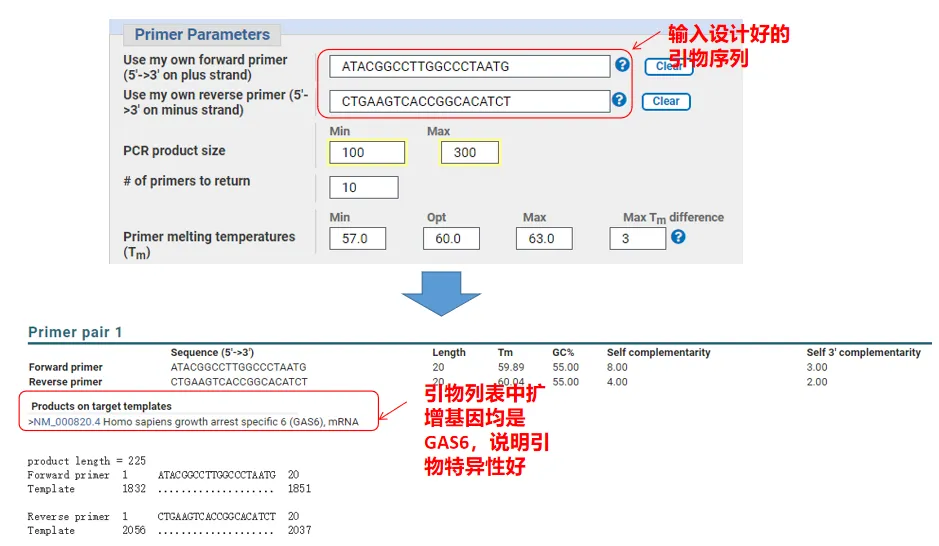
Tatsächlich kann Primer-Blast nicht nur Primer entwerfen, sondern auch die von uns selbst entwickelten Primer bewerten.Kehren Sie zur Primer-Designseite zurück, geben Sie die von uns entworfenen Upstream- und Downstream-Primer ein. Andere Parameter werden nicht angepasst.Nach dem Absenden können Sie sehen, ob das Primerpaar auch auf anderen Genen vorhanden ist.Wenn sie alle auf dem Gen angezeigt werden, das wir amplifizieren möchten, ist die Spezifität dieses Primerpaars großartig!(Dies ist zum Beispiel das einzige Ergebnis der Primer-Abfrage!)
05 Beurteilung der Primerqualität
Welche Art von Primer ist der „perfekte“ Primer, der „Verstärkungseffizienz bis zum Standard“, „verstärkte Produkteigenschaften“ und „zuverlässige experimentelle Ergebnisse“ kombiniert?
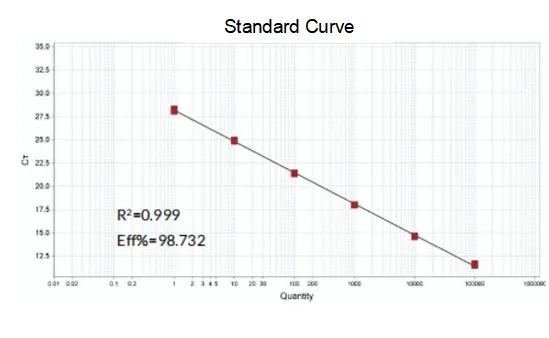 Verstärkungseffizienz
Verstärkungseffizienz
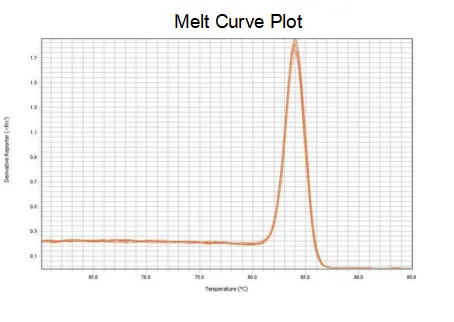 Schmelzkurve
Schmelzkurve
Die Amplifikationseffizienz der Primer erreicht 90–110 %, was bedeutet, dass die Amplifikationseffizienz gut ist, und die Schmelzkurve weist einen einzelnen Peak und normalerweise Tm>80 °C auf, was bedeutet, dass die Amplifikationsspezifität gut ist.
Verwandte Produkte:
Echtzeit-PCR Easy – SYBR GREEN I
Echtzeit-PCR Easy-Taqman
Zeitpunkt der Veröffentlichung: 10. Februar 2023








