Das RT-qPCR-Experiment umfasst drei Schritte der RNA-Extraktion und -Qualitätsbewertung, der reversen Transkription und der qPCR. Jeder Schritt weist viele Vorsichtsmaßnahmen auf, die wir im Folgenden ausführlich vorstellen.
Ⅰ.Bewertung der RNA-Qualität
Im RT-qPCR-Experiment muss nach Abschluss der RNA-Extraktion die Qualität der RNA bewertet werden, und das Folgeexperiment kann erst durchgeführt werden, nachdem es qualifiziert ist.Zu den Bewertungsmethoden gehören Spektrophotometer, Agilent-Gelelektrophorese und Agilent 2100-Analyse, darunter das am häufigsten verwendete Spektrophotometer und die Agarose-Gelelektrophorese-Methode zur Erkennung.Es ist zu beachten, dass diese beiden Methoden zusammen verwendet werden müssen, um den Nachweis und die Analyse der RNA-Konzentration, -Reinheit und -Integrität abzuschließen und so die Qualität der RNA sicherzustellen.
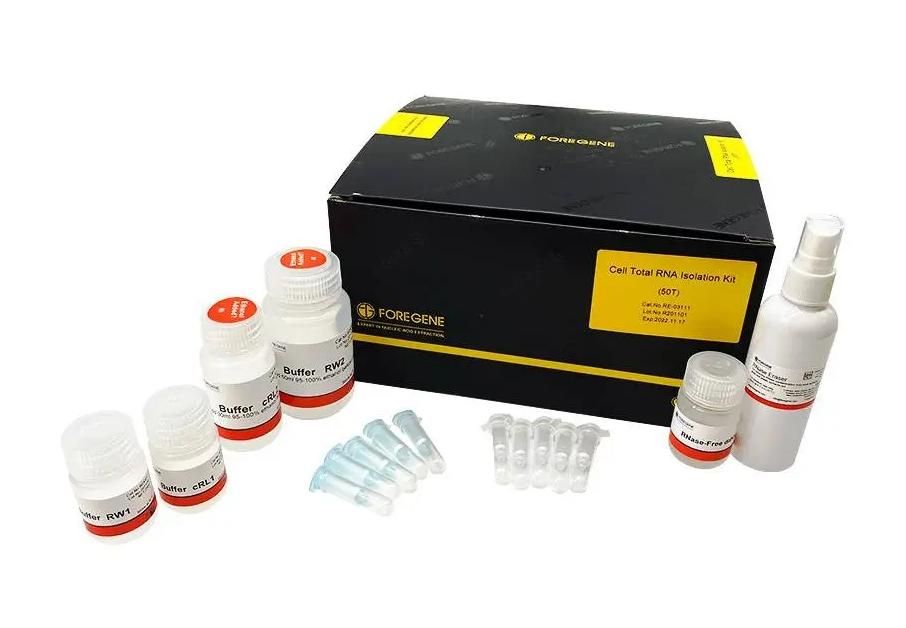
Zugehöriges RNA-Isolierungskit:
Zell-Gesamt-RNA-Isolierungskit
Aus verschiedenen kultivierten Zellen kann in 11 Minuten hochreine und hochwertige Gesamt-RNA gewonnen werden.
Tier-Gesamt-RNA-Isolierungskit
Extrahieren Sie schnell und effizient hochreine und hochwertige Gesamt-RNA aus verschiedenen tierischen Geweben.
Spektrophotometer:
Das Spektrophotometer wird hauptsächlich zur Bestimmung der Konzentration und Reinheit von RNA verwendet, kann jedoch nicht die Integrität von RNA und genomischen Rückständen erkennen.Unter diesen sind A260/280 und A260/230 wichtige Parameter für den Nachweis der RNA-Reinheit, und die RNA-Reinheit kann anhand der Schwankung ihrer Werte nachgewiesen werden:
1. 1,9< A260/280< 2,1, was darauf hinweist, dass die RNA-Reinheit gut ist;A260/280<1,9, was darauf hinweist, dass möglicherweise Proteinreste in der RNA vorhanden sind;A260/280 > 2,1, was auf einen möglichen teilweisen Abbau der RNA hinweist, der durch Agarosegelelektrophorese weiter bestätigt werden kann.
2. 2,0< A260/230< 2,2, was darauf hinweist, dass die RNA-Reinheit gut ist;A260/230< 2,0, was darauf hinweist, dass in der RNA möglicherweise Rückstände organischer Reagenzien wie Phenole, Ethanol oder Zucker vorhanden sind.
Agarose-Gelelektrophorese:
Der Agarose-Gelelektrophorese-Assay kann die RNA-Integrität, das Genom und Proteinrückstände analysieren, kann jedoch die RNA-Konzentration nicht genau quantifizieren oder die Rückstände organischer Reagenzien nachweisen.Nehmen wir zum Beispiel eukaryotische RNA-Vorlagen:
1. Die RNA wurde einer Agarosegelelektrophorese unterzogen.Wenn auf der Gelkarte nur drei einzelne Banden von 28sRNA, 18sRNA und 5,8sRNA vorhanden waren, deutet dies darauf hin, dass die extrahierte RNA intakt ist.Wenn ein Dragging-Phänomen auftritt, weist dies auf einen teilweisen Abbau der RNA hin.
2. Wenn sich zwischen dem Klebeloch und der 28sRNA-Bande eine einzelne helle Bande befindet, sind möglicherweise genomische DNA-Reste vorhanden.
3. Wenn im Klebeloch Streifen erscheinen, deutet dies darauf hin, dass sich möglicherweise Rückstände von Proteinen und anderen makromolekularen Substanzen befinden.
Ⅱ. Reverse Transkription
Nachdem die RNA-Extraktion abgeschlossen ist, muss sie für nachfolgende Experimente in cDNA umgewandelt werden, daher ist der Umkehrschritt unerlässlich.Die Reverse Transkription wird aus der Auswahl der Reverse Transkriptase und des Primers eingeführt:
Auswahl der Reverse-Transkriptase:
Zu den typischen Reverse Transkriptasen gehören AMV-RTase und MMLV-RTase.Die RNase H von AMV RTase hat eine starke Aktivität, eine kurze Syntheselänge, eine geringe Synthesemenge und eine gute thermische Stabilität (42 ~ 55℃).Die RNase-H-Aktivität der MMLV-RTase ist schwach, die Syntheselänge ist lang, die Synthesemenge ist hoch und die thermische Stabilität ist schlecht (37 ~ 42℃).
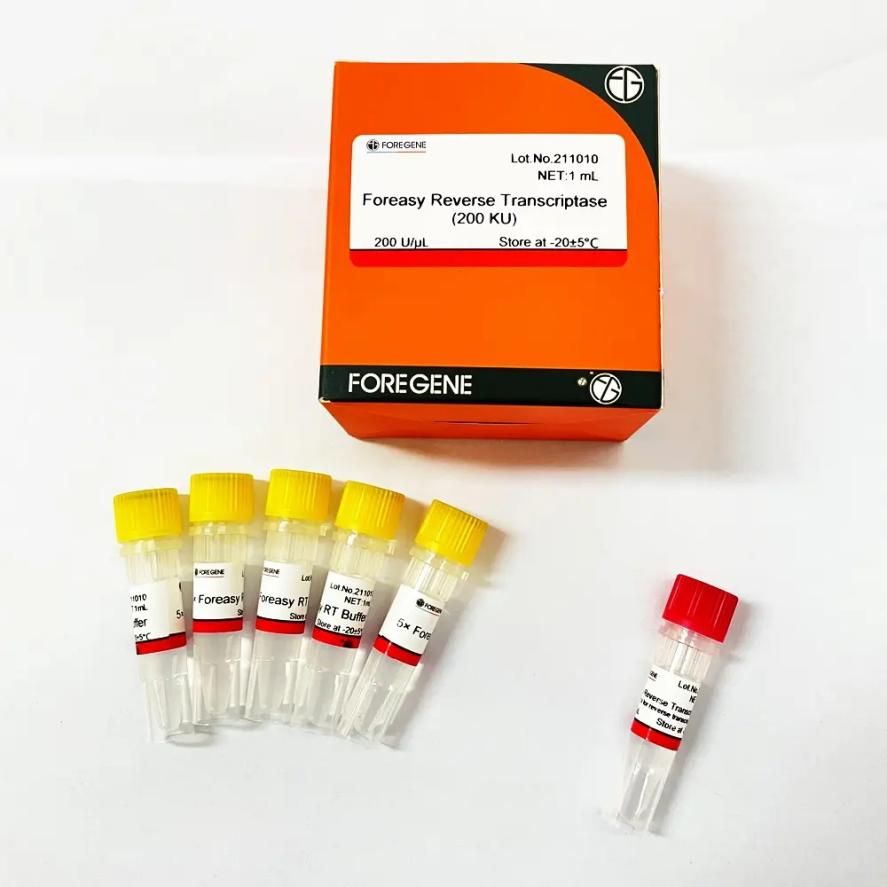
Da das RNase H-Enzym die Funktion hat, RNA-Template abzubauen, sollte MMLV mit schwacher RNase H-Aktivität während der Reverse Transkription bevorzugt ausgewählt werden, und nach späterer Gentechnik hat die thermische Stabilität von MMLV einen qualitativen Sprung erreicht.Einnahme von ForegeneForeasy Reverse Transkriptase (M-MLV für Reverse Transkription) Es handelt sich beispielsweise um eine neue Reverse Transkriptase, die in E. coli-Bakterien mithilfe genetischer Rekombinationstechnologie exprimiert wird.Es handelt sich um eine rekombinante DNA-Polymerase, die einen komplementären DNA-Strang aus einzelsträngiger RNA, DNA oder einem RNA:DNA-Hybrid synthetisiert.Es hat keine RNase H-Aktivität, starke Stabilität, starke RNA-Affinität und hohe Nachweisempfindlichkeit.
Foreasy Reverse Transkriptase (M-MLV für Reverse Transkription)
Auswahl der Grundierung:
Im Allgemeinen lassen sich RT-Primer in drei Kategorien einteilen: Oligo-dT, Zufallsprimer und genspezifische Primer.Wählen Sie je nach experimentellen Anforderungen geeignete Primer zur Verwendung aus.
1. Wenn die Vorlage eukaryotischen Ursprungs ist und die späte cDNA für die routinemäßige PCR-Amplifikation verwendet wird, wird Oligo (dT) empfohlen;Wenn das nachfolgende Experiment nur für qPCR verwendet wird, wird empfohlen, Oligo (dT) mit Zufallsprimern zu mischen, um die Effizienz der reversen Transkription zu verbessern.
2. Wenn die Vorlage von Prokaryoten stammt, sollten Zufallsprimer oder genspezifische Primer für die reverse Transkription ausgewählt werden.
Ⅲ.qPCR
Die Fluoreszenzquantifizierung wird hauptsächlich durch die Auswahl quantitativer Methoden, Primer-Designprinzipien, ROX-Auswahl, Konfiguration des Reaktionssystems und Einstellung der Reaktionsbedingungen usw. erarbeitet.
Auswahl quantitativer Methoden:
Quantitative Methoden werden in relative quantitative Methoden und absolute quantitative Methoden unterteilt.Die relative Quantifizierung kann verwendet werden, um die Wirkung bestimmter Behandlungsmethoden auf die Genexpression zu erkennen, den Unterschied in der Genexpression zu verschiedenen Zeiten zu erkennen und den Unterschied in der Genexpression in verschiedenen Geweben zu vergleichen.Durch absolute Quantifizierung kann die Menge an Nukleinsäure im Virus usw. nachgewiesen werden.Bei der Durchführung von Experimenten müssen wir die geeigneten quantitativen Methoden entsprechend unseren eigenen Experimenten auswählen.
Prinzipien des Primer-Designs:
Das Design des Primers für qPCR steht in direktem Zusammenhang mit der Amplifikationseffizienz und Produktspezifität.Daher ist das richtige Design guter Primer der erste Schritt einer erfolgreichen qPCR.Bei der Gestaltung von Grundierungen sollten die folgenden Grundsätze beachtet werden, um dem Prinzip der herkömmlichen Grundierungsgestaltung gerecht zu werden:
1. Die Länge des Zielfragments wird zwischen 100 und 300 bp kontrolliert;
2. Cross-Exon-Design zur Vermeidung des Einflusses genomischer DNA;
3. Die entworfenen Primer müssen auf ihre Amplifikationseffizienz getestet werden. Erst wenn die Amplifikationseffizienz den Standard (90–110 %) erreicht, können sie für quantitative Experimente verwendet werden.
4. Die Primerkonzentration wird normalerweise zwischen 0,1 uM und 1,0 uM optimiert.
Auswahl vonROX:
Im Prozess der quantitativen Reaktion kann ROX den durch Verdunstung und Kondensation verursachten optischen Wegunterschied, Pipettierfehler oder Volumenunterschied gleichmäßig anpassen und so die Wiederholbarkeit der Ergebnisse verbessern.Allerdings ist zu beachten, dass die Auswahl von ROX instrumentenabhängig ist.Wenn das qPCR-Gerät über die Funktion verfügt, den Unterschied zwischen Löchern automatisch zu korrigieren, muss kein ROX hinzugefügt werden;Andernfalls muss eine ROX-Korrektur hinzugefügt werden.Kleine Partner müssen beim Kauf von Reagenzien entsprechend dem verwendeten Instrument den richtigen ROX auswählen, um spätere Fehler zu vermeiden.
Vorbereitung des Reaktionssystems:
Reaktionsvolumina von 20 µl und 50 µl werden bevorzugt.Bei der Formulierung des Systems sollten folgende Punkte beachtet werden:
1. Das Reaktionssystem muss durch Belüftung in der ultrareinen Werkbank, neue ddH, vorbereitet werden2O wird für jedes Experiment verwendet;
2. Jedes Experiment muss NTC vorbereiten, um zu überprüfen, ob das System verschmutzt ist, und jedes Primerpaar muss bei der Vorbereitung des Systems NTC durchführen.
3. Um festzustellen, ob in der RNA-Matrize gDNA-Reste vorhanden sind, kann für jede Probe eine NRT zum Nachweis vorbereitet werden.
4. Bei der Vorbereitung des Systems wird empfohlen, mindestens 3 technische Wiederholungen für eine Probe durchzuführen;
5. Wenn es sich bei der Vorlage um cDNA handelt, wird eine 5- bis 10-fache Verdünnung empfohlen, um die Hemmwirkung des Reverse-Transkriptions-Systems auf das qPCR-Experiment zu verringern.Es ist besser, die Vorlagenmenge anhand des Gradienten zu untersuchen, sodass der CT-Wert zwischen 20 und 30 liegt.
6. Bestimmen Sie die erforderliche Anzahl an Reaktionen, erhöhen Sie sie um 5–10 % auf der Grundlage der Anzahl an Reaktionen und berechnen Sie die Volumenkonfigurationszahl.
7, das System wird nach dem Vormischprinzip vorbereitet, wobei nach dem Zentrifugieren gemischt wird und sichergestellt wird, dass keine Blasen entstehen;
8. Soweit möglich, unterstützende Verbrauchsmaterialien auszuwählen.
Zugehöriges RT-qPCR-Kit
Das Kit verwendet ein einzigartiges Foregene-Reverse-Transkriptionsreagenz und Foregene HotStar Taq DNA-Polymerase in Kombination mit einem einzigartigen Reaktionssystem, um die Amplifikationseffizienz und Spezifität der Reaktion effektiv zu verbessern.
Zeitpunkt der Veröffentlichung: 23. April 2023