Spezifität der Erkennung
In den meisten Fällen besteht der Zweck des Primerdesigns darin, die Spezifität der PCR zu maximieren.Dies wird durch den mehr oder weniger vorhersehbaren Einfluss vieler Variablen bestimmt.Eine wichtige Variable ist die Sequenz am 3′-Ende des Primers.
Wichtig ist, dass PCR-Assays, die auf Spezifität ausgelegt sind, mit größerer Wahrscheinlichkeit eine hohe Effizienz über einen weiten dynamischen Bereich beibehalten, da der Assay keine unspezifischen Amplifikationsprodukte produziert und dadurch mit PCR-Reagenzien konkurriert oder die Hauptamplifikationsreaktion hemmt.
Natürlich ist die Spezifität in manchen Fällen nicht das Wichtigste, beispielsweise wenn das Ziel darin besteht, eng verwandte, aber unterschiedliche Krankheitserreger zu quantifizieren, sind spezielle Design-, Optimierungs- und Verifizierungsstandards erforderlich.
Die Schmelzkurve ist eine Standardmethode zur Beurteilung der Spezifität von Amplikons, zumindest im Hinblick darauf, ob ein einzelnes Ziel amplifiziert werden soll.Es muss jedoch betont werden, dass die Schmelzkurven irreführend sein können, da sie beispielsweise durch die kombinierten Effekte von suboptimalen Primern und niedrigen Templatkonzentrationen beeinflusst werden können.
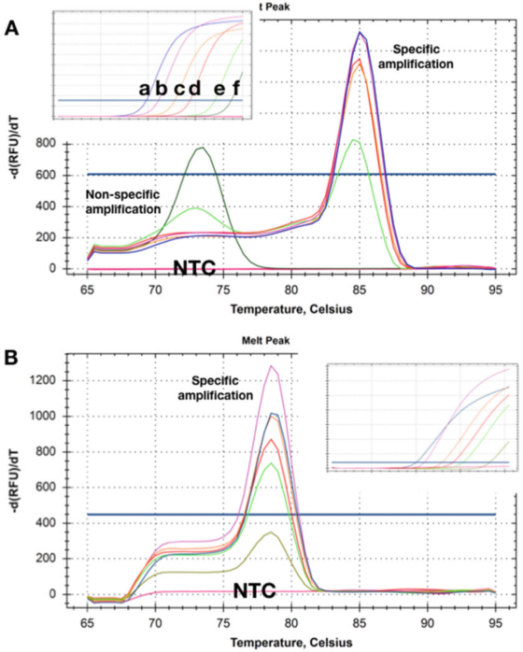
P5 |Die Schmelzkurve zeigt die Tm-Verschiebungen, die aus zwei Nachweisen unterschiedlicher Mengen zweier Ziel-DNAs erhalten wurden.
A. Bei höheren Konzentrationen (ad)) gibt es nach Abschluss der qPCR-Messung kein offensichtliches Primer-Dimer.Wenn die Matrizenkonzentration auf 50 Kopien sinkt (e), beginnt ein unspezifisches Produkt zu erscheinen und wird zum einzigen Produkt mit der niedrigsten Konzentration (f).
B. Der Test zeichnete bei allen Zielkonzentrationen die gleichen Tms auf und selbst bei der niedrigsten Konzentration (5 Kopien) gab es kein offensichtliches Primer-Dimer.Bei Verwendung dieser beiden Nachweismethoden wurden in NTCs keine Amplifikationsprodukte nachgewiesen.
P5 zeigt die Auflösungskurven, die mit Proben erhalten wurden, in denen das Templat in unterschiedlichen Konzentrationen vorhanden ist.P 5a zeigt, dass bei den beiden niedrigsten Konzentrationen die Tms der erzeugten unspezifischen Amplifikationsprodukte niedriger sind als die der spezifischen Amplifikate.
Offensichtlich kann diese Nachweismethode nicht zuverlässig zur Erkennung von Zielen verwendet werden, die in geringen Konzentrationen vorhanden sind.
Interessanterweise zeichneten NTCs, also Proben ohne DNA, keine (unspezifischen) Amplifikationsprodukte auf, was darauf hindeutet, dass genomische Hintergrund-DNA an der unspezifischen Amplifikation/Polymerisation beteiligt sein kann.
Manchmal können solche Hintergrundprimer und unspezifische Amplifikation nicht behoben werden, aber es ist oft möglich, eine Nachweismethode zu entwerfen, die keine unspezifische Amplifikation bei jeder Template-Konzentration und NTC aufweist (P 5b).
Hier führt selbst die Aufzeichnung der Verstärkung der Zielkonzentration mit einem Cq von 35 zu einer spezifischen Auflösungskurve.Ebenso zeigten NTCs keine Anzeichen einer unspezifischen Amplifikation.Manchmal hängt das Nachweisverhalten von der Mutterlauge ab und in bestimmten Pufferzusammensetzungen wird nur eine unspezifische Amplifikation nachgewiesen, die mit unterschiedlichen Mg2+-Konzentrationen zusammenhängen kann.
Stabilität der Erkennung
Die Optimierung von Ta ist ein nützlicher Schritt im empirischen Verifizierungs- und Optimierungsprozess der qPCR-Erkennung.Es liefert einen direkten Hinweis auf die Robustheit des Primersatzes, indem es die Temperatur (oder den Temperaturbereich) anzeigt, die den niedrigsten Cq erzeugt, ohne den NTC zu verstärken.
Der zwei- bis vierfache Unterschied in der Empfindlichkeit mag für Menschen mit hoher mRNA-Expression nicht wichtig sein, kann aber bei diagnostischen Tests den Unterschied zwischen positiven und falsch negativen Ergebnissen ausmachen.
Die Ta-Eigenschaften von qPCR-Primern können stark variieren.Einige Tests sind nicht sehr robust, und wenn sie nicht unter dem optimalen Ta-Wert der Primer durchgeführt werden, brechen sie schnell zusammen.
Dies ist wichtig, da diese Art des Nachweises in der Praxis häufig problematisch ist und die Reinheit der Probe, die DNA-Konzentration oder das Vorhandensein anderer DNA möglicherweise nicht optimal sind.
Darüber hinaus kann die Zielkopienzahl in einem weiten Bereich variieren und die Reagenzien, Plastikutensilien oder Instrumente können sich von denen unterscheiden, die bei der Testvorbereitung verwendet wurden.
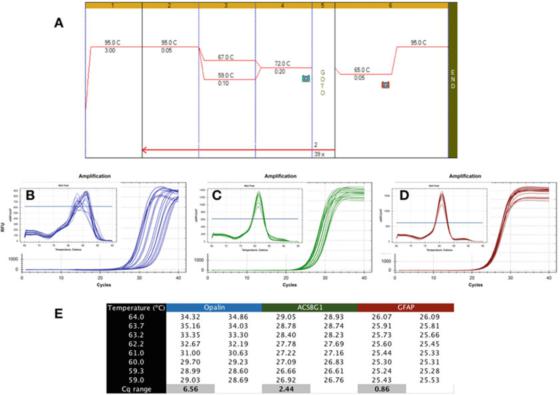
P6|Der Temperaturgradient zeigt die unterschiedliche Robustheit der PCR-Detektion.
A. Verwenden Sie den Sensifast SYBR-Mastermix von Bioline (Katalognummer BIO-98050), um eine PCR mit cDNA durchzuführen, die aus menschlicher Gehirn-RNA hergestellt wurde.
B. Verwenden Sie das CFX qPCR-Gerät von Bio-Rad, um die Amplifikationskarte und Auflösungskurve von Apalen aufzuzeichnen (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Amplifikationsdiagramm und Schmelzkurve von ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Amplifikationsdiagramm und Auflösungskurve von GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs, die bei unterschiedlichen Glühtemperaturen aufgezeichnet wurden, zeigen den Unterschied im Cq, der bei einem Temperaturgradienten von 7 °C aufgezeichnet wurde.
P 6 zeigt ein typisches Ergebnis eines unerwünschten Tests, bei dem qPCR mit einem Tas-Gradienten zwischen 59 °C und 67 °C (P 6a) durchgeführt wurde und Primer für drei menschliche hirnspezifische Gene verwendet wurden.
Aus dem Amplifikationsdiagramm ist ersichtlich, dass Opalin-Primer alles andere als ideal sind, da ihr optimaler Ta-Bereich sehr eng ist (Abbildung 6b), d.
Diese Nachweismethode ist instabil und kann zu einer suboptimalen Verstärkung führen.Daher sollte dieses Primerpaar neu gestaltet werden.Darüber hinaus zeigt die Schmelzkurvenanalyse (Einschub), dass die Spezifität dieser Nachweismethode ebenfalls problematisch sein kann, da die Schmelzkurve jedes Ta unterschiedlich ist.
Die in P 6c gezeigte ACSBG1-Nachweismethode ist robuster als die oben genannte Opalin-Nachweismethode, aber sie ist noch lange nicht ideal und kann wahrscheinlich verbessert werden.
Wir betonen jedoch, dass kein notwendiger Zusammenhang zwischen Robustheit und Spezifität besteht, da die mit dieser Nachweismethode erstellte Auflösungskurve in allen Tas den gleichen Spitzenwert zeigt (Einschub).
Andererseits ist der Robustheitstest viel toleranter und liefert ähnliche Cqs in einem weiten Bereich von Tas, wie im GFAP-Test, der in P 6d gezeigt wird.
Der im gleichen 8-Grad-Celsius-Bereich erhaltene Unterschied in den Cqs beträgt weniger als 1, und die Auflösungskurve (Einschub) bestätigt die Nachweiseigenschaften in diesem Temperaturbereich.Es ist zu beachten, dass der berechnete Tas-Bereich und der tatsächliche Ta-Bereich sehr unterschiedlich sein können.
Es gibt viele Richtlinien, die Forschern bei der Entwicklung effizienter Primer helfen sollen. Die meisten davon basieren auf seit langem etablierten Regeln und dem 3′-Ende der Primer wurde große Aufmerksamkeit geschenkt.Es wird oft empfohlen, ein G oder C am 3'-Ende und zwei G- oder C-Basen (GC-Klemme) einzubauen, jedoch nicht mehr als zwei der letzten 5 Bases.
In der Praxis können diese Regeln den Forschern Orientierung geben, sie sind jedoch nicht unbedingt unter allen Umständen korrekt.
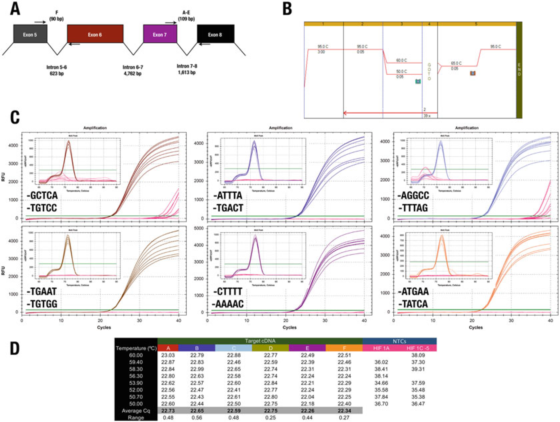
P7 |Das 3'-Ende des Primers hat kaum Einfluss auf die Spezifität oder Effizienz.
A. Die Position der Primer für das menschliche HIF-1α-Gen (NM_181054.2).
B. Verwenden Sie Agilent Brilliant III SYBR Green Mutterlauge (Kat.-Nr. 600882), um sechs Testobjekte zu verstärken.
C. Amplifikationsdiagramm und Schmelzkurve, aufgezeichnet mit dem CFX qPCR-Instrument und 3′-End-Primern von Bio-Rad.NTCs werden rot dargestellt.
D. CQS-Aufzeichnung jedes Testelements
Beispielsweise widerspricht das Ergebnis in P 7 der 3′-Ende-Regel.Alle Designs liefern grundsätzlich die gleichen Ergebnisse, wobei nur zwei Primerkombinationen zu einer unspezifischen Amplifikation im NTC führen.
Allerdings können wir die Wirkung des GC-Clips nicht unterstützen, da in diesem Fall die Verwendung von A oder T als maximal 30 Basen die Spezifität nicht verringert.
Test C, bei dem der F-Primer in GGCC endet, zeichnete Cqs in NTCs auf, was darauf hindeutet, dass man diese Sequenzen am 30-Ende möglicherweise vermeiden möchte.Wir betonen, dass die einzige Möglichkeit, die beste 3′-Endsequenz eines Primerpaars zu bestimmen, darin besteht, einige Kandidatenprimer experimentell zu bewerten.
Verstärkungseffizienz
Wichtig ist, dass zwar ein unspezifischer PCR-Nachweis niemals spezifisch werden kann, die Amplifikationseffizienz jedoch auf viele verschiedene Arten angepasst und maximiert werden kann, indem das Enzym, die Mutterlauge, die Zusatzstoffe und die Zyklusbedingungen geändert werden.
Um die Effizienz des PCR-Nachweises zu bewerten, ist es am besten, eine Reihenverdünnung des 10- oder 5-fachen der Zielnukleinsäure zu verwenden, also die „Standardkurvenmethode“.
Wenn PCR-Amplikons oder synthetische DNA-Ziele zur Erstellung einer Standardkurve verwendet werden, sollten serielle Verdünnungen dieser Ziele mit einer konstanten Menge Hintergrund-DNA (z. B. genomischer DNA) gemischt werden.
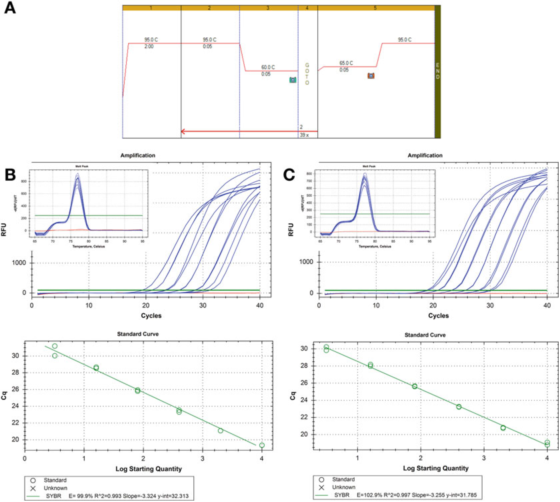
P8 |Verdünnungskurve zur Bewertung der Effizienz der PCR.
A. Verwenden Sie Primer für HIF-1: F: AAGAACTTTTAGGCCGCTCA und R: TGTCCTGTGGTGACTTGTCC und Agilents Brilliant III SYBR Green Mastermix (Katalognummer 600882) für PCR- und Schmelzkurvenbedingungen.
B. 100 ng RNA wurden revers transkribiert, zweifach verdünnt und seriell verdünnte cDNA-Proben wurden fünffach verdünnt, um 1 ng menschliche genomische DNA zu erhalten.Die Schmelzkurve ist im Einschub dargestellt.
C. Die RT-Reaktion, Verdünnung und Reihenverdünnung wurden für die zweite cDNA-Probe wiederholt und die Ergebnisse waren ähnlich.
P 8 zeigt zwei Standardkurven, bei denen dieselbe Nachweismethode an zwei verschiedenen cDNA-Proben verwendet wurde. Das Ergebnis ist die gleiche Effizienz, etwa 100 %, und der R2-Wert ist ebenfalls ähnlich, dh der Grad der Übereinstimmung zwischen den experimentellen Daten und der Regressionslinie oder der Daten Grad der Linearität.
Die beiden Standardkurven sind vergleichbar, aber nicht genau gleich.Wenn der Zweck darin besteht, das Ziel genau zu quantifizieren, muss beachtet werden, dass es nicht akzeptabel ist, eine Berechnung der Kopienzahl bereitzustellen, ohne die Unsicherheit zu erklären
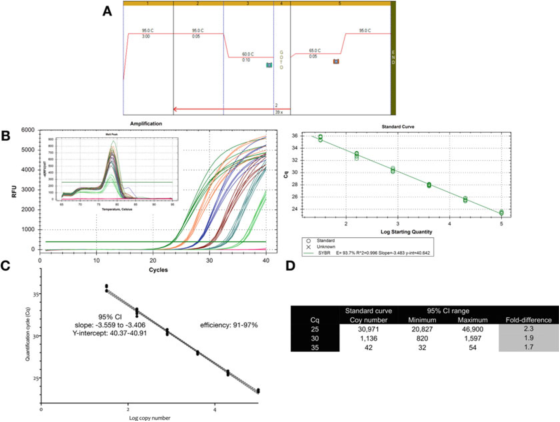
P9 |Messunsicherheit im Zusammenhang mit der Quantifizierung mithilfe einer Standardkurve.
A. Verwenden Sie Primer für GAPDH (NM_002046), um PCR- und Schmelzkurvenbedingungen durchzuführen.F: ACAGTTGCCATGTAGACC und R: TAACTGGTTGAGCACAGG und Biolines Sensifast SYBR-Mastermix (Katalognummer BIO-98050).
B. Amplifikationsdiagramm, Schmelzkurve und Standardkurve, aufgezeichnet mit dem CFX qPCR-Gerät von Bio-Rad.
C. Standardkurvendiagramm und 95 %-Konfidenzintervall (KI).
D. Die Kopienzahl und das 95 %-Konfidenzintervall der drei aus der Verdünnungskurve abgeleiteten Cq-Werte.
P 9 zeigt, dass für einen optimierten Test die inhärente Variabilität einer einzelnen Standardkurve ungefähr das Zweifache beträgt (95 %-Konfidenzintervall, Minimum bis Maximum), was möglicherweise die kleinste zu erwartende Variabilität ist.
Ähnliches Produkt:
Zeitpunkt der Veröffentlichung: 30. September 2021