Großer Rabatt China High Sensitivity One-Step Probe Rt-Qpcr Kit V2
Wir sind erfahrener Hersteller.Gewinn der Mehrheit bei den entscheidenden Zertifizierungen seines Marktes für China High Sensitivity One-Step Probe Rt mit großem Rabatt.QpcrKit V2, Qualität ist das Leben in der Fabrik, die Konzentration auf die Nachfrage der Kunden ist die Quelle des Überlebens und der Entwicklung des Unternehmens. Wir halten an Ehrlichkeit und gutem Glauben fest und freuen uns auf Ihr Kommen!
Wir sind erfahrener Hersteller.Gewinnen Sie die Mehrheit bei den entscheidenden Zertifizierungen seines MarktesChina Taq DNA-Polymerase, Qpcr, Unser Unternehmen verspricht: angemessene Preise, kurze Produktionszeiten und einen zufriedenstellenden Kundendienst. Wir heißen Sie auch jederzeit willkommen, unsere Fabrik zu besuchen.Ich wünschte, wir hätten jetzt ein angenehmes und langfristiges gemeinsames Geschäft!!!
Beschreibungen
Dieses Kit verwendet ein einzigartiges Lysepuffersystem, das RNA aus kultivierten Zellproben für RT-qPCR-Reaktionen schnell freisetzen kann, wodurch der zeitaufwändige und mühsame RNA-Reinigungsprozess entfällt.Die RNA-Vorlage kann in nur 7 Minuten erhalten werden.Mit den im Kit enthaltenen 5×Direct RT Mix- und 2×Direct qPCR Mix-SYBR-Reagenzien können Sie schnell und effektiv quantitative PCR-Ergebnisse in Echtzeit erhalten.
5×Direct RT Mix und 2×Direct qPCR Mix-SYBR weisen eine starke Inhibitortoleranz auf und das Lysat der Proben kann direkt als Vorlage für RT-qPCR verwendet werden.Dieses Kit enthält die einzigartige hochaffine RNA-Foregene-Reverse-Transkriptase und Hot D-Taq DNA-Polymerase, dNTPs und MgCl2, Reaktionspuffer, PCR-Optimierer und Stabilisator.
Spezifikationen
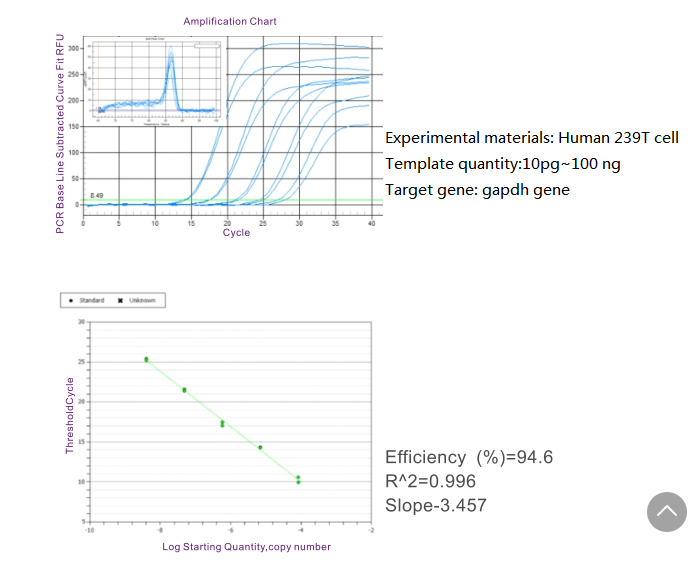
200×20μl Rxns, 1000×20μl Rxns
Kit-Komponenten
| Teil I | Puffer CL |
| Foregene Protease Plus II | |
| Puffer ST | |
| Teil II | DNA-Radierer |
| 5× Direkter RT-Mix | |
| 2× Direkter qPCR-Mix-SYBR | |
| 50× ROX-Referenzfarbstoff | |
| RNase-freies ddH2O | |
| Anweisungen | |
Merkmale und Vorteile
■ Einfach und effektiv: Mit der Cell Direct RT-Technologie können RNA-Proben in nur 7 Minuten gewonnen werden.
■ Der Probenbedarf ist gering, es können nur 10 Zellen getestet werden.
■ Hoher Durchsatz: Es kann RNA in Zellen, die in 384-, 96-, 24-, 12- und 6-Well-Platten kultiviert wurden, schnell nachweisen.
■ DNA Eraser kann freigesetzte Genome schnell entfernen und die Auswirkungen auf nachfolgende experimentelle Ergebnisse erheblich reduzieren.
■ Das optimierte RT- und qPCR-System macht die zweistufige RT-PCR-Reverse-Transkription effizienter, die PCR spezifischer und resistenter gegen RT-qPCR-Reaktionsinhibitoren.
Kit-Anwendung
Anwendungsbereich: kultivierte Zellen.
- Durch Probenlyse freigesetzte RNA: Gilt nur für die RT-qPCR-Vorlage dieses Kits.
- Das Kit kann für folgende Zwecke verwendet werden: Genexpressionsanalyse, Überprüfung des siRNA-vermittelten Gen-Silencing-Effekts, Arzneimittelscreening usw.
Diagramm
Lagerung und Haltbarkeit
Teil I dieses Kits sollte bei 4℃ gelagert werden;Teil II sollte bei -20℃ gelagert werden.
Foregene Protease Plus II sollte bei 4℃ gelagert werden, nicht bei -20℃ einfrieren.
Reagenz 2×Direct qPCR Mix-SYBR sollte bei -20℃ im Dunkeln gelagert werden;Bei häufigem Gebrauch kann es zur kurzfristigen Lagerung auch bei 4℃ gelagert werden (innerhalb von 10 Tagen aufbrauchen). Wir sind erfahrene Hersteller.Gewinnen Sie die Mehrheit bei den entscheidenden Zertifizierungen seines Marktes für China High Sensitivity One-Step Probe Rt-Qpcr Kit V2 mit großem Preisnachlass. Qualität ist das Leben in der Fabrik. Der Fokus auf die Nachfrage der Kunden ist die Quelle des Überlebens und der Entwicklung des Unternehmens.
Großer RabattChina Taq DNA-Polymerase, Qpcr, Unser Unternehmen verspricht: angemessene Preise, kurze Produktionszeit und zufriedenstellenden Kundendienst. Wir heißen Sie auch jederzeit willkommen, unsere Fabrik zu besuchen.Ich wünschte, wir hätten jetzt ein angenehmes und langfristiges gemeinsames Geschäft!!!
QuickEeinfachTM Cell Direct RT-qPCR Kit -Taqman
Kat.Nr.DRT-01021/01022
Für zelldirekte RT-qPCR mit ≤ 1.000.000 Zellen
Produkteinführung
Dieses Produkt verwendet ein einzigartiges Lysepuffersystem, um RNA aus kultivierten Zellproben für RT-qPCR-Reaktionen schnell freizusetzen, wodurch der zeitaufwändige und mühsame RNA-Reinigungsprozess entfällt und die erforderliche RNA-Vorlage in nur 7 Minuten erhalten wird. Mit dem im Kit enthaltenen 5× Direct RT Mix und 2× Direct qPCR Mix-Taqman können Sie schnell und effizient quantitative PCR-Ergebnisse in Echtzeit erhalten.
5× Direct RT Mix und 2× Direct qPCR Mix-Taqman weisen eine starke Inhibitortoleranz auf und können unter Verwendung des Lysats der zu messenden Probe als Vorlage eine effiziente Umkehrung und spezifische Amplifikation durchführen.Das Reagenz enthält Foregene Reverse Transkriptase, Hot D-Taq DNA Polymerase, dNTPs, MgCl2, Reaktionspuffer, PCR-Optimierer und Stabilisator, der mit Lysepuffer zum schnellen und einfachen Nachweis von Proben verwendet werden kann und sich durch hohe Empfindlichkeit, Spezifität und Stabilität auszeichnet.
Produktmerkmale
Einfache, effektive Cell Direct RT-Technologie, die für die Gewinnung von RNA-Proben nur 7 Minuten benötigt.
Der Probenbedarf ist gering und es können mindestens 10 kultivierte Zellen für Experimente verwendet werden.
Hoher Durchsatz für die schnelle RNA-Erfassung kultivierter Zellen wie 384-, 96-, 24-, 12- und 6-Well-Platten.
DNA Eraser ist in der Lage, freigesetzte Genome schnell zu entfernen, wodurch die Auswirkungen auf nachfolgende experimentelle Ergebnisse erheblich reduziert werden.
Optimierte RT- und qPCR-Systeme ermöglichen eine zweistufige RT-PCR mit effizienterer reverser Transkription, Spezifität und stärkerer Toleranz gegenüber RT-qPCR-Reaktionsinhibitoren.
Kit-Anwendung
Anwendungsbereich: Kultivierte Zellen.
Durch Probenlyse interpretierte RNA: Wird nur als zweistufige RT-qPCR-Vorlage verwendet.
Kits können für folgende Zwecke verwendet werden: Analyse der Genregulation, Alleltests, Drogenscreening usw.
Kit-Einschränkungen
Amplifizierte Fragmente ≤ 300 bp.
Kits werden zur Frischkultur von Zellen verwendet.
Produktqualitätskontrolle
Gemäß dem Total Quality Management System von FOREGENE wird jede Charge der Kits der Cell Direct RT-qPCR-Serie mehrmals gründlich getestet, um die Zuverlässigkeit und Stabilität der Qualität jeder Kit-Charge sicherzustellen.
Inhalt des Kits
| QuickEasyTM Cell Direct RT-qPCR Kit-Taqman | ||||
| Kit-Komponenten: 20 μl qPCR-Reaktionssystem | DRT-01021 | DRT-01022 | Notiz | |
| 200 T | 1000 T | |||
| Teil I | Puffer CL | 4 ml | 20 ml |
Zelllyse |
| Foregene Protease Plus II | 80 μl | 400 μl | ||
| Puffer ST | 400 μl | 1 ml × 2 | ||
|
Teil II | DNA-Radierer | 80 μl | 400 μl | |
| 5×Direct RT Mix * | 160 μl | 800 μl | RT | |
| 2× Direktes qPCR Mix-Taqman * | 1 ml × 2 | 1,7 ml × 6 | qPCR | |
| 20×ROX-Referenzfarbstoff | 40 μl | 200 μl | ||
| RNase-freies ddH2O | 1,7 ml | 10 ml | ||
| Bedienungsanleitung | 1 Stück | 1 Stück | ||
*:Zelllyse, 5×Direct RT Mix, 2× Direct qPCR Mix-Taqman können separat erworben werden, Einzelheiten finden Sie in Anhang 1 (SEITE 13).
Lagerbedingungen
1. Versandbedingungen
Der gesamte Prozess des Kühlboxtransports bei niedrigen Temperaturen, um sicherzustellen, dass sich das Kit in einem Zustand von <4 °C befindet.
2. Lagerbedingungen
Lagern Sie Teil I bei 4 °C und Teil II bei -20 °C.
Die Foregene Protease Plus II sollte bei 4 °C gelagert und nicht bei -20 °C eingefroren werden.
Reagenz 2× Direct qPCR Mix-Taqman wird bei -20 °C oder bei 4 °C für den kurzfristigen Gebrauch bei häufiger Verwendung (innerhalb von 10 Tagen) gelagert.
Informationen zu Kit-Komponenten
Puffer CL: Bietet die für Zelllysereaktionen erforderliche Umgebung.
Puffer ST: Terminiert den Wirkstoff im Lysat, um Auswirkungen auf die nachfolgende RT zu vermeiden.
DNA Eraser: DNA-Entferner, der Effekt der Entfernung des Genoms auf nachfolgende Experimente.
5× Direct RT Mix: Enthält Foregene Reverse Transkriptase mit hoher RNA-Affinität, RNase-Inhibitor, dNTPs, Stabilisatoren, Enhancer, Optimierer und Reverse-Transkriptions-Primer für eine optimale Ausrichtung (Random Primer, Oligo(dT)18Grundierung).
Foregene Protease Plus II: Im Zusammenhang mit Lysepuffer werden Zellen lysiert, um Nukleinsäuren freizusetzen.
2× Direct qPCR Mix-Taqman: Dieses Reagenz enthält Hot D-Taq DNA Polymerase, dNTPs, MgCl2, Reaktionspuffer, PCR-Optimierer und Stabilisator.
20× ROX-Referenzfarbstoff: Wird im Allgemeinen auf Echtzeit-PCR-Amplifikationsgeräten von ABI, Stratagene und anderen Unternehmen verwendet und dient dazu, den Unterschied zwischen PCR-Röhrchen und Röhrchen auszugleichen, die durch PCR-Dosierungsfehler verursacht werden.Die für verschiedene Instrumente erforderliche Konzentration des 20-fachen ROX-Referenzfarbstoffs ist unterschiedlich und der Benutzer kann sie entsprechend der empfohlenen Konzentration des Instruments hinzufügen.
RNase-freies ddH2O: RNase-freies sterilisiertes Reinstwasser für zweistufige RT-qPCR-Reaktionen.
Vorsichtsmaßnahmen:(Lesen Sie die Vorsichtsmaßnahmen sorgfältig durch, bevor Sie das Kit verwenden.)
Achten Sie auf die Durchführungsmethode des Experiments, um eine Kreuzkontamination zwischen den Proben zu vermeiden.
Achten Sie auf die Sauberkeit der Versuchsumgebung und der Utensilien, um eine RNase-Kontamination und einen RNA-Abbau zu vermeiden.
Nehmen Sie frische oder gut konservierte Zellproben und verwenden Sie niemals wiederholt eingefrorene und aufgetaute Zellproben.
5× Direkter qPCR-Mix, 2× Direkter qPCR-Mix-Taqman sollte wiederholtes Einfrieren und Auftauen vermeiden, da es sonst die Reverse Transkription und die PCR-Effizienz beeinträchtigt.
VorbereitungVerpflegungVorBetrieb
Lesen Sie unbedingt die Anweisungen sorgfältig durch, bevor Sie dieses Kit verwenden.Das Cell Direct RT-qPCR-Kit ist einfach, bequem und schnell zu bedienen. Die Anweisungen enthalten umfassende Informationen zum gesamten Kit und zur korrekten Verwendung.Bitte bereiten Sie vor der Verwendung die notwendigen experimentellen Materialien und Geräte vor.
Experimentelle Materialien und Ausrüstung
◆ Kulturzellen.
◆ 1,5 ml oder 2 ml, RNase-/DNase-freies Zentrifugenröhrchen, RNase-/DNase-freie Spitze, 0,2 ml steriles qPCR-Röhrchen.
◆ qPCR-Gerät, Pipette, Tischzentrifuge (≥13.400×g) (je nach experimentellem Bedarf) usw.
Sicherheit
◆ Dieses Produkt dient ausschließlich wissenschaftlichen Forschungszwecken. Bitte verwenden Sie es nicht für pharmazeutische, klinische, Lebensmittel- oder Kosmetikzwecke.
◆ Tragen Sie bei der Verwendung von Chemikalien geeignete Laborkleidung, Handschuhe, Schutzbrille usw.
BetriebFührer
Zelllysesysteme, RT-Systeme und Ergänzungspakete mit qPCR-Reaktionslösungen können separat erworben werden. Einzelheiten finden Sie in Anhang 1 (SEITE 13).
Bedienerführung
A: Proben-RNA-Freisetzung
1.Zellen wurden vorbehandelt: Waschen Sie die Zellkulturplatte mit kaltem PBS und lysieren Sie dann die Zellen (10-10).6), 106 als die Anzahl der Zellen, wird Foregene the Cell ein RNA Isolation Kit the Total (DE-03111) oder Animal Total RNA Isolation Kit (DE-03011) für die RNA-Extraktion und -Reinigung empfohlen.
1.1.Adhärente Zellen (Beispiel: 24-Well-Platte)
1.1.1.Bestimmen Sie die Anzahl der Zellen in jeder Vertiefung und stellen Sie fest, dass die Anzahl der Zellen 1 × 10 beträgt5, und entfernen Sie das Kulturmedium mit einer Pipette aus der Kulturschale.
1.1.2.200 μl vorgekühltes 1 × PBS in jede Vertiefung geben.Pipettieren Sie nicht wiederholt und entfernen Sie PBS aus den Vertiefungen.Kippen Sie die Platte und entfernen Sie so viel PBS wie möglich.Fahren Sie mit Schritt 2 fort.
1.1.3.Zum Waschen der Zellen wurde eine andere Zellkulturschale oder eine Referenznummertabelle 1-1 in eine Zellkulturschale mit vorgekühltem 1 × PBS gegeben.
Tabelle 1-1: PBS-Dosierung für unterschiedliche Zellzahlen
| Kulturplattentyp | Anzahl Zellen/Well | 1 × PBS/Well |
| 6-gut | 1× 106 | 1000 μl |
| 12-well | 2× 105 | 400 μl |
| 24-well | 105 | 200 μl |
| 96-well | 104 | 50 μl |
| 384-naja | 5× 103 | 25 μl |
Notiz::Um eine feste Adhäsion der Zellen zu gewährleisten,Beim Waschen wird ein großer Zellverlust vermieden.
1.2.Suspensionszellen oder adhärente Zellen, kultiviert in nicht porösen Platten
1.2.1.Anhaftende Zellen, die in Nicht-Multi-Well-Platten kultiviert werden (Suspensionszellen beginnen mit dem nächsten Schritt 1.2.2), sammeln und trennen die Zellen gemäß der normalen Zellsammelmethode und legen sie in eine Kulturplatte oder ein Zentrifugenröhrchen;Wenn Trypsinisierung verwendet wird, ist eine Zentrifugation erforderlich, um die Zellen zu sammeln und restliches Trypsin zu entfernen. Durch Zugabe von PBS wurden die Zellen in einzelne Zellen resuspendiert, um die Zellen zu dispergieren.
1.2.2.Nachdem die Anzahl der Zellen gezählt wurde, wurden 1×10 Zellen aliquotiert5 Eines in Zentrifugenröhrchen, Zellen durch 10-minütige Zentrifugation bei 1000 × g sammeln.
1.2.3.200 μl PBS in das Zentrifugenröhrchen geben, nicht wiederholt pipettieren und das PBS direkt ansaugen.Fahren Sie mit Schritt 2 fort. (Wenn die Ausfällung schwierig ist und die Zellen erneut resuspendiert wurden, kann eine Zentrifugation bei 1000 × g durchgeführt werden. 10 Minuten nach dem Verwerfen des Überstands und dem Zellpellet fahren Sie mit Schritt 2 fort.)
2. Zelllyse: Entfernen Sie Puffer CL, dessen Temperatur auf Raumtemperatur äquilibriert ist, DNA Eraser und Foregene Protease Plus II gemäß der folgenden Tabelle 1-2. Bereiten Sie das Lysesystem vor: (Die Lyselösung ist gebrauchsfertig).
Tabelle 1-2: Spaltung Systemvorbereitung (Hinweis: bei der Vorbereitung auf Eis)
| Komponente (Zelllyse-Mastermix) | 6-Well-Platte | 12-Well-Platte | 24-Well-Platte | 96-Well-Platte | 384-Well-Platte |
| 1000 μl/Well | 400 μl/Well | 200 μl/Well | 50 μl/Well | 25 μl/Well | |
| Puffer CL | 960μl | 384μl | 192μl | 48μl | 24μl |
| DNA-Radierer | 20μl | 8μl | 4μl | 1μl | 0,5 μl |
| Foregene Protease Plus II | 20μl | 8μl | 4μl | 1 μl | 0,5 μl |
3. (24-Well-Platte als Beispiel) 200 μl Zelllyse-Mastermix in jedes Well pipettieren, wiederholt 5–10 Mal blasen, 5 Minuten bei Raumtemperatur (20–25 °C) inkubieren.
Notiz::Um die Bildung von Blasen zu vermeiden, stellen Sie bitte beim Pipettieren die Pipettenskala auf 200 μl oder weniger ein.Die Zellen können nach der Lyse trüb erscheinen, was normal ist.
4. (24-Well-Platte als Beispiel) werden 20 μl Puffer ST in die Flüssigkeit gegeben (verschiedene Lysesysteme geben Puffer ST in der in Tabelle 1-3 angegebenen Menge hinzu), das Pipettieren wird 5–10 Mal wiederholt und bei Raumtemperatur (20–25 °C) wird 2 Minuten lang inkubiert.
Notiz::Die Pipettenspitze befindet sich unterhalb der Oberfläche und stellt so sicher, dass das Lysat hinzugefügt wird,Um die Bildung von Blasen zu vermeiden, stellen Sie bitte beim Pipettieren die Pipettenskala auf 200 μl oder weniger ein.
Tabelle 1-3::Puffer ST hinzufügen
| Puffer ST | 6-Well-Platte | 12-Well-Platte | 24-Well-Platte | 96-Well-Platte | 384-Well-Platte |
| 100 μl/Well | 40 μl/Well | 20 μl/Well | 5 μl/Well | 2,5 μl/Well |
5. Das Lysat wird für nachfolgende RT-qPCR-Experimente verwendet.Wenn die nachfolgenden Experimente nicht rechtzeitig durchgeführt werden können, lassen Sie es bitte nicht länger als 2 Stunden auf Eis und lagern Sie es bei -20 °C oder -80 °C (nicht länger als drei Monate).
B: Vorbereitung des RT-Systems
1. Nehmen Sie 5 × Direct RT Mix heraus und legen Sie es auf ein Eisbad, lassen Sie es auf natürliche Weise schmelzen und mischen Sie es vorsichtig für die spätere Verwendung.Nehmen Sie RNase-Free ddH2O heraus, schmelzen Sie es und legen Sie es zur späteren Verwendung auf ein Eisbad.Bereiten Sie das Reaktionssystem auf Eis gemäß Tabelle 2-1 unten vor.
Tabelle 2-1: Vorbereitung des RT-Reaktionssystems
| RT-System fügt Inhalte hinzu | Mit der Menge | Endgültige Konzentration | |
| 5 × Direkter RT-Mix | 4μl | 8 μl | 1 × |
| Zelllysate (RNA-Vorlage) | 4 μl | 8 μl | Bereichsanpassung hinzufügen (10 -40%) |
| RNase-freies ddH2O | 12 μl | 24 μl | - |
| Volle Lautstärke | 20 μl | 40 μl | - |
2. Nach Abschluss der Systemformulierung vorsichtig mischen und kurz zentrifugieren unter den folgenden Reaktionsbedingungen (RT-Reaktion) in Tabelle 2-2.
Tabelle 2-2: Einstellung der RT-Reaktionsbedingungen
| Schritt | Temperatur | Zeit | Inhalt |
| 1 | 42 °C | 15-30 Min | cDNA-Synthese |
| 2 | 95 °C | 5 Minuten | Inaktivierte Reverse Transkriptase |
| 3 | 4 °C | N / A |
3. Nach Abschluss der Reaktion wurde das Reaktionsprodukt für qPCR direkt auf Eis gelegt, bitte stellen Sie die Langzeitkonservierung auf -20 ℃ oder -80 ℃.
Hinweis: Aufgrund der Verwendung einer nicht gereinigten Vorlage können im Reverse-Transkriptionsprodukt weiße Niederschläge auftreten.Dies ist ein normales Phänomen.Zentrifugieren Sie den Überstand sofort für nachfolgende Experimente.
Die resultierende RT-Reaktionslösung wird zu den Real-Time-PCR-Reaktionssystemen des nächsten Schritts hinzugefügt. Es wird empfohlen, Mengen im Bereich von 10–30 % des Reaktionssystems hinzuzufügen.
C: Vorbereitung des qPCR-Reaktionssystems
1. Angemessene Menge an B, hergestellt in Schritt cDNA-Vorlage gemäß der folgenden Tabelle 3-1, um ein Reaktionssystem vorzubereiten.
Hinweis: Die Menge der cDNA-Vorlage macht 10–30 % des qPCR-Systems aus.Fügen Sie beispielsweise in einem 20-μl-qPCR-System 2–6 μl Lysepuffer hinzu, jedoch nicht mehr als 6 μl.
2. Die Optimierung guter qPCR-Bedingungen (Annealing-Temperatur usw.) für die qPCR-Reaktion (die Reaktionsbedingungen sind in Tabelle 3-2 angegeben).
Hinweis: Versuchen Sie, optimierte Bedingungen für qPCR-Reaktionen zu verwenden, um bessere Ergebnisse zu erzielen.
Tabelle 3-1: Vorbereitung des PCR-Reaktionssystems
| RT-System fügt Inhalte hinzu | Mit der Menge | Endgültige Konzentration |
| 2× Direktes qPCR Mix-Taqman | 10 μl | 1× |
| Vorwärtsprimer (10 μM) | 0,4 μl | 50-900 nM 1* |
| Reverse Primer (10 μM) | 0,4 μl | 50-900 nM 1* |
| Sonde (10 μM) | 0,2 μl | 200 nm |
| cDNA-Vorlage (erhalten in Schritt B) | 4 μl | 10-30 % |
| RNase-freies ddH2O | — | |
| 20×ROX-Referenzfarbstoff 3* | — | — |
| Volle Lautstärke | 20 μl |
1*: Die Primerkonzentration kann im Bereich von 50–900 nM angepasst werden, wenn die Primerreaktionsleistung schlecht ist.
Hinweis: Das qPCR-System kann je nach experimentellen Anforderungen und Fluoreszenz-Cycler-Modell angepasst werden.Für qPCR in einem 50μl-System, passen Sie die Reagenziendosierung proportional gemäß 20 anμl-System.
| Echtzeit-PCR-Gerät | Endkonzentration des ROX-Referenzfarbstoffs |
| ABI PRISM7000/7300/7700/7900HT/Step One usw. | 1× (z. B. 20-μl-System, 1 μl 20×ROX-Referenzfarbstoff hinzufügen) |
| ABI 7500/7500 Fast und StratageneMx3000P/Mx3005P/Mx4000 usw. | 0,5 × (z. B. 20 μl-System, 0,5 μl 20 × ROX-Referenzfarbstoff hinzufügen) |
2*: Wählen Sie die geeignete Endkonzentration des ROX-Referenzfarbstoffs entsprechend dem quantitativen Fluoreszenz-Thermocycler.Die am besten geeigneten ROX-Referenzfarbstoffkonzentrationen für gängige quantitative Fluoreszenzcycler sind in der folgenden Tabelle aufgeführt:
Tabelle 3-2: qPCR-Reaktionsbedingungen werden bereitgestellt
| Zwei Schritte | Temperatur | Zeit | Fahrräder | Inhalt |
| 1 | 95℃ | 3 Minuten | 1 | Vordenaturierung |
| 2 | 95℃ | 5-10 Sek | 40 | Template-Denaturierung |
| 3 | 60-65℃ | 20-30 Sek | Glühen / Erweiterung |
Hinweis: Um den besten qPCR-Effekt zu erzielen, kann die Gradienten-PCR verwendet werden, um die Reaktionsbedingungen für verschiedene Templates und verschiedene Primer zu optimieren.Die PCR-Reaktionsbedingungen variieren je nach Fluoreszenzanalysator, Matrize, Primer usw. Im spezifischen Betrieb müssen die optimalen Reaktionsbedingungen entsprechend den spezifischen Bedingungen des quantitativen Fluoreszenz-Thermocyclers, des Matrizentyps, der Größe des interessierenden Fragments, der Basensequenz des amplifizierten Fragments sowie des GC-Gehalts und der Länge der Primer, einschließlich Annealing-Temperatur, Reaktionszeit usw., entworfen werden.
Prinzipien des Real-Time-PCR-Primer-Designs
Vorwärts-Primer und Rückwärts-Primer
Für die Echtzeit-PCR ist das Primerdesign sehr wichtig.Primer hängen mit der Spezifität und Effizienz der PCR-Amplifikation zusammen und können unter Bezugnahme auf die folgenden Prinzipien entworfen werden:
◆ Primerlänge: 18–30 bp.
◆ GC-Gehalt: 40–60 %.
◆ Tm-Wert: Primer-Design-Software wie Primer 5 kann den Tm-Wert des Primers angeben.Die Tm-Werte der Upstream- und Downstream-Primer sollten möglichst nahe beieinander liegen.Es kann auch die Tm-Berechnungsformel verwendet werden: Tm = 4 °C (G + C) + 2 °C (A + T).Bei der Durchführung einer PCR wird im Allgemeinen eine Temperatur unterhalb des Primer-Tm-Werts von 5 °C als Annealing-Temperatur gewählt (die entsprechende Erhöhung der Annealing-Temperatur kann die Spezifität der PCR-Reaktion erhöhen).
◆ Primer und PCR-Produkte:
◆ Die Länge des Design-Primers für das PCR-Amplifikationsprodukt beträgt vorzugsweise 100–150 bp.
◆ Designprimer im sekundären Strukturbereich der Vorlage sollten so weit wie möglich vermieden werden.
◆ Vermeiden Sie die Bildung von zwei oder mehr komplementären Basen zwischen den 3′-Enden der Upstream- und Downstream-Primer.
◆ Die terminale Basis des Primers 3‘ kann nicht mit 3 weiteren aufeinanderfolgenden G oder C vorhanden sein.
◆ Die Primer selbst dürfen keine komplementären Strukturen aufweisen, da sonst eine Haarnadelstruktur gebildet wird, die die PCR-Amplifikation beeinträchtigt.
◆ ATCG sollte möglichst gleichmäßig in der Primersequenz verteilt sein und die 3′-terminale Base sollte als T vermieden werden.
Anhang1::Cell DirectRT-qPCR Kit-Komponentent Ergänzungspaket
1. Zelllyselösung
|
| |||
| Kit-Komponenten (24-Well-Lysesystem / Well) | DRT-01011-A1 | DRT-01011-A2 | |
| 100 T | 500 T | ||
| TeilICH | Puffer CL | 20 ml | 100 ml |
| Foregene Protease Plus II | 400 μl | 1 ml × 2 | |
| Puffer ST | 1 ml × 2 | 10 ml | |
| TeilII | DNA-Radierer | 400 μl | 1 ml × 2 |
|
| |
| Kit-Komponenten (20 μl Reaktionssystem) | DRT-01011-B1 |
| 200 T | |
| 5× Direkter RT-Mix | 800 μl |
| RNase-freies ddH2O | 1,7 ml × 2 |
3.qPCR-Mix
|
| ||
| Kit-Komponenten (20 μl Reaktionssystem) | DRT-01021-C1 | DRT-01021-C2 |
| 200 T | 1000 T | |
| 2× Direktes qPCR Mix-Taqman | 1 ml × 2 | 1,7 ml × 6 |
| 20× ROX-Referenzfarbstoff | 40 μl | 200 μl |
| RNase-freies ddH2O | 1,7 ml | 10 ml |
Das Foregene der Welt
Foregene Co., Ltd
Tel: 028-83360257, 028-83361257
E-mail :info@foregene.com
Http://www.foregene.com