Gesamt-RNA-Isolierungskit für Pflanzen Gesamt-RNA-Reinigungskit für Pflanzen mit niedrigem Gehalt an Polysacchariden und Polyphenolen
Spezifikationen
50 Vorbereitungen, 200 Vorbereitungen
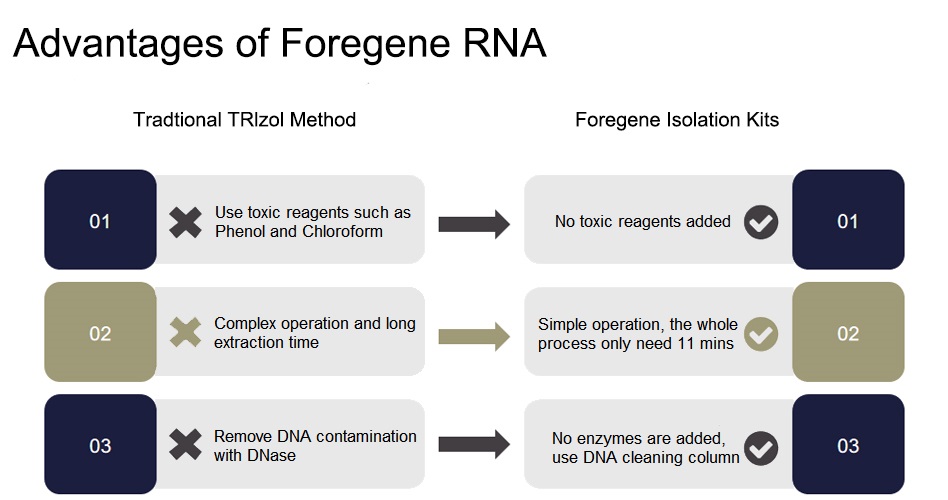
Das Kit nutzt die von Foregene entwickelte Spin-Säule und Formel, mit der hochreine und hochwertige Gesamt-RNA aus verschiedenen Pflanzengeweben mit niedrigem Polysaccharid- und Polyphenolgehalt effizient extrahiert werden kann.Für Pflanzenproben mit hohem Polysaccharid- oder Polyphenolgehalt wird die Verwendung des Plant Total RNA Isolation Plus Kit empfohlen, um bessere RNA-Extraktionsergebnisse zu erzielen.Das Kit enthält die DNA-Reinigungssäule, mit der sich genomische DNA problemlos aus dem Überstand und dem Gewebelysat entfernen lässt.Nur-RNA-Säulen können RNA effektiv binden.Das Kit kann eine große Anzahl von Proben gleichzeitig verarbeiten.
Das gesamte System enthält keine RNase, sodass die gereinigte RNA nicht abgebaut wird.Puffer PRW1 und Puffer PRW2 können sicherstellen, dass die gewonnene RNA nicht durch Proteine, DNA, Ionen und organische Verbindungen kontaminiert ist.
Kit-Komponenten
| Puffer PSL1, Puffer PS, Puffer PSL2 |
| Puffer PRW1, Puffer PRW2 |
| RNase-freies ddH2O, DNA-Reinigungssäule |
| Nur-RNA-Säule |
| Anweisungen |
Merkmale und Vorteile
■ Betrieb bei Raumtemperatur (15-25℃) während des gesamten Prozesses, ohne Eisbad und Zentrifugation bei niedriger Temperatur.
■ Komplettes Kit RNase-frei, kein Grund zur Sorge über RNA-Abbau.
■ Die DNA-Reinigungssäule bindet spezifisch an DNA, sodass das Kit genomische DNA-Kontaminationen ohne Zugabe von DNase entfernen kann.
■ Hohe RNA-Ausbeute: Nur-RNA-Säule und einzigartige Formel können RNA effizient reinigen.
■ Hohe Geschwindigkeit: einfach zu bedienen und innerhalb von 30 Minuten fertig.
■ Sicherheit: Es ist kein organisches Reagenz erforderlich.
■ Hohe Qualität: Die gereinigten RNA-Fragmente sind von hoher Reinheit, frei von Proteinen und anderen Verunreinigungen und können für verschiedene nachgelagerte experimentelle Anwendungen verwendet werden.
Kit-Anwendung
Es eignet sich für die Extraktion und Reinigung von Gesamt-RNA aus frischen oder gefrorenen Pflanzengewebeproben (insbesondere frischem Pflanzenblattgewebe) mit niedrigem Polysaccharid- und Polyphenolgehalt.
Arbeitsablauf
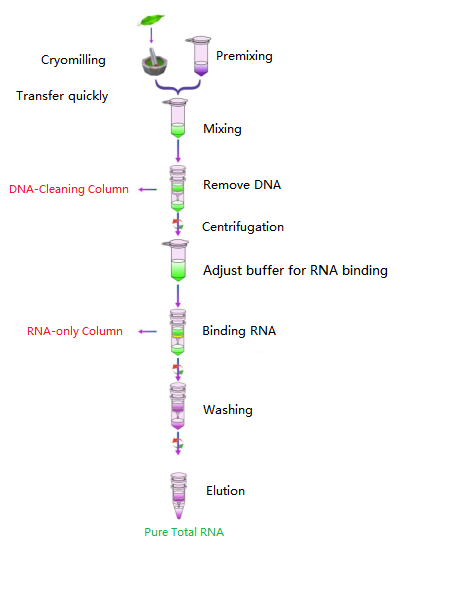
Diagramm
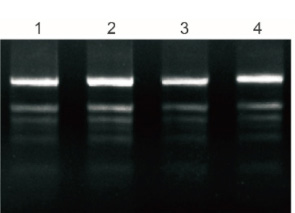
Das Plant Total RNA Isolation Kit Plus verarbeitete 50 mg frische Blätter von Polysacchariden und Polyphenolen, und 5 % gereinigte RNA wurden durch Elektrophorese getestet.
1: Banane
2: Ginkgo
3: Baumwolle
4: Granatapfel
Lagerung und Haltbarkeit
Das Kit kann 12 Monate lang bei Raumtemperatur (15–25 °C) in trockener Umgebung und bei 2–8 °C länger (24 Monate) gelagert werden.
Puffer PSL1 kann nach Zugabe von 2-Hydroxy-1-ethanthiol (optional) einen Monat lang bei 4 °C gelagert werden.
Leitfaden zur Problemanalyse
Die folgende Analyse der Probleme, auf die Sie stoßen könnenGesamtanlageRNA extraction will help you with your experiments. In addition, for other experimental or technical problems in addition to operating instructions and problem analysis, we have dedicated technical support to help you. If you have any needs, please contact us at: 028-83360257 or E-mali : Tech@foregene.com.
Die Schleudersäule ist verstopft
Eine Blockierung der Spin-Säule führt dazu, dass die RNA-Ausbeute verringert wird oder sogar nicht mehr gereinigt werden kann, um RNA zu erhalten, und die Qualität der erhaltenen RNA wird gering sein.
Analyse gemeinsamer Ursachen:
1. Die Probe ist nicht vollständig zerbrochen.
Eine unvollständige Probenfragmentierung kann die DNA-Reinigungssäule blockieren, was sich auch auf die RNA-Ausbeute und -Qualität auswirken kann.Wir empfehlen, bei der Probenfragmentierung schnell ausreichend flüssigen Stickstoff einzumahlen, um das Gewebe wie Zellwände und Zellmembranen der Proben so weit wie möglich aufzubrechen.Für Pflanzenproben von Polyphenolpolysacchariden empfehlen wir die Verwendung des Plant Total RNA Isolation Kit Plus.
2.Aspirieren Sie den isolierten Überstand der DNA-Reinigungssäule und das mögliche Zelltrümmerpellet.
Angesaugte Zelltrümmerpellets können die reine RNA-Säule während der RNA-Adsorptionsverfahren verstopfen (siehe Schritt 5 des Verfahrens, Schritt 6 des Polysaccharid-Polyphenol-Verfahrens).Wir empfehlen, beim Absaugen dieses Überstands Vorsicht walten zu lassen, um das Absaugen von Zelltrümmern zu vermeiden.
3. Die anfängliche Probenmenge ist zu groß.
Eine übermäßige Probenverwendung führt zu einer unvollständigen Probenfragmentierung oder einer unvollständigen Lyse der Zellen durch Puffer PRL1 oder Puffer PSL1, was zu einer Verstopfung der Reinigungssäule für Reinigungsvorgänge führt.Das Plant Total RNA Isolation Kit hat ein anfängliches Maximum von 50 mg pro Einzelreinigung einer behandelten Probe.Für Pflanzenproben von Polyphenolpolysacchariden empfehlen wir Ihnen, das Plant Total RNA Isolation Kit Plus auszuprobieren.
4. Die Temperatur der Zentrifuge ist zu niedrig.
Die Isolierung und Reinigung der gesamten RNA, mit Ausnahme des Aufschlusses des Probengewebes mit flüssigem Stickstoff, erfolgt bei Raumtemperatur (20–25 °C).Einige Niedertemperaturzentrifugen haben Temperaturen unter 20 °C, was zu Verstopfungen in der DNA-Reinigungssäule und/oder der reinen RNA-Säule führen kann.Wenn dies auftritt, stellen Sie die Zentrifugentemperatur auf 20–25 °C ein und erwärmen Sie die Lysemischung und/oder die Zugabe des Ethanol-Trennüberstands auf 37 °C vor.
Es wird keine RNA extrahiert oder die RNA-Ausbeute ist gering
Normalerweise gibt es viele Faktoren, die die Rückgewinnungseffizienz beeinflussen, wie zum Beispiel: der RNA-Gehalt der Probe, die Betriebsmethode, das Elutionsvolumen usw.
Analyse der häufigsten Ursachen wie folgt:
1. Während des Vorgangs wurde ein Eisbad oder eine Zentrifugation bei niedriger Temperatur (4 °C) durchgeführt.
Vorschlag: Während des gesamten Prozesses bei Raumtemperatur (15–25 °C) arbeiten, kein Eisbad und keine Zentrifugation bei niedriger Temperatur durchführen.
2.Die RNA wurde aufgrund einer unsachgemäßen Konservierung der Probe oder einer Langzeitkonservierung der Probe abgebaut.
Empfehlung: Frisch entnommene Proben sollten schnell in flüssigem Stickstoff eingefroren und dann für längere Zeit bei -80 °C gelagert werden. Wiederholtes Einfrieren und Auftauen der Proben ist zu vermeiden.oder die Proben sofort in RNA-Stabilisator-RNAlater-Lösung einweichen (tierische Proben).
3.Eine unzureichende Probenfragmentierung und Lyse führen zur Verstopfung der Reinigungssäule.
Vorschlag: Stellen Sie beim Mahlen des Gewebes bitte sicher, dass das Gewebe ausreichend gemahlen ist, und übertragen Sie es schnell auf den vorbereiteten Puffer PSL1 (bestätigen Sie, dass der richtige Anteil an β-ME hinzugefügt wurde, siehe Schritt 1 des Verfahrens).
4.Der Eluent wurde falsch hinzugefügt.
Vorschlag: Stellen Sie sicher, dass RNase-Free ddH2O wird in die Mitte der Membran der Reinigungssäule getropft.
5. Dem Puffer PSL2 oder dem Puffer PRW2 wurde nicht das richtige Volumen an absolutem Ethanol zugesetzt.
Vorschlag: Bitte befolgen Sie die Anweisungen, geben Sie die richtige Menge absolutes Ethanol zu Puffer PSL2 und Puffer PRW2 hinzu und mischen Sie gut, bevor Sie das Kit verwenden.
6. Die Menge der Gewebeprobe ist unangemessen.
Vorschlag: Verwenden Sie 50 mg Gewebe pro 500 μl Puffer PSL1.Wenn zu viel Gewebe verwendet wird, verringert sich die Menge der extrahierten RNA und die Reinheit der resultierenden RNA wird ebenfalls verringert.Wir empfehlen dringend, dass die anfängliche Probendosis 50 mg pro RNA-Extraktionsvorgang nicht überschreiten sollte.
7. Unangemessenes Elutionsvolumen oder unvollständige Elution.
Vorschlag: Das Eluentenvolumen der Reinigungssäule beträgt 50–200 μl;Wenn der Elutionseffekt nicht zufriedenstellend ist, wird empfohlen, die Zeit bei Raumtemperatur nach Zugabe von vorgewärmtem RNase-Free ddH zu verlängern2O, etwa 5-10min.
8. Die Reinigungssäule weist nach dem Waschen mit Puffer PRW2 Ethanolrückstände auf.
Vorschlag: Wenn das leere Röhrchen 1 Minute lang zentrifugiert wird und nach dem Waschen im Puffer PRW2 noch Ethanol übrig ist, können Sie die Zeit für die Zentrifugation des leeren Röhrchens auf 2 Minuten verlängern oder die Reinigungssäule 5 Minuten lang auf Raumtemperatur stellen, um das restliche Ethanol vollständig zu entfernen.
9. Das Kit wurde falsch verwendet.
Vorschlag: Bei Pflanzenproben polyphenolischer Polysaccharide können mit gängigen Kits wie dem Plant Total RNA Isolation Kit möglicherweise keine idealen RNA-Proben erhalten werden.Wir empfehlen Ihnen die Verwendung des Plant Total RNA IsolationKit Plus, das speziell für polyphenolische Polysaccharid-Pflanzenproben entwickelt wurde.Ein Kit, das speziell für die Extraktion von RNA aus Polyphenol- und Polysaccharid-Pflanzenproben entwickelt wurde.
Der OD260/OD280-Wert ist niedrig
RNA-Elution mit ddH2O und für Spektrophotometer-Messungen verwendet, führt zu niedrigen OD260/OD280-Werten.Wir empfehlen die Verwendung von 10 mM Tris-HCl, pH 7,5 (anstelle von RNase-Free ddH).2O, um RNA zu eluieren), um relativ korrekte OD260/OD280-Werte zu erhalten, siehe „RNA-Konzentrations- und Reinigungsassays“ auf Seite 19.
Die gereinigte RNA wird abgebaut
Die Qualität der gereinigten RNA hängt von Faktoren wie Probenkonservierung, RNase-Kontamination und Manipulation ab.
Analyse häufiger Ursachen:
1. Gewebeproben wurden nicht rechtzeitig nach der Entnahme gelagert.
Empfehlung: Wenn die Gewebeproben nicht rechtzeitig nach der Entnahme verwendet werden, lagern Sie sie bitte sofort in flüssigem Stickstoff bei niedriger Temperatur oder übertragen Sie sie zur Langzeitlagerung nach schnellem Einfrieren in flüssigem Stickstoff auf -80 °C oder tauchen Sie die Proben sofort in die RNA-Stabilisator-RNAlater-Lösung (Tierproben).Versuchen Sie für die RNA-Extraktion frisch gesammelte Gewebeproben zu verwenden.
2. Wiederholtes Einfrieren und Auftauen von Gewebeproben.
Vorschlag: Bei der Lagerung von Gewebeproben ist es am besten, diese zur Konservierung in kleine Stücke zu schneiden und bei der Verwendung einen Teil davon herauszunehmen, um den Abbau der RNA durch wiederholtes Einfrieren und Auftauen der Proben zu vermeiden.
3. RNase wird im Operationssaal eingeführt oder es werden keine Einweghandschuhe, Masken usw. getragen.
Vorschlag: RNA-Extraktionsexperimente werden am besten in separaten RNA-Vorgängen durchgeführt, der Labortisch sollte vor dem Experiment gereinigt werden und während des Experiments sollten Einweghandschuhe und -masken getragen werden, um den RNA-Abbau durch die Einführung von RNase weitestgehend zu vermeiden.
4. Das Reagenz wird während der Verwendung durch RNase kontaminiert.
Vorschlag: Ersetzen Sie es durch eine neue Serie pflanzlicher Gesamt-RNA-Extraktionskits für entsprechende Experimente.
5. Die zur RNA-Manipulation verwendeten Zentrifugenröhrchen und Pipettenspitzen sind mit RNase kontaminiert.
Vorschlag: Stellen Sie sicher, dass die bei der RNA-Extraktion verwendeten Zentrifugenröhrchen, Pipettenspitzen, Pipetten usw. alle RNase-frei sind.
Bedienungsanleitung: