Es ist allgemein bekannt, dass im zentralen Dogma RNA der Transkriptionsvermittler zwischen DNA- und Proteinexpression ist.Im Vergleich zum Nachweis von DNA kann der Nachweis von RNA die Genexpression in Organismen objektiver widerspiegeln.Experimente mit RNA umfassen: qRT-PCR, RNA-Seq und Fusionsgen-Detektion usw. Aufgrund der Eigenschaften der RNA selbst (der Zuckerring der RNA hat eine freie Hydroxylgruppe mehr als der Zuckerring der DNA) in Verbindung mit einer großen Anzahl von RNasen in der Umgebung ist RNA instabiler und lässt sich leichter abbauen als DNA.Müll rein, Müll raus: Wenn die Qualität der RNA nicht gut ist, müssen die experimentellen Ergebnisse unbefriedigend sein, was sich insbesondere in ungenauen Daten oder schlechter Wiederholbarkeit äußert.Daher sollte der Verarbeitung von RNA mehr Aufmerksamkeit gewidmet werden, und auch die Verknüpfung der Qualitätskontrolle ist wichtiger, um die Präzision und Genauigkeit nachfolgender experimenteller Daten sicherzustellen.
Für die Qualitätskontrolle von RNA gibt es im Allgemeinen die folgenden gängigen Methoden:
- Spektrophotometrie
- Agarose-Gelelektrophorese
- Agilent Bioanalyzer
- quantitative Echtzeit-Fluoreszenz-PCR
- Qubit-Fluoreszenzfarbstoffmethode
01 Spektrophotometrie
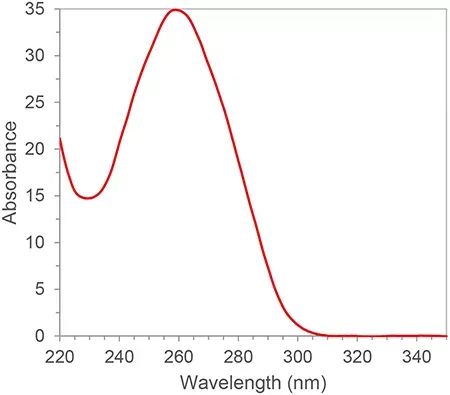
RNA verfügt über konjugierte Doppelbindungen und weist einen Absorptionspeak bei einer Wellenlänge von 260 nm auf.Nach dem Lambert-Beerschen Gesetz können wir die RNA-Konzentration aus dem Absorptionspeak bei 260 nm berechnen.Darüber hinaus können wir die Reinheit der RNA auch anhand des Verhältnisses der Absorptionspeaks bei 260 nm, 280 nm und 230 nm berechnen.280 nm und 230 nm sind die Absorptionspeaks von Proteinen bzw. kleinen Molekülen.Das Verhältnis von A260/A280 und A260/A230 der qualifizierten RNA-Reinheit sollte größer als 2 sein. Wenn es weniger als 2 beträgt, bedeutet dies, dass die RNA-Probe eine Protein- oder Kleinmolekülverunreinigung aufweist und erneut gereinigt werden muss.Kontaminationsquellen beeinträchtigen nachfolgende Experimente, indem sie beispielsweise die Amplifikationseffizienz von PCR-Reaktionen hemmen, was zu ungenauen quantitativen Ergebnissen führt.Die Reinheit der RNA hat großen Einfluss auf die späteren Ergebnisse, daher ist die Spektrophotometrie im ersten Schritt von Nukleinsäureexperimenten generell ein unverzichtbares Glied zur Qualitätskontrolle.
Abbildung 1. Typisches RNA/DNA-Absorptionsspektrum
02 Agarosegelelektrophorese
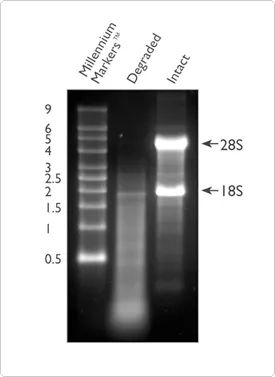
Neben der Reinheit ist auch die Integrität der RNA einer der wichtigen Indikatoren zur Beurteilung der Qualität der RNA.Der Abbau der RNA führt zu einer großen Anzahl kurzer Fragmente in der Probe, sodass die Anzahl der RNA-Fragmente, die effektiv nachgewiesen und von der Referenzsequenz abgedeckt werden können, verringert wird.Die RNA-Integrität kann durch Elektrophorese der Gesamt-RNA auf einem 1 %igen Agarosegel überprüft werden.Mit dieser Methode können Sie das Gel selbst konfigurieren oder das vorgefertigte E-Gel™-System zur Integritätsprüfung verwenden.Mehr als 80 % der Gesamt-RNA ist ribosomale RNA, der Großteil davon besteht aus 28S- und 18S-rRNA (in Säugetiersystemen).RNA von guter Qualität zeigt zwei offensichtliche helle Balken, nämlich 28S- bzw. 18S-helle Balken, bei 5 Kb und 2 Kb, und das Verhältnis liegt tendenziell nahe bei 2:1.Wenn es sich in einem diffusen Zustand befindet, bedeutet dies, dass die RNA-Probe möglicherweise abgebaut wurde. Es wird empfohlen, die später beschriebene Methode zu verwenden, um die Qualität der RNA weiter zu testen.
Abbildung 2. Vergleich von degradierter (Spur 2) und intakter RNA (Spur 3) bei der Agarosegelelektrophorese
03 Agilent Bioanalyzer
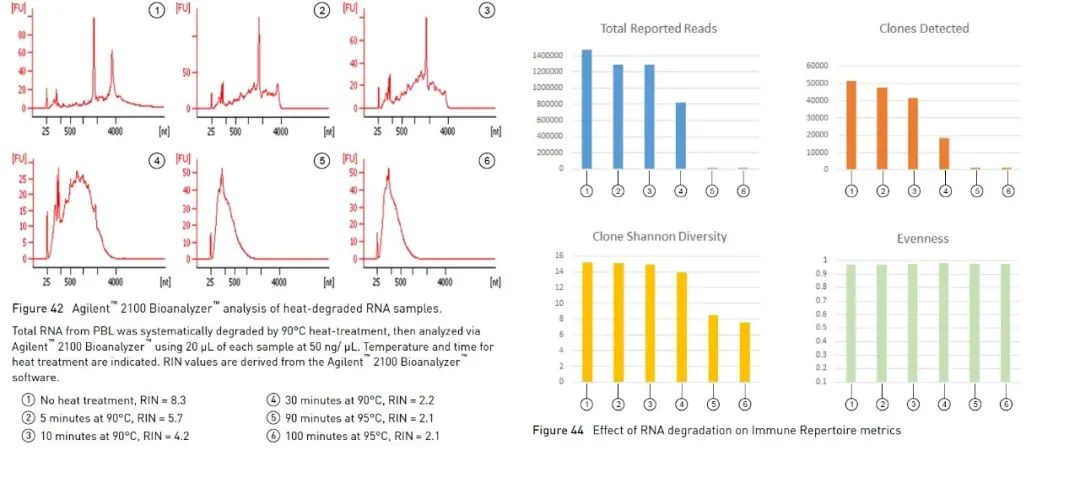
Zusätzlich zur oben beschriebenen Agarosegel-Elektrophorese-Methode, die uns helfen kann, die Integrität von RNA einfach und schnell zu identifizieren, können wir auch den Agilent Bioanalyzer verwenden, um die Integrität von RNA zu bestimmen.Es nutzt eine Kombination aus Mikrofluidik, Kapillarelektrophorese und Fluoreszenz, um die RNA-Konzentration und -Integrität zu beurteilen.Mithilfe des integrierten Algorithmus zur Analyse des Profils der RNA-Probe kann der Agilent Bioanalysator einen Referenz-RNA-Integritätswert, die RNA-Integritätszahl (im Folgenden als RIN bezeichnet) berechnen [1].Je größer der RIN-Wert ist, desto höher ist die Integrität der RNA (1 ist extrem abgebaut, 10 ist am vollständigsten).Einige Experimente mit RNA schlagen vor, RIN als Parameter zur Qualitätsbewertung zu verwenden.Am Beispiel von Hochdurchsatz-Sequenzierungsexperimenten (im Folgenden als NGS bezeichnet) legen die Richtlinien von Oncomine™ Human Immune Repertoire, das zum Nachweis von B-Zell- und T-Zell-Antigenrezeptoren in der Oncomine-Panel-Serie von Thermo Fisher verwendet wird, nahe, dass Proben mit RIN-Werten über 4, effektivere Lesevorgänge und Klone gemessen werden können (Abbildung 3).Es gibt unterschiedliche empfohlene Bereiche für verschiedene Panels, und oft kann ein höherer RIN effektivere Daten liefern.
Abbildung 3: In Oncomine™ Human Immune Repertoire-Experimenten können Proben mit einem RIN von mehr als 4 effektivere Lesevorgänge und T-Zell-Klone erkennen.【2】
Allerdings weist der RIN-Wert auch einige Einschränkungen auf.Obwohl RIN eine hohe Korrelation mit der Qualität experimenteller NGS-Daten aufweist, ist es für FFPE-Proben nicht geeignet.FFPE-Proben werden seit langem chemisch behandelt und die extrahierte RNA weist im Allgemeinen einen relativ niedrigen RIN-Wert auf.Dies bedeutet jedoch nicht, dass die effektiven Daten des Experiments unbefriedigend sein müssen.Um die Qualität von FFPE-Proben genau beurteilen zu können, müssen wir andere Messungen als RIN verwenden.Zusätzlich zur RIN kann der Agilent Bioanalyzer auch den DV200-Wert als Bewertungsparameter für die RNA-Qualität berechnen.DV200 ist ein Parameter, der den Anteil von Fragmenten größer als 200 bp in einer RNA-Probe berechnet.DV200 ist ein besserer Indikator für die FFPE-Probenqualität als RIN.Für die durch FFPE extrahierte RNA besteht eine sehr hohe Korrelation mit der Anzahl der Gene, die effektiv nachgewiesen werden können, und der Vielfalt der Gene [3].Obwohl DV200 die Mängel bei der Qualitätserkennung von FFPE ausgleichen kann, kann der Agilent-Bioanalysator die Qualitätsprobleme in RNA-Proben immer noch nicht umfassend analysieren, einschließlich der Frage, ob die Proben Inhibitoren enthalten.Inhibitoren selbst können die Amplifikationseffizienz nachgeschalteter Experimente beeinträchtigen und die Menge an nützlichen Daten verringern.Um herauszufinden, ob sich in der Probe ein Inhibitor befindet, können wir die im Folgenden beschriebene quantitative Echtzeit-Fluoreszenz-PCR-Methode anwenden.
04 quantitative Echtzeit-Fluoreszenz-PCR
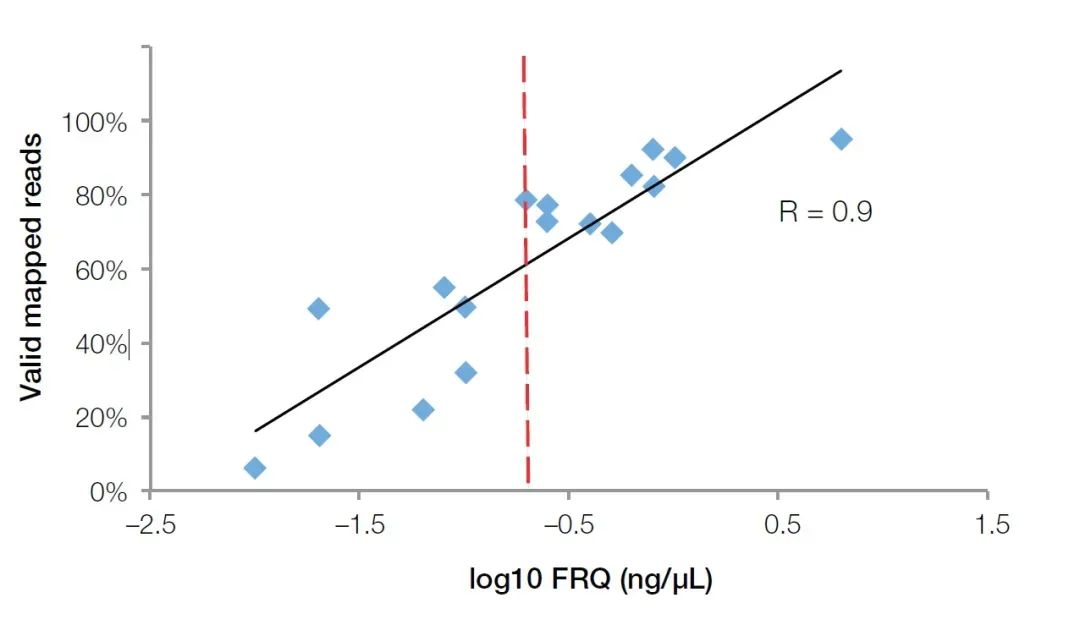
Die quantitative Echtzeit-Fluoreszenz-PCR-Methode kann nicht nur die Inhibitoren in der Probe nachweisen, sondern auch die Qualität der RNA in der FFPE-Probe genau wiedergeben.Im Vergleich zu biologischen Analysegeräten von Agilent erfreuen sich quantitative Echtzeit-Fluoreszenzinstrumente in großen biologischen Laboratorien aufgrund ihrer breiteren Anwendungsmöglichkeiten größerer Beliebtheit.Um die Qualität von RNA-Proben zu testen, müssen wir lediglich Primersonden für interne Referenzgene wie GUSB (Kat.-Nr. Hs00939627) kaufen oder vorbereiten.Durch die Verwendung dieses Satzes von Primern, Sonden und Standards (Gesamt-RNA bekannter Konzentration) zur Durchführung absolut quantitativer Experimente kann die effektive RNA-Fragmentkonzentration als Bewertungsstandard für die RNA-Qualität berechnet werden (kurz: Functional RNA Quantitation (FRQ)).In einem NGS-Test haben wir festgestellt, dass die FRQ von RNA-Proben eine sehr hohe Korrelation mit dem effektiven Datenvolumen aufweist.Bei allen Proben mit mehr als 0,2 ng/ul FRQ können mindestens 70 % der Messwerte die Referenzsequenz effektiv abdecken (Abbildung 4).
Abbildung 4: Der mit der quantitativen Fluoreszenzmethode ermittelte FRQ-Wert weist eine sehr hohe Korrelation (R2>0,9) mit den im NGS-Experiment erhaltenen effektiven Daten auf.Die rote Linie ist der FRQ-Wert von 0,2 ng/uL (log10 = -0,7).【4】
Die quantitative Echtzeit-PCR-Methode ist nicht nur auf FFPE-Proben anwendbar, sondern kann auch Inhibitoren in Proben effektiv überwachen.Wir können die nachzuweisende Probe mit der internen Positivkontrolle (IPC) und ihrem Assay in das Reaktionssystem geben und dann eine Fluoreszenzquantifizierung durchführen, um den Ct-Wert zu erhalten.Wenn der Ct-Wert hinter dem Ct-Wert in der Reaktion ohne Probe zurückbleibt, weist dies darauf hin, dass der Inhibitor in der Probe vorhanden ist und die Amplifikationseffizienz in der Reaktion hemmt.
05 Qubit-Fluoreszenzfarbstoffmethode
Das Qubit-Fluorometer ist das am häufigsten verwendete kleine Gerät zur Bestimmung der Nukleinsäurekonzentration und -reinheit, das einfach zu bedienen ist und in fast jedem molekularbiologischen Labor vorhanden ist.Es berechnet die Konzentration der Nukleinsäure genau, indem es einen Nukleinsäure-bindenden Fluoreszenzfarbstoff (Qubit-Nachweisreagenz) erkennt.Qubit verfügt über eine hohe Empfindlichkeit und Spezifität und kann RNA bis auf eine pg/µL-Konzentration genau quantifizieren.Zusätzlich zu der bekannten Fähigkeit, die Nukleinsäurekonzentration genau zu quantifizieren, kann das neueste Modell von Thermo Fisher, Qubit 4.0, auch die Integrität von RNA erkennen.Das RNA-Nachweissystem (RNA IQ Assay) von Qubit 4.0 erkennt die Integrität der RNA durch den gleichzeitigen Nachweis zweier spezifischer Fluoreszenzfarbstoffe.Diese beiden Fluoreszenzfarbstoffe können an große bzw. kleine RNA-Fragmente binden.Diese beiden Fluoreszenzfarbstoffe zeigen den Anteil großer RNA-Fragmente in der Probe an und daraus lässt sich der IQ-Wert (Integrität und Qualität) berechnen, der die RNA-Qualität repräsentiert.Der IQ-Wert gilt sowohl für FFPE- als auch für Nicht-FFPE-Proben und hat großen Einfluss auf die spätere Sequenzierungsqualität.Nehmen wir als Beispiel NGS-Experimente: In den RNA-Seq-Testexperimenten, die auf der Ion Torrent™-Plattform durchgeführt wurden, hatten die meisten Proben mit IQ-Werten über 4 mindestens 50 % effektive Messwerte (Abbildung 5).Im Vergleich zu den oben genannten Nachweismethoden ist der Qubit IQ Assay nicht nur bequemer zu bedienen und benötigt weniger Zeit (innerhalb von fünf Minuten), sondern weist auch eine große Korrelation zwischen dem gemessenen Parameter-IQ-Wert und der Datenqualität nachgeschalteter Experimente auf.
Abbildung 5 zeigt eine große Korrelation zwischen dem Qubit-RNA-IQ-Wert und den kartierten Reads von RNA-Seq.【5】
Durch die obige Einführung glaube ich, dass jeder über ein ausreichendes Verständnis der verschiedenen Methoden zur RNA-Qualitätskontrolle verfügt.In der Praxis können Sie wählen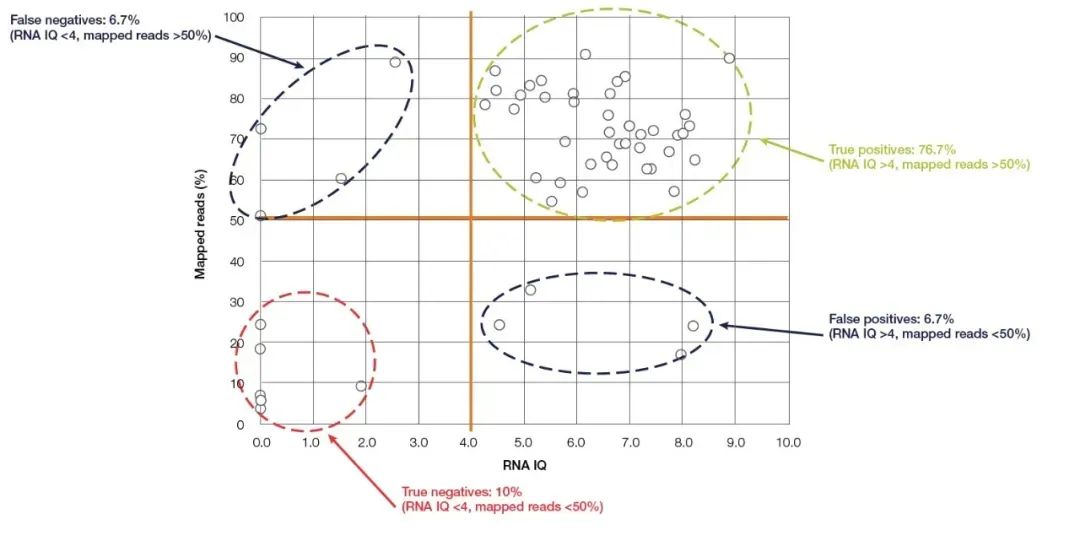 die entsprechende Methode je nach Probentyp und vorhandenen Instrumenten.Nur durch eine gute Kontrolle der RNA-Qualität können wir das Scheitern nachfolgender Experimente aufgrund schlechter Probenqualität vermeiden und so wertvolle Zeit, Energie und Kosten sparen.
die entsprechende Methode je nach Probentyp und vorhandenen Instrumenten.Nur durch eine gute Kontrolle der RNA-Qualität können wir das Scheitern nachfolgender Experimente aufgrund schlechter Probenqualität vermeiden und so wertvolle Zeit, Energie und Kosten sparen.
Referenzprodukte:
Tier-Gesamt-RNA-Isolierungskit
Zell-Gesamt-RNA-Isolierungskit
Verweise
【1】Schroeder, A., Mueller, O., Stocker, S. et al.Die RIN: eine RNA-Integritätsnummer zur Zuordnung von Integritätswerten zu RNA-Messungen.BMC Molecular Biol 7, 3 (2006).https:// doi.org/10.1186/1471-21 99-7-3
【2】Benutzerhandbuch zum Oncomine Human Immune Repertoire (Veröffentlichungsnr. MAN0017438 Rev. C.0).
[3] Leah C Wehmas, Charles E Wood, Brian N Chorley, Carole L Yauk, Gail M Nelson, Susan D Hester, Enhanced Quality Metrics for Assessing RNA Derived From Archival Formalin-Fixed Paraffin-Embedded Tissue Samples, Toxicological Sciences, Band 170, Ausgabe 2, August 2019, Seiten 357–373,https://doi.org/10.1093/toxsci/
Zeitpunkt der Veröffentlichung: 12. Juni 2023