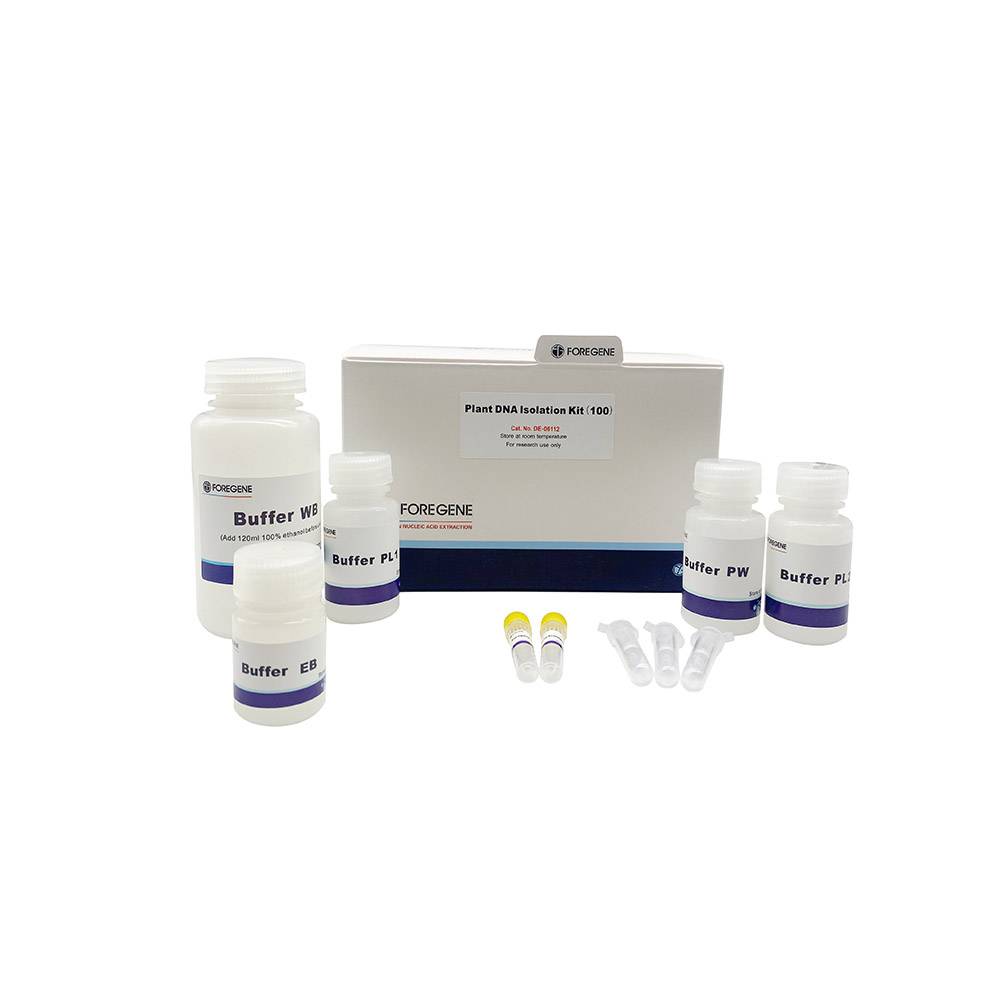
Fabrikquelle High Fidelity Hotstart Taq Mix PCR Kit PCR Master Mix 2 × A8 Fasthifi PCR Mastermix
Wir legen Wert auf die Weiterentwicklung und bringen fast jedes Jahr neue Lösungen auf den Markt für High Fidelity Hotstart Taq Mix PCR Kit PCR Master Mix 2 × A8 Fasthifi PCR Mastermix. Wir sind bestrebt, Ihnen kompetente Reinigungstechnologie und -optionen anzubieten!
Wir legen Wert auf Weiterentwicklung und bringen fast jedes Jahr neue Lösungen auf den MarktChina PCR Kit und PCRBisher wurde die Warenliste regelmäßig aktualisiert und zog Kunden aus der ganzen Welt an.Auf unserer Website finden Sie ausführliche Informationen und unser Kundendienstteam bietet Ihnen einen erstklassigen Beratungsservice.Sie werden Ihnen helfen, sich eingehend über unsere Produkte zu informieren und eine zufriedenstellende Verhandlung zu führen.Auch ein Firmenbesuch in unserem Werk in Brasilien ist jederzeit willkommen.Ich hoffe, Ihre Anfragen für eine angenehme Zusammenarbeit zu erhalten.
Bedienungsanleitung:
Wir legen Wert auf die Weiterentwicklung und bringen fast jedes Jahr neue Lösungen auf den Markt für High Fidelity Hotstart Taq Mix PCR Kit PCR Master Mix 2 × A8 Fasthifi PCR Mastermix. Wir sind bestrebt, Ihnen kompetente Reinigungstechnologie und -optionen anzubieten!
FabrikquelleChina PCR Kit und PCRBisher wurde die Warenliste regelmäßig aktualisiert und zog Kunden aus der ganzen Welt an.Auf unserer Website finden Sie ausführliche Informationen und unser Kundendienstteam bietet Ihnen einen erstklassigen Beratungsservice.Sie werden Ihnen helfen, sich eingehend über unsere Produkte zu informieren und eine zufriedenstellende Verhandlung zu führen.Auch ein Firmenbesuch in unserem Werk in Brasilien ist jederzeit willkommen.Ich hoffe, Ihre Anfragen für eine angenehme Zusammenarbeit zu erhalten.
Leitfaden zur Problemanalyse
The following is an analysis of the problems that may be encountered in the extraction of plant genomic DNA, hoping to be helpful to your experiments. In addition, for other experimental or technical problems other than operation instructions and problem analysis, we have dedicated technical support to help you. If you have any needs, please contact us: 028-83360257 or E-mali: Tech@foregene.com.
Geringe Ausbeute oder keine DNA
Normalerweise gibt es viele Faktoren, die die Ausbeute genomischer DNA beeinflussen, darunter die Quelle der Probe, das Alter der Probe, die Lagerbedingungen der Probe und der Vorgang.
Bei der Extraktion konnte keine genomische DNA gewonnen werden
1. Die Gewebeproben werden unsachgemäß oder zu lange gelagert, was zum Abbau der genomischen DNA führt.
Empfehlung: Gewebeproben in flüssigem Stickstoff oder -20 lagern°C;Versuchen Sie, neu gesammelte Proben für die Extraktion genomischer DNA zu verwenden.
2. Eine zu geringe Probenmenge kann dazu führen, dass die entsprechende genomische DNA nicht extrahiert wird.
Vorschlag: Bei Gewebeproben, die lange gelagert wurden oder einen starken Abbau der genomischen DNA aufweisen, kann die Menge der Gewebeproben entsprechend erhöht werden, um beträchtliche Mengen an genomischer DNA zu extrahieren.Die Menge der Probe kann je nach DNA-Bedarf bestimmt werden, die frische Probe sollte jedoch 100 mg und die trockene Probe 30 mg nicht überschreiten.
3. Die Probe wird nicht mit flüssigem Stickstoff gemahlen oder zu lange nach flüssigem Stickstoff platziert.
Vorschlag: Während der DNA-Extraktion muss die Probe vollständig mit flüssigem Stickstoff zerkleinert werden, um die Zellwand aufzubrechen;Nach dem Mahlen überführen Sie bitte das Probenpulver in den auf 65 °C vorgeheizten PL1°C so schnell wie möglich (sobald das gemahlene Pulver geschmolzen ist, beginnt die genomische DNA schnell abzubauen).
4. Eine unsachgemäße Lagerung der Foregene-Protease führt zu einer verringerten oder inaktivierten Aktivität.
Empfehlung: Bestätigen Sie die Lagerbedingungen der Foregene Protease oder ersetzen Sie sie durch eine neue Foregene Protease für die enzymatische Hydrolyse.
5. Das Kit wurde unsachgemäß oder zu lange gelagert, was dazu führte, dass einige Komponenten im Kit versagten.
Empfehlung: Erwerben Sie für entsprechende Vorgänge ein neues Extraktionskit für genomische Pflanzen-DNA.
6. Unsachgemäße Verwendung des Kits.
Vorschlag: Kaufen Sie ein Pflanzen-DNA-Isolierungskit speziell für Proben zur Extraktion und Reinigung pflanzlicher genomischer DNA.
7. Puffer WB ohne Zugabe von awasserhaltig Ethanol.
Empfehlung: Stellen Sie sicher, dass Sie dem Puffer WB das richtige Volumen an absolutem Ethanol hinzufügen.
8. Der Eluent wurde nicht korrekt auf die Silica-Membran getropft.
Vorschlag: Den vorgewärmten Eluenten auf 65°C zugeben℃Tropfen Sie es in die Mitte der Kieselgelmembran und lassen Sie es 5 Minuten lang bei Raumtemperatur stehen, um die Elutionseffizienz zu erhöhen.
Extraktion zur Gewinnung genomischer DNA mit geringer Ausbeute
1. Die Probe wird unsachgemäß oder zu lange gelagert, was zum Abbau der genomischen DNA führt.
Empfehlung: Gewebeproben bei -20 °C lagern℃;Versuchen Sie, neu gesammelte Gewebeproben für die Extraktion genomischer DNA zu verwenden.
2. Wenn die Menge an Gewebeproben zu gering ist, ist die extrahierte genomische DNA geringer.
Vorschlag: Einige Pflanzenproben sind wasserreich, etwa Wasserpflanzen wie Algen etc., die Dosierung kann entsprechend erhöht werden oder das Wasser vor der Operation etwas entwässert werden.
3. Die Proben wurden nicht gründlich mit flüssigem Stickstoff gemahlen oder wurden nach dem Mahlen zu lange bei Raumtemperatur belassen.
Vorschlag: Das Mahlen von flüssigem Stickstoff muss ausreichend sein und die Zellwand der Probe sollte so weit wie möglich durchbrochen werden.Unmittelbar nach dem Mahlen sollte das Probenpulver in 65 überführt werden℃Vorgewärmter Puffer PL1 für den nächsten Schritt.
4. Nicht das richtige Kit verwenden.
Empfehlung: Verwenden Sie ein spezielles Pflanzen-DNA-Isolierungskit, um genomische Pflanzen-DNA zu extrahieren und zu reinigen.
5. Eine unsachgemäße Lagerung der Foregene-Protease führt zu einer verringerten oder inaktivierten Aktivität.
Empfehlung: Bestätigen Sie die Lagerbedingungen der Foregene Protease oder ersetzen Sie sie durch eine neue Foregene Protease für die enzymatische Hydrolyse.
6. Eluentenproblem
Empfehlung: Zur Elution bitte Puffer EB verwenden;bei Verwendung von ddH2O oder andere Eluenten, stellen Sie sicher, dass der pH-Wert des Eluenten zwischen 7,0 und 8,5 liegt.
7. Der Eluent wird nicht richtig getropft
Vorschlag: Bitte geben Sie den Elutionstropfen in die Mitte der Silica-Membran und lassen Sie ihn 5 Minuten lang bei Raumtemperatur stehen, um die Elutionseffizienz zu erhöhen.
8. Das Eluentenvolumen ist zu klein
Vorschlag: Bitte verwenden Sie den Eluenten für die genomische DNA-Elution gemäß den Anweisungen, mindestens jedoch nicht weniger als 100μl.
Extrahierte genomische DNA mit geringer Reinheit
Die geringe Reinheit der genomischen DNA führt zum Scheitern oder zu einer schlechten Wirkung nachgelagerter Experimente, z. B.: Das Enzym kann nicht geschnitten werden und das Zielgenfragment kann nicht durch PCR erhalten werden.
1. Verschiedene Proteinkontaminationen, RNA-Kontaminationen.
Analyse: Puffer PW wurde nicht zum Waschen der Säule verwendet;Puffer PW wurde nicht zum Waschen der Säule mit der richtigen Zentrifugationsgeschwindigkeit verwendet.
Vorschlag: Stellen Sie sicher, dass sich im Überstand kein Niederschlag bildet, wenn der Überstand durch die Säule geleitet wird.Stellen Sie sicher, dass Sie die Reinigungssäule gemäß den Anweisungen mit Puffer PW waschen. Dieser Schritt kann nicht ausgelassen werden.
2. Verunreinigung durch Ionen.
Analyse: Die Puffer-WB-Waschsäule wurde weggelassen oder nur einmal gewaschen, was zu einer Restionenkontamination führte.
Empfehlung: Waschen Sie unbedingt zweimal mit Buffer WB gemäß den Anweisungen, um Restionen so weit wie möglich zu entfernen.
3. RNase-Kontamination.
Analyse: Dem Puffer wird exogene RNase hinzugefügt;Ein falscher Waschvorgang im Puffer PW führt zu Rest-RNase und beeinträchtigt nachgelagerte experimentelle RNA-Vorgänge, wie z. B. die In-vitro-Transkription.
Vorschlag: Nukleinsäure-Extraktionskits der Foregene-Serie können RNA ohne zusätzliche RNase entfernen, und alle Reagenzien im Plant DNA Isolation Kit benötigen keine RNase;Stellen Sie sicher, dass Sie die Reinigungssäule gemäß den Anweisungen mit Puffer PW waschen. Dieser Schritt kann nicht ausgelassen werden.
4. Ethanolrückstände.
Analyse: Nach dem Waschen der Reinigungssäule mit Puffer WB wurde keine Zentrifugation mit leeren Röhrchen durchgeführt.
Empfehlung: Befolgen Sie die Anweisungen für die ordnungsgemäße Zentrifugation leerer Röhrchen.
Bedienungsanleitung:
Bedienungsanleitung für das Pflanzen-DNA-Isolierungskit