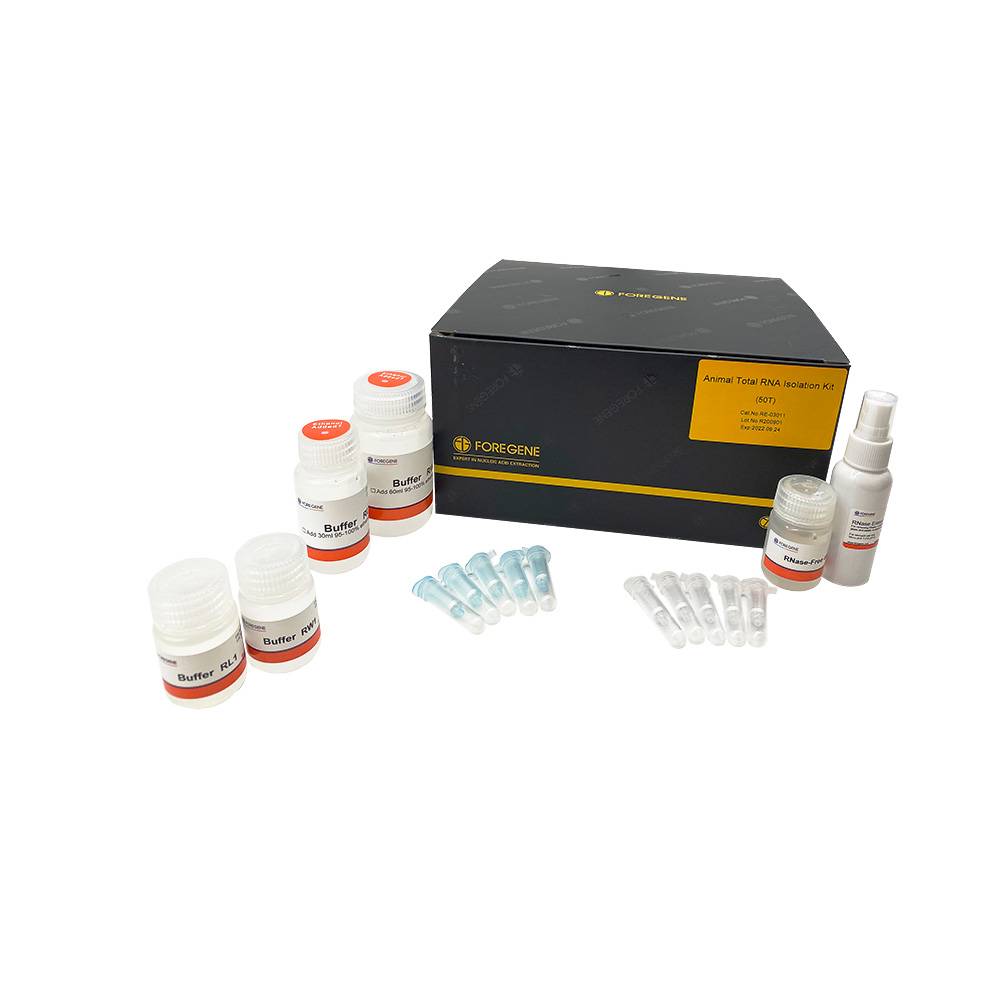
„Gesamt-RNA-Extraktion“
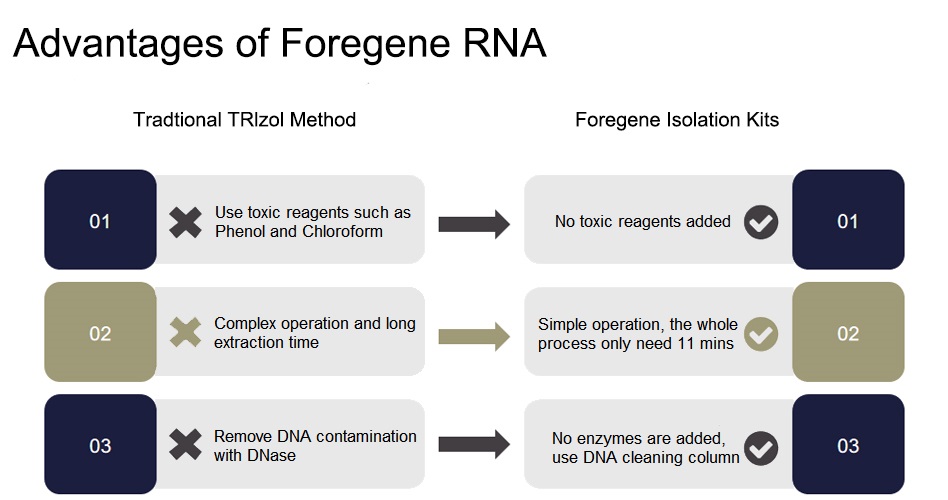
Wir halten an dem Grundsatz fest, „Produkte von höchster Qualität zu schaffen und Menschen aus der ganzen Welt anzufreunden“, und stellen stets den Wunsch der Käufer nach einer „vollständigen RNA-Extraktion“ in den Vordergrund. Wir sind bereit, mit Geschäftsfreunden aus dem In- und Ausland zusammenzuarbeiten und gemeinsam eine großartige Zukunft zu schaffen.
Getreu dem Motto „Produkte von höchster Qualität herstellen und Freundschaften mit Menschen aus der ganzen Welt schließen“ setzen wir stets den Wunsch der Verbraucher in den VordergrundGesamt-RNA-ExtraktionAufgrund der guten Qualität und der angemessenen Preise wurden unsere Produkte in mehr als 10 Länder und Regionen exportiert.Wir freuen uns auf die Zusammenarbeit mit allen Kunden aus dem In- und Ausland.Darüber hinaus ist die Zufriedenheit unserer Kunden unser ewiges Streben.
Beschreibungen der Kits
50 Vorbereitungen, 200 Vorbereitungen
Dieses Kit verwendet die von unserem Unternehmen entwickelte Spin-Säule und die Formel, die mit hoher Effizienz hochreine und hochwertige Gesamt-RNA aus verschiedenen tierischen Geweben extrahieren kann. Es bietet eine effiziente DNA-Reinigungssäule, die genomische DNA einfach und zeitsparend vom Überstand und Gewebelysat trennen und adsorbieren kann;Die reine RNA-Säule kann RNA effizient binden und kann mit einer einzigartigen Formel viele Proben gleichzeitig verarbeiten.
Das gesamte System ist RNase-frei, sodass die extrahierte RNA nicht abgebaut wird;Pufferwaschsystem für Puffer RW1 und Puffer RW2, sodass die erhaltene RNA frei von Protein-, DNA-, Ionen- und organischen Verbindungen ist.
Kit-Komponenten:
| Puffer RL1, Puffer RL2 |
| Puffer RW1, Puffer RW2 |
| RNase-freies ddH2O, DNA-Reinigungssäule |
| Nur-RNA-Säule |
Merkmale und Vorteile
■ Sie müssen sich keine Sorgen über den RNA-Abbau machen;das gesamte System ist RNase-frei
■ Entfernen Sie effektiv DNA mithilfe der DNA-Reinigungssäule
■ DNA entfernen, ohne DNase hinzuzufügen
■ Einfach – alle Vorgänge werden bei Raumtemperatur durchgeführt
■ Schnell – der Vorgang kann in 30 Minuten abgeschlossen werden
■ Sicher – kein organisches Reagenz erforderlich
■ Hohe Reinheit -OD260/280≈1,8-2,1
Kit-Anwendung
Es eignet sich für die Extraktion und Reinigung von Gesamt-RNA aus einer Vielzahl frischer oder gefrorener tierischer Gewebe oder kultivierter Zellen.
Produktparameter
■ Downstream-Anwendungen: Erststrang-cDNA-Synthese, RT-PCR, molekulares Klonen, Northern Blot usw.
■ Proben: tierische Gewebe, kultivierte Zellen
■ Dosierung: Gewebe 10–20 mg, Zellen (2–5)×106
■ Maximale DNA-Bindungskapazität der Reinigungssäule: 80 μg
■ Elutionsvolumen: 50–200 μl
Arbeitsablauf
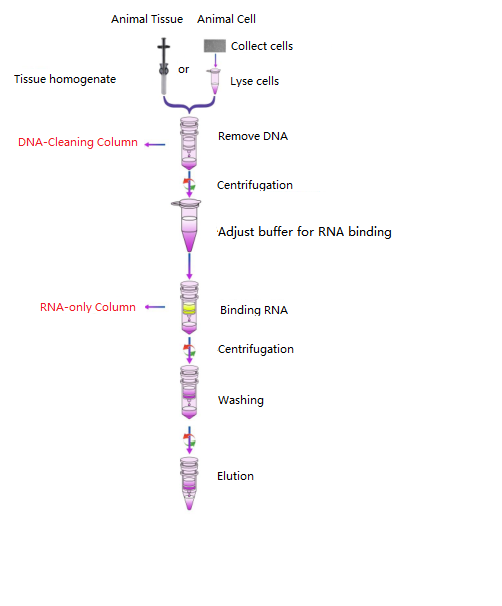
Diagramm
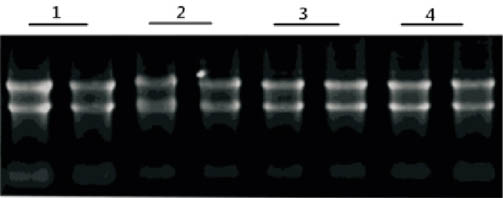
Animal Total RNA Isolation Kit behandelt 20 mg
Frische Mäuseproben, 5 % gereinigte Gesamt-RNA, 1 % Agar
Glykogel-Elektrophorese
1: Milz 2: Niere
3: Leber 4: Herz
Lagerung und Haltbarkeit
Das Kit kann 24 Monate lang bei Raumtemperatur (15–25 °C) oder länger bei 2–8 °C gelagert werden.Puffer RL1 kann nach Zugabe von β-Mercaptoethanol (optional) einen Monat lang bei 4 °C gelagert werden. Getreu dem Motto „Produkte von höchster Qualität herstellen und Menschen aus der ganzen Welt befreunden“ wecken wir stets den Wunsch von Käufern nach erneuerbarem Design für den chinesischen Lieferanten organischer Chemikalien Isopropylalkohol CAS 67-63-0 zu einem guten Preis. Wir sind bereit, mit Geschäftsfreunden aus dem In- und Ausland zusammenzuarbeiten und gemeinsam eine großartige Zukunft zu schaffen.
Erneuerbares Design für China Isopropylalkohol, Lieferant von Isopropylalkohol. Aufgrund der guten Qualität und angemessenen Preise wurden unsere Produkte in mehr als 10 Länder und Regionen exportiert.Wir freuen uns auf die Zusammenarbeit mit allen Kunden aus dem In- und Ausland.Darüber hinaus ist die Zufriedenheit unserer Kunden unser ewiges Streben.
RNA wird nicht extrahiert oder die RNA-Ausbeute ist gering
Es gibt oft eine Vielzahl von Faktoren, die die Rückgewinnungseffizienz beeinflussen, wie zum Beispiel: RNA-Gehalt der Gewebeprobe, Betriebsmethode, Elutionsvolumen usw.
1. Während des Betriebs wurde eine Eisbad- oder kryogene (4 °C) Zentrifugation durchgeführt.
Empfehlung: Während des gesamten Prozesses bei Raumtemperatur (15–25 °C) arbeiten, kein Eisbad durchführen und bei niedrigen Temperaturen zentrifugieren.
2. Unsachgemäße Probenkonservierung oder übermäßig lange Probenlagerung.
Empfehlung: Proben bei -80 °C lagern oder in flüssigem Stickstoff einfrieren und wiederholtes Einfrieren und Auftauen vermeiden;Versuchen Sie, frisches Gewebe oder kultivierte Zellen für die RNA-Extraktion zu verwenden.
3. Unzureichende Probenlyse.
Empfehlung: Achten Sie bei der Homogenisierung von Gewebe darauf, dass das Gewebe ausreichend homogenisiert ist und die Gewebezellen ausreichend gespalten sind, um die RNA-Freisetzung zu erklären.
4. Der Eluent wurde nicht korrekt hinzugefügt.
Empfehlung: Bestätigen Sie, dass RNase-Free ddH2O wird tropfenweise in die Mitte der Membran der Reinigungssäule gegeben.
5. Es wurde nicht das richtige Volumen an absolutem Ethanol zu Puffer RL2 oder Puffer RW2 hinzugefügt.
Empfehlung: Befolgen Sie die Anweisungen, geben Sie die richtige Menge absolutes Ethanol zu Puffer RL2 und Puffer RW2 hinzu und mischen Sie gut, bevor Sie das Kit verwenden.
6. Die Dosierung der Gewebeprobe ist nicht angemessen.
Empfehlung: Verwenden Sie 10–20 mg Gewebe oder (1–5) × 106Zellen pro 500 μl Puffer RL1, da eine übermäßige Gewebenutzung zu einer verringerten RNA-Extraktion führen kann.
7. Falsches Elutionsvolumen oder unvollständige Elution.
Empfehlung: Das Elutionsvolumen der Reinigungssäule beträgt 50-200 μl;Wenn der Elutionseffekt nicht zufriedenstellend ist, wird empfohlen, die Platzierungszeit bei Raumtemperatur nach Zugabe von vorgewärmtem RNase-Free ddH zu verlängern2O, zB für 5-10 Min.
8. Die Reinigungssäule weist nach dem Waschen mit Puffer RW2 Ethanolrückstände auf.
Empfehlung: Wenn nach dem Waschen des Puffers RW2 und der Zentrifugation des leeren Röhrchens für 1 Minute Ethanolrückstände vorhanden sind, kann die Zeit für die Zentrifugation des leeren Röhrchens auf 2 Minuten erhöht werden, oder die Reinigungssäule kann 5 Minuten lang auf Raumtemperatur gestellt werden, um das restliche Ethanol ausreichend zu entfernen.
Gereinigte RNA wird abgebaut
Die Qualität der gereinigten RNA hängt von Faktoren wie der Konservierung der Probe, RNase-Kontamination und Manipulation usw. ab.
1. Gewebeproben werden nicht rechtzeitig aufbewahrt.
Empfehlung: Wenn Gewebeproben oder Zellen nicht rechtzeitig nach der Entnahme verwendet werden, kryokonservieren Sie sie sofort bei -80 °C oder flüssigem Stickstoff.Verwenden Sie zur Extraktion von RNA nach Möglichkeit eine frisch entnommene Gewebe- oder Zellprobe.
2. Wiederholtes Einfrieren und Auftauen von Gewebeproben.
Empfehlung: Bei der Lagerung von Gewebeproben ist es am besten, diese zur Konservierung in kleine Stücke zu schneiden und bei der Verwendung eines der Stücke zu entfernen, um ein wiederholtes Einfrieren und Auftauen der Probe und den Abbau der RNA zu vermeiden.
3. RNase wird eingeführt oder es werden während der Operation keine Einweghandschuhe, Masken usw. getragen.
Empfehlung: RNA-Extraktionsexperimente werden am besten in separaten RNA-Manipulationsräumen durchgeführt und der Tisch wird vor dem Experiment abgeräumt.
Tragen Sie während des Experiments Einweghandschuhe und -masken, um den durch die Einführung von RNase verursachten RNA-Abbau zu minimieren.
4. Reagenzien werden während des Gebrauchs mit RNase kontaminiert.
Empfehlung: Für entsprechende Experimente durch ein neues Animal Total RNA Isolation Kit ersetzen.
5. Die bei der RNA-Manipulation verwendeten Zentrifugenröhrchen, Spitzen usw. sind mit RNase kontaminiert.
Empfehlung: Stellen Sie sicher, dass die bei der RNA-Extraktion verwendeten Zentrifugenröhrchen, Spitzen, Pipetten usw. alle RNase-frei sind.
Die gereinigte gewonnene RNA wirkt sich auf nachgelagerte Experimente aus
Durch die Reinigungssäule gereinigte RNA. Wenn die Salzionen und der Proteingehalt zu groß sind, wirkt sich dies auf nachgelagerte Experimente aus, wie zum Beispiel: Reverse Transkription,Northern Blot et al.
1. Die eluierte RNA weist Salzionenreste auf.
Empfehlung: Bestätigen Sie, dass dem Puffer RW2 das richtige Volumen Ethanol zugesetzt wurde, und führen Sie zwei Reinigungssäulenwaschvorgänge mit der für den Betrieb angegebenen Zentrifugalgeschwindigkeit durch.Wenn Salzionenrückstände vorhanden sind, lassen Sie die Reinigungssäule 5 Minuten lang bei Raumtemperatur auf Puffer RW2 stehen und führen Sie eine Zentrifugation durch, um die Entfernung von Salzverunreinigungen zu maximieren.
2. Ethanolrückstände in eluierter RNA.
Empfehlung: Bestätigen Sie, dass Sie nach dem Waschen mit Puffer RW2 den Zentrifugationsvorgang des leeren Röhrchens mit der für den Betrieb angegebenen Zentrifugationsgeschwindigkeit durchführen, die Zeit des Zentrifugationsvorgangs des leeren Röhrchens auf 2 Minuten erhöhen, wenn noch Ethanolrückstände vorhanden sind, oder das Gerät nach der Zentrifugation des leeren Röhrchens 5 Minuten lang bei Raumtemperatur belassen, um die Entfernung von Ethanolrückständen zu maximieren.